Saving Seurat objects
2025-04-16
Last updated: 2025-04-16
Checks: 7 0
Knit directory: muse/
This reproducible R Markdown analysis was created with workflowr (version 1.7.1). The Checks tab describes the reproducibility checks that were applied when the results were created. The Past versions tab lists the development history.
Great! Since the R Markdown file has been committed to the Git repository, you know the exact version of the code that produced these results.
Great job! The global environment was empty. Objects defined in the global environment can affect the analysis in your R Markdown file in unknown ways. For reproduciblity it’s best to always run the code in an empty environment.
The command set.seed(20200712) was run prior to running
the code in the R Markdown file. Setting a seed ensures that any results
that rely on randomness, e.g. subsampling or permutations, are
reproducible.
Great job! Recording the operating system, R version, and package versions is critical for reproducibility.
Nice! There were no cached chunks for this analysis, so you can be confident that you successfully produced the results during this run.
Great job! Using relative paths to the files within your workflowr project makes it easier to run your code on other machines.
Great! You are using Git for version control. Tracking code development and connecting the code version to the results is critical for reproducibility.
The results in this page were generated with repository version 5dd72fd. See the Past versions tab to see a history of the changes made to the R Markdown and HTML files.
Note that you need to be careful to ensure that all relevant files for
the analysis have been committed to Git prior to generating the results
(you can use wflow_publish or
wflow_git_commit). workflowr only checks the R Markdown
file, but you know if there are other scripts or data files that it
depends on. Below is the status of the Git repository when the results
were generated:
Ignored files:
Ignored: .Rproj.user/
Ignored: data/1M_neurons_filtered_gene_bc_matrices_h5.h5
Ignored: data/293t/
Ignored: data/293t_3t3_filtered_gene_bc_matrices.tar.gz
Ignored: data/293t_filtered_gene_bc_matrices.tar.gz
Ignored: data/5k_Human_Donor1_PBMC_3p_gem-x_5k_Human_Donor1_PBMC_3p_gem-x_count_sample_filtered_feature_bc_matrix.h5
Ignored: data/5k_Human_Donor2_PBMC_3p_gem-x_5k_Human_Donor2_PBMC_3p_gem-x_count_sample_filtered_feature_bc_matrix.h5
Ignored: data/5k_Human_Donor3_PBMC_3p_gem-x_5k_Human_Donor3_PBMC_3p_gem-x_count_sample_filtered_feature_bc_matrix.h5
Ignored: data/5k_Human_Donor4_PBMC_3p_gem-x_5k_Human_Donor4_PBMC_3p_gem-x_count_sample_filtered_feature_bc_matrix.h5
Ignored: data/97516b79-8d08-46a6-b329-5d0a25b0be98.h5ad
Ignored: data/Parent_SC3v3_Human_Glioblastoma_filtered_feature_bc_matrix.tar.gz
Ignored: data/brain_counts/
Ignored: data/cl.obo
Ignored: data/cl.owl
Ignored: data/jurkat/
Ignored: data/jurkat:293t_50:50_filtered_gene_bc_matrices.tar.gz
Ignored: data/jurkat_293t/
Ignored: data/jurkat_filtered_gene_bc_matrices.tar.gz
Ignored: data/pbmc20k/
Ignored: data/pbmc20k_seurat/
Ignored: data/pbmc3k/
Ignored: data/pbmc3k_bpcells_mat/
Ignored: data/pbmc3k_seurat.rds
Ignored: data/pbmc4k_filtered_gene_bc_matrices.tar.gz
Ignored: data/pbmc_1k_v3_filtered_feature_bc_matrix.h5
Ignored: data/pbmc_1k_v3_raw_feature_bc_matrix.h5
Ignored: data/refdata-gex-GRCh38-2020-A.tar.gz
Ignored: data/seurat_1m_neuron.rds
Ignored: data/t_3k_filtered_gene_bc_matrices.tar.gz
Ignored: r_packages_4.4.1/
Untracked files:
Untracked: analysis/bioc_scrnaseq.Rmd
Untracked: pbmc3k_save_rds.rds
Untracked: rsem.merged.gene_counts.tsv
Note that any generated files, e.g. HTML, png, CSS, etc., are not included in this status report because it is ok for generated content to have uncommitted changes.
These are the previous versions of the repository in which changes were
made to the R Markdown (analysis/seurat_save.Rmd) and HTML
(docs/seurat_save.html) files. If you’ve configured a
remote Git repository (see ?wflow_git_remote), click on the
hyperlinks in the table below to view the files as they were in that
past version.
| File | Version | Author | Date | Message |
|---|---|---|---|---|
| Rmd | 5dd72fd | Dave Tang | 2025-04-16 | Save additional assay |
| html | a22d5e1 | Dave Tang | 2025-04-15 | Build site. |
| Rmd | 77f2810 | Dave Tang | 2025-04-15 | Add miscellaneous data |
| html | 478564c | Dave Tang | 2025-04-15 | Build site. |
| Rmd | deec653 | Dave Tang | 2025-04-15 | Saving Seurat objects |
if ("BPCells" %in% row.names(installed.packages()) == FALSE){
remotes::install_github("bnprks/BPCells/r")
}suppressPackageStartupMessages(library(BPCells))
suppressPackageStartupMessages(library(Seurat))Load Data
Load from my server.
pbmc3k <- readRDS(url("https://davetang.org/file/pbmc3k_seurat.rds", "rb"))
pbmc3kAn object of class Seurat
32738 features across 2700 samples within 1 assay
Active assay: RNA (32738 features, 0 variable features)
1 layer present: countsUse BPCells
Sparse matrix.
class(pbmc3k@assays$RNA$counts)[1] "dgCMatrix"
attr(,"package")
[1] "Matrix"Write a matrix directory and load the matrix using {BPCells}.
my_outdir <- "data/pbmc3k_bpcells_mat"
if(!dir.exists(my_outdir)){
BPCells::write_matrix_dir(
mat = pbmc3k@assays$RNA$counts,
dir = my_outdir
)
}
# Now that we have the matrix on disk, we can load it
pbmc3k.mat <- open_matrix_dir(dir = my_outdir)
pbmc3k.mat32738 x 2700 IterableMatrix object with class MatrixDir
Row names: MIR1302-10, FAM138A ... AC002321.1
Col names: AAACATACAACCAC-1, AAACATTGAGCTAC-1 ... TTTGCATGCCTCAC-1
Data type: double
Storage order: column major
Queued Operations:
1. Load compressed matrix from directory /home/rstudio/muse/data/pbmc3k_bpcells_matCreate a Seurat object.
pbmc3k_bpcells <- CreateSeuratObject(
counts = pbmc3k.mat,
project = 'pbmc3k',
min.cells = 3,
min.features = 200
)
pbmc3k_bpcells@assays$RNA$counts13714 x 2700 IterableMatrix object with class RenameDims
Row names: AL627309.1, AP006222.2 ... SRSF10.1
Col names: AAACATACAACCAC-1, AAACATTGAGCTAC-1 ... TTTGCATGCCTCAC-1
Data type: double
Storage order: column major
Queued Operations:
1. Load compressed matrix from directory /home/rstudio/muse/data/pbmc3k_bpcells_mat
2. Select rows: 6, 9 ... 32733 and cols: 1, 2 ... 2700
3. Reset dimnamesSeurat version 4
Mitochondrial percent.
mito.genes <- grep(pattern = "^MT-", x = rownames(x = pbmc3k_bpcells@assays$RNA), ignore.case = TRUE, value = TRUE)
pbmc3k_bpcells[["percent.mt"]] <- PercentageFeatureSet(pbmc3k_bpcells, features = mito.genes)
VlnPlot(pbmc3k_bpcells, features = c("nFeature_RNA", "nCount_RNA", "percent.mt"), ncol = 3, layer = "counts")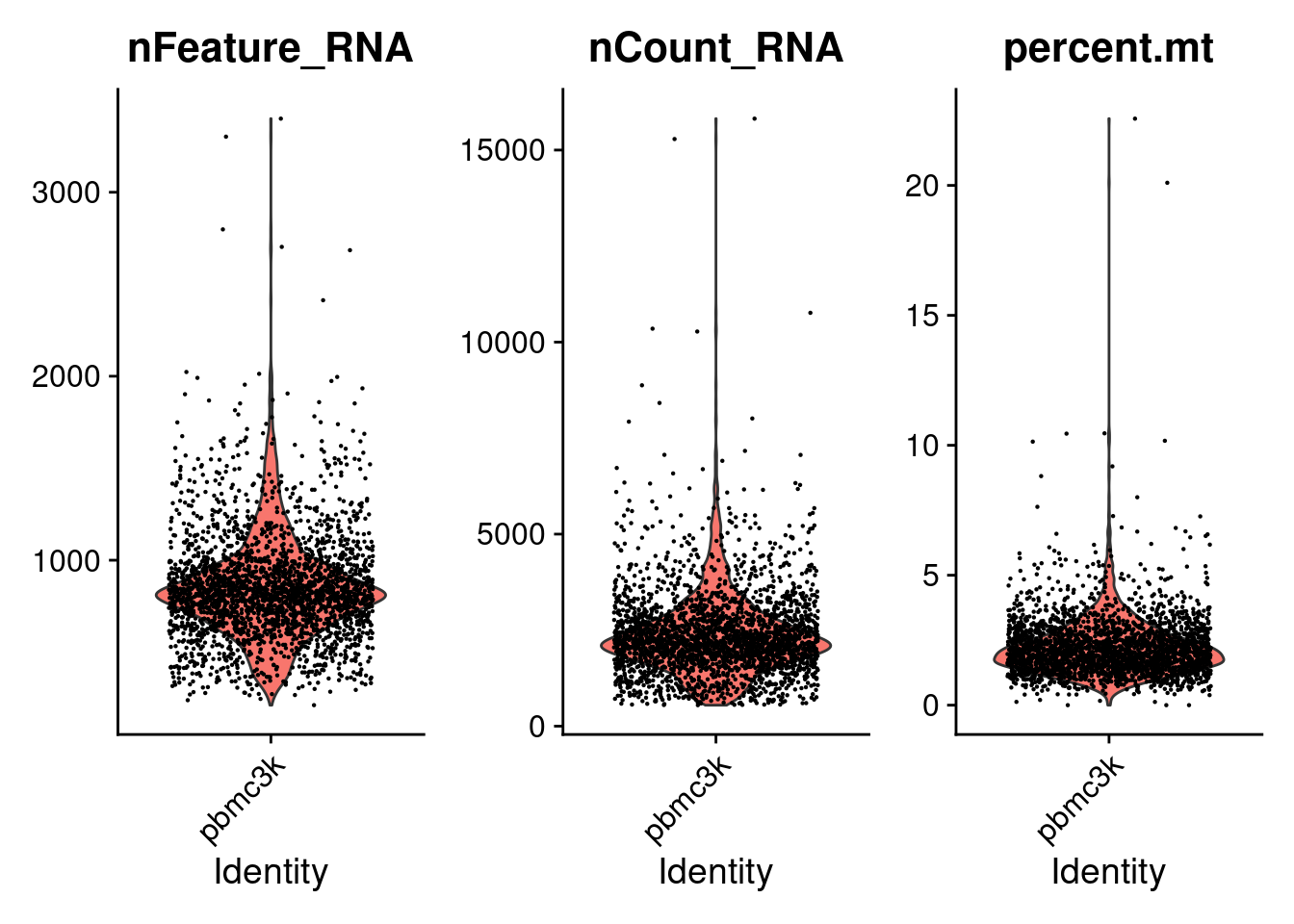
Save original data into RAW assay before filtering.
# build a new independent assay
raw_counts <- GetAssayData(pbmc3k_bpcells, layer = "counts", assay = "RNA")
raw_assay <- CreateAssayObject(counts = raw_counts)
pbmc3k_bpcells[["RAW"]] <- raw_assay
Seurat::Assays(pbmc3k_bpcells)[1] "RNA" "RAW"Seurat::DefaultAssay(pbmc3k_bpcells)[1] "RNA"Filter.
pbmc3k_bpcells <- subset(pbmc3k_bpcells, subset = percent.mt < 15)
pbmc3k_bpcellsAn object of class Seurat
27428 features across 2698 samples within 2 assays
Active assay: RNA (13714 features, 0 variable features)
1 layer present: counts
1 other assay present: RAWUnfortunately, the RAW assay becomes filtered as
well.
dim(pbmc3k_bpcells@assays$RNA$counts)[1] 13714 2698dim(pbmc3k_bpcells@assays$RAW$counts)[1] 13714 2698Seurat workflow.
debug_flag <- FALSE
start_time <- Sys.time()
pbmc3k_bpcells <- NormalizeData(pbmc3k_bpcells, normalization.method = "LogNormalize")Normalizing layer: countspbmc3k_bpcells <- FindVariableFeatures(pbmc3k_bpcells, selection.method = 'vst', nfeatures = 2000, verbose = debug_flag)
pbmc3k_bpcells <- ScaleData(pbmc3k_bpcells, verbose = debug_flag)
pbmc3k_bpcells <- RunPCA(pbmc3k_bpcells, verbose = debug_flag)
pbmc3k_bpcells <- RunUMAP(pbmc3k_bpcells, dims = 1:30, verbose = debug_flag)Warning: The default method for RunUMAP has changed from calling Python UMAP via reticulate to the R-native UWOT using the cosine metric
To use Python UMAP via reticulate, set umap.method to 'umap-learn' and metric to 'correlation'
This message will be shown once per sessionpbmc3k_bpcells <- FindNeighbors(pbmc3k_bpcells, dims = 1:30, verbose = debug_flag)
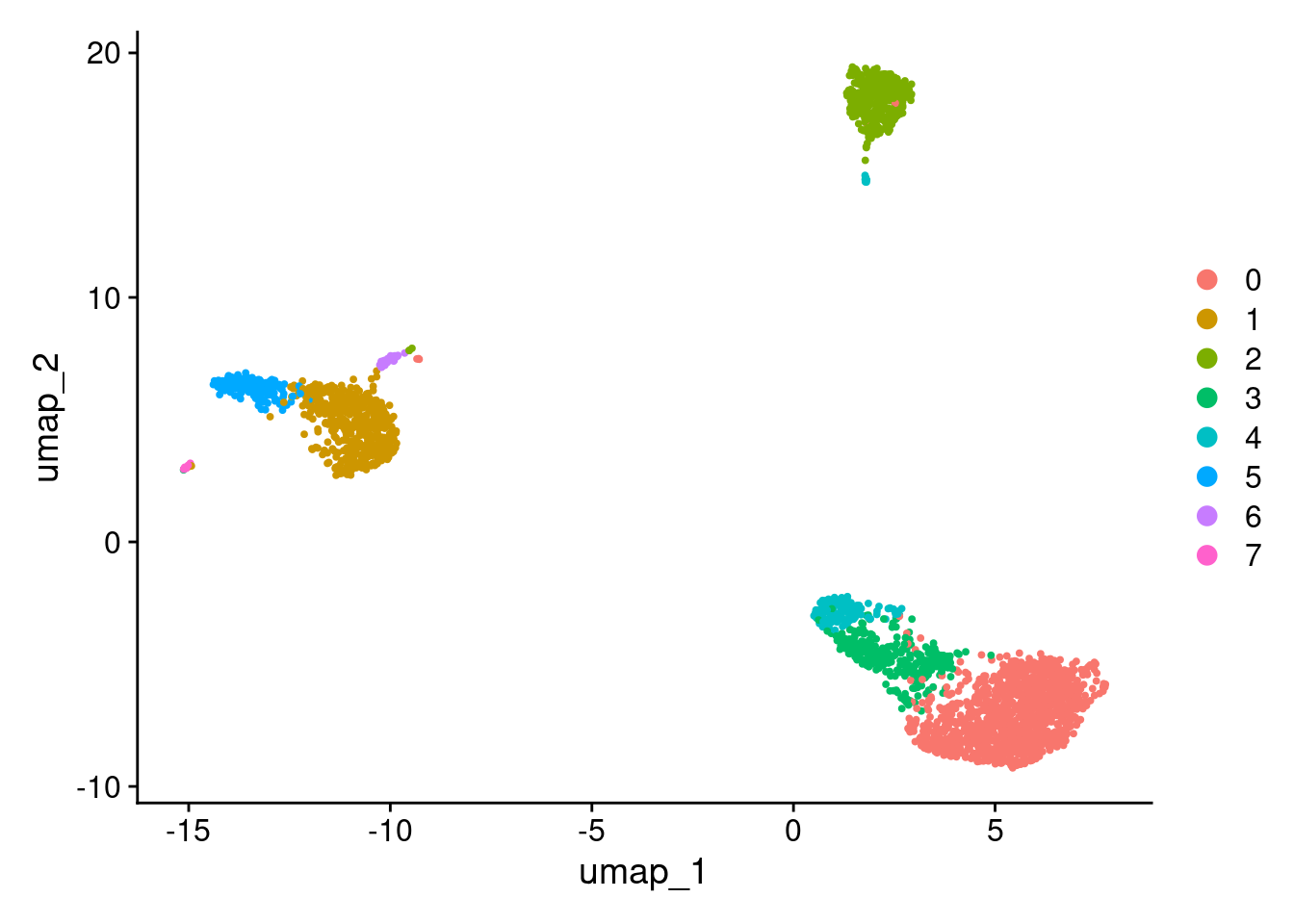
pbmc3k_bpcells <- FindClusters(pbmc3k_bpcells, resolution = 0.5, verbose = debug_flag)
pbmc3k_bpcellsAn object of class Seurat
27428 features across 2698 samples within 2 assays
Active assay: RNA (13714 features, 2000 variable features)
3 layers present: counts, data, scale.data
1 other assay present: RAW
2 dimensional reductions calculated: pca, umapend_time <- Sys.time()
end_time - start_timeTime difference of 11.83351 secsCounts.
pbmc3k_bpcells@assays$RNA$counts13714 x 2698 IterableMatrix object with class RenameDims
Row names: AL627309.1, AP006222.2 ... SRSF10.1
Col names: AAACATACAACCAC-1, AAACATTGAGCTAC-1 ... TTTGCATGCCTCAC-1
Data type: double
Storage order: column major
Queued Operations:
1. Load compressed matrix from directory /home/rstudio/muse/data/pbmc3k_bpcells_mat
2. Select rows: 6, 9 ... 32733 and cols: 1, 2 ... 2700
3. Reset dimnamesData.
pbmc3k_bpcells@assays$RNA$data13714 x 2698 IterableMatrix object with class RenameDims
Row names: AL627309.1, AP006222.2 ... SRSF10.1
Col names: AAACATACAACCAC-1, AAACATTGAGCTAC-1 ... TTTGCATGCCTCAC-1
Data type: double
Storage order: column major
Queued Operations:
1. Load compressed matrix from directory /home/rstudio/muse/data/pbmc3k_bpcells_mat
2. Select rows: 6, 9 ... 32733 and cols: 1, 2 ... 2700
3. Reset dimnames
4. Scale by 1e+04
5. Scale columns by 0.000413, 0.000204 ... 0.000504
6. Transform log1p
7. Reset dimnamesScale data.
pbmc3k_bpcells@assays$RNA$scale.data2000 x 2698 IterableMatrix object with class RenameDims
Row names: ISG15, CPSF3L ... MT-ND6
Col names: AAACATACAACCAC-1, AAACATTGAGCTAC-1 ... TTTGCATGCCTCAC-1
Data type: double
Storage order: column major
Queued Operations:
1. Load compressed matrix from directory /home/rstudio/muse/data/pbmc3k_bpcells_mat
2. Select rows: 43, 63 ... 32708 and cols: 1, 2 ... 2700
3. Reset dimnames
4. Scale by 1e+04
5. Scale columns by 0.000413, 0.000204 ... 0.000504
6. Transform log1p
7. Select rows: 514, 139 ... 580 and cols: all
8. Reset dimnames
9. Transform min by row: 5.2, 19.4 ... 2.3
10. Scale rows by 1.95, 0.55 ... 4.4
11. Shift rows by -0.143, -0.645 ... -0.132
12. Select rows: 636, 1273 ... 455 and cols: all
13. Reset dimnamesAdd miscellaneous data
Get and set miscellaneous data.
Misc(pbmc3k_bpcells)list()Set and output.
Misc(pbmc3k_bpcells, slot = "seed") <- 1984
Misc(pbmc3k_bpcells, slot = "author") <- "Davo"
Misc(pbmc3k_bpcells)$seed
[1] 1984
$author
[1] "Davo"Get specific slot.
Misc(pbmc3k_bpcells, slot = "author")[1] "Davo"Exporting
Save.
saveRDS(object = pbmc3k_bpcells, file = "pbmc3k_save_rds.rds")Load.
pbmc3k_read_rds <- readRDS("pbmc3k_save_rds.rds")
pbmc3k_read_rds@assays$RNA$counts13714 x 2698 IterableMatrix object with class RenameDims
Row names: AL627309.1, AP006222.2 ... SRSF10.1
Col names: AAACATACAACCAC-1, AAACATTGAGCTAC-1 ... TTTGCATGCCTCAC-1
Data type: double
Storage order: column major
Queued Operations:
1. Load compressed matrix from directory /home/rstudio/muse/data/pbmc3k_bpcells_mat
2. Select rows: 6, 9 ... 32733 and cols: 1, 2 ... 2700
3. Reset dimnamespbmc3k_read_rds@assays$RNA$data13714 x 2698 IterableMatrix object with class RenameDims
Row names: AL627309.1, AP006222.2 ... SRSF10.1
Col names: AAACATACAACCAC-1, AAACATTGAGCTAC-1 ... TTTGCATGCCTCAC-1
Data type: double
Storage order: column major
Queued Operations:
1. Load compressed matrix from directory /home/rstudio/muse/data/pbmc3k_bpcells_mat
2. Select rows: 6, 9 ... 32733 and cols: 1, 2 ... 2700
3. Reset dimnames
4. Scale by 1e+04
5. Scale columns by 0.000413, 0.000204 ... 0.000504
6. Transform log1p
7. Reset dimnamespbmc3k_read_rds@assays$RNA$scale.data2000 x 2698 IterableMatrix object with class RenameDims
Row names: ISG15, CPSF3L ... MT-ND6
Col names: AAACATACAACCAC-1, AAACATTGAGCTAC-1 ... TTTGCATGCCTCAC-1
Data type: double
Storage order: column major
Queued Operations:
1. Load compressed matrix from directory /home/rstudio/muse/data/pbmc3k_bpcells_mat
2. Select rows: 43, 63 ... 32708 and cols: 1, 2 ... 2700
3. Reset dimnames
4. Scale by 1e+04
5. Scale columns by 0.000413, 0.000204 ... 0.000504
6. Transform log1p
7. Select rows: 514, 139 ... 580 and cols: all
8. Reset dimnames
9. Transform min by row: 5.2, 19.4 ... 2.3
10. Scale rows by 1.95, 0.55 ... 4.4
11. Shift rows by -0.143, -0.645 ... -0.132
12. Select rows: 636, 1273 ... 455 and cols: all
13. Reset dimnamespbmc3k_read_rdsAn object of class Seurat
27428 features across 2698 samples within 2 assays
Active assay: RNA (13714 features, 2000 variable features)
3 layers present: counts, data, scale.data
1 other assay present: RAW
2 dimensional reductions calculated: pca, umapGet miscellaneous data.
Misc(pbmc3k_read_rds)$seed
[1] 1984
$author
[1] "Davo"Check RAW assay.
pbmc3k_read_rds@assays$RAW$counts13714 x 2698 sparse Matrix of class "dgCMatrix" [[ suppressing 32 column names 'AAACATACAACCAC-1', 'AAACATTGAGCTAC-1', 'AAACATTGATCAGC-1' ... ]]
[[ suppressing 32 column names 'AAACATACAACCAC-1', 'AAACATTGAGCTAC-1', 'AAACATTGATCAGC-1' ... ]]
AL627309.1 . . . . . . . . . . . . . . . . . . . . . . . . .
AP006222.2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-206L10.2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-206L10.9 . . . . . . . . . . . . . . . . . . . . . . . . .
LINC00115 . . . . . . . . . . . . . . . . . . . . . . . . .
NOC2L . . . . . . . . . . . 1 . . . . . . . . . . 1 . .
KLHL17 . . . . . . . . . . . . . . . . . . . . . . . . .
PLEKHN1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-54O7.17 . . . . . . . . . . . . . . . . . . . . . . . . .
HES4 . . . . . . . . . . . . . . . . . . 1 . . . . . .
RP11-54O7.11 . . . . . . . . . . . . . . . . . . . . . . . . .
ISG15 . . 1 9 . 1 . . . 3 . . 1 5 . 2 1 1 1 . . . 1 . 1
AGRN . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf159 . . . . . . . . . . . . . . . . . . . . . . . . .
TNFRSF18 . 2 . . . . . . . . . . . . . . . . . . . . . . .
TNFRSF4 . . . . . . . . 1 . . . . . . . . . . 12 . . . . .
SDF4 . . 1 . . . . . . . . . . . . . . . . 1 1 . . . .
B3GALT6 . . . . . . 1 . . . . . . . . . . . . . . . . . .
FAM132A . . . . . . . . . . . . . . . . . . . . . . . . .
UBE2J2 . . . . . . . . 1 . 1 . . 1 . . . . . 1 . . . . .
ACAP3 . . . . . . . . . . . . . . 1 . . . . . . . . . .
PUSL1 . . . . . . 1 . . . . . . . . . . . . . . . . . .
CPSF3L . . . . . . . . . . 1 . . . . . . . . . . . . . .
GLTPD1 . . . . . . . . . . . . . . . . . . . . . . . . .
DVL1 . . . . . . . . . . . . . . . . . . . . . . . . .
MXRA8 . . . . . . . . . . . . . . . . . . . . . . . . .
AURKAIP1 . 1 . 1 . . . 1 . . 2 . 2 1 . . . . . . . . 1 . 1
CCNL2 . . . . . . . . . . . . . 1 . . . . 2 1 . . . . .
RP4-758J18.2 . . . . . . . . . . 1 . . . . . . . . . . . . . .
MRPL20 1 . 1 . . . . 1 . . 2 . . 1 . . . . . 1 . . . . 1
ATAD3C . . . . . . . . . . . . . . . . . . . . . . . . .
ATAD3B . . . . . . . . . . . 1 . . . . . . . . 1 . . . .
ATAD3A . . . . . . . . . . . . . . . . . . . . . . . . .
SSU72 . 1 . 3 . . . . . . . . . 1 . . . 1 3 . 2 . . 1 .
AL645728.1 . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf233 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-345P4.9 . . . . . . . . . . . . . . . . . . . . . . . . .
MIB2 . 2 . . . . 1 . . . . . . 1 . . . . . . . . . . .
MMP23B . . . . . . . . . . . . . . . . . . . . . . . . .
CDK11B . . . . . . . . . . . . . . . . . . . . . . . . 1
SLC35E2B . . . . . . . . . . . . . . . . . . . . . . . . .
CDK11A . . . . . . . . . . . . . 1 . . . . . . . . . . 1
SLC35E2 . . . . . . . . . . . . . . . . . . . . . . . . .
NADK . . . 1 . . . . . . . . . 2 . . . . . . . . . . .
GNB1 . . . . . . . . 1 . . . . . . . . . 1 . 1 . . . .
RP1-140A9.1 . . . . . . . . . . . . . . . . . . . . . . . . .
TMEM52 . . . . . . . . . . . . . . . . . . . . . . . . .
PRKCZ . . . . . . . . . . . . . . . . . . . . . . . . .
RP5-892K4.1 . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf86 . . . . . . . . 1 . . . . . . . . . . . . . . . .
AL590822.2 . . . . . . . . . . . . . . . . . . . . . . . . .
SKI . . . . . . . . . . . . . . . . . . . . . . . . .
RER1 . 1 1 1 . . . . . . . 1 . 1 . . . . 2 . . . . . .
PEX10 . . . . . . . . . . . . . . . . . . . . . . . . .
PLCH2 . . . . . . . . . . . . . . . . . . . . . . . . .
PANK4 . . 1 . . 1 . . . . . . . . . . . . . . . . . . .
RP3-395M20.12 . 1 . . . . . . . . . . . . . . . . . . . . . . .
TNFRSF14 . . . 1 . 1 . . . . 1 . . . . . . . 1 . . 1 . . .
RP3-395M20.9 . . . . . . . . . . . . . . . . . . . . . . . . .
FAM213B . . . . . . . . . . . . . . . . . . . . . . . . .
MMEL1 . . . . . . . . . . . . . . . . . . . . . . . . .
TTC34 . . . . . . . . . . . . . . . . . . . . . . . . .
MEGF6 . . . . . . . . . . . . . . . . . . . . . . . . .
TPRG1L . . . . . . . . . . . . . . . . . . . . . . . . .
WRAP73 . . . . . . . . . . . . . . . . . . . . . . . . .
TP73-AS1 . . . . . . . . . . . 1 . . . . . . . . . . . . .
SMIM1 . . . . . . . . . . . . . . . . . . . . . . . . .
LRRC47 . . 1 . . . . . . . . . . . . . . . . . . . . . .
CEP104 . . . . . . . . . . . . . . . . . . . . . . . . .
DFFB . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf174 . . . . . . . . 1 . . . . . . . . . . . 1 . . . .
NPHP4 . . . . . . . . . . . . . . . . . . . . . . . . .
KCNAB2 . . . . . . . . . . . . . . . . . . 1 . 1 . . . .
RPL22 1 3 1 2 . . . 1 . . 3 2 2 1 1 2 . 2 4 1 6 4 4 . 1
RNF207 . . . . . . . . . . . . . . . . . . . . . . . . .
ICMT . . . . . . . . . . . . . . . . . . . . . . . . .
GPR153 . . . . . . . . . . . . . . . . . . . . . . . . .
ACOT7 . . . . . . 1 . . . . . . . . . . . . . . . . . .
RP1-202O8.3 . . . . . . . . . . . . . . . . . . . . . . . . .
ESPN . . . . . . . . . . . . . . . . . . . . . . . . .
TNFRSF25 2 . . . . . . 1 . . . . . . . . . . . . . . . . .
PLEKHG5 . . . . . . . . . . . . . . . . . . . . . . . . .
NOL9 . . . . . . . . . . . . . . . . . . . . . . . . .
ZBTB48 . . . . . . . . . . . . . . . . . . . . . . . . .
KLHL21 . . . . . . . . . . . . . . . . . . . . . . . . .
PHF13 . . . . . . . . . . . . . . . . . . . . . . . . .
THAP3 . . . . . . . 1 . . . . . 1 . . . . . . . . . . .
DNAJC11 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-312B8.1 . . . . . . . . . . . . . . . . . . . . . . . . .
CAMTA1 . 1 . . . . . . . . . . . . . . . . . . . . . . .
VAMP3 . . . . . . . . . . . . . 1 . . . 1 . . . . . . .
PER3 . . . . . . . . . . . . . . . . . . . . . . . . .
UTS2 . . . . . . . . . . . . . . . . . . . . . . . . .
TNFRSF9 . . . . . . . . . . . . . . . . . . . . . . . . .
PARK7 . . 2 . . . . . . 1 1 . 2 . . . 2 1 3 1 3 . 1 . .
SLC45A1 . . . . . . . . . . . . . . . . . . . . . . . . .
RERE . . . . . . 1 . . . . . . . . . . . . . . . . . .
RP5-1115A15.1 . . . . . . . . . . . . . . . . . . . . . . . . .
ENO1 . 2 2 2 . . 1 . . . 1 . . 1 . . . . 6 1 4 . 2 . .
ENO1-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
CA6 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC2A5 . . . . . . . . . . . . . . . . . . . . . . . . .
GPR157 . . . . . . . . . . . . . . . . . . . . . . . . .
H6PD . . . . . . . . . . . . . . . . . . . . . . . . .
SPSB1 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC25A33 . . . . . . . . . . . . . . . . . . . . . . . . .
TMEM201 . . . . . . . . . . . . . . . . . . . . . . . . .
PIK3CD . . . . . . . . . . . . . 1 . . . . . . . . . . .
C1orf200 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-558F24.4 . . . . . . . . . . . . . . . . . . . . . . . . .
CLSTN1 . . . . . . . . . . . . . . . . . . . . . . . . .
CTNNBIP1 . . . . 1 . . . . . . . 1 . . . . . 1 . . . . . 1
LZIC . . . . . . . . . 1 . . . 1 . . . . . . . . . . .
NMNAT1 . . . . . . . . . . . . . . . . . . . . . . . . .
RBP7 . . . . . . . . . . . . . . . . . . . . . . . 1 5
UBE4B . . . . . . . . . . . . . . . . . . . . . . . . .
KIF1B . . . . . . . . . . . . . . . . . . . . . . . . .
PGD . . . . . . . . . . . 1 . . . . 1 . . . . . . . .
APITD1 . . . . . . . . . . . . . . . . . . . . 1 . . . .
DFFA . . . . . . . . . . . . . 1 . . 1 . . . . . . . .
PEX14 . . 1 . . . . . . . . . . . . . . . . . . . . . .
CASZ1 . . . . . . . . . . . . . . . . . . . . . . . . .
TARDBP . . . . . . . . . . . . . . . 1 . . . . . . . . .
RP4-635E18.8 . . . . . . . . . . . . . . . . . . . . . . . . .
SRM . . . . . . 1 . . . . . . . . . . . . 1 2 . . . .
EXOSC10 . . . . . . . . . . . . . . . . . . . . . . . . .
MTOR . . . . . . . . . . . . . . . . . . . . . . . . .
UBIAD1 . . . . . . . . . . . . . . . . . . . . 1 . . . .
FBXO2 . . . . . . . . . . . . . . . . . . . . . . . . .
FBXO44 . . . . . . . . . . . . . . . . . 1 . . . . . . .
FBXO6 . . . 1 . . . . . . . . . . . . . . . . . . . . .
MAD2L2 . . . . . . . . . . . . . . . . . . . 1 . . . . .
DRAXIN . . . . . . . . . . . . . . . . . . . . . . . . .
AGTRAP . . . . . . . . . . . . . 1 . . 2 . . . 1 . . . 1
MTHFR . . . . . . . . . . . . . . . . . . . . 1 . . . .
CLCN6 . . . . . . . . . . . . . . . . . . . 1 . . 1 . .
NPPA-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
KIAA2013 . . . . . . . . . . . . . . . . . . . . . . . . .
PLOD1 . . . . . . . . . . . . . . . . . . . . . . . . .
MFN2 . . . . . . . . . . . . . . . . . . . . . . . . .
MIIP . . 1 . . . . . . . . . . . . . . . . . 1 . . . .
TNFRSF8 . . . . . . . . . . . . . . . . . . 2 . . . . . .
TNFRSF1B . 1 1 1 . . . . . . . . . . . . . . . . . . . . .
VPS13D . . . . . . . . . . . . . . . . . . . . . . . . .
DHRS3 . . . . . . . . . . . . . . . . . . . . . . . . .
PRDM2 . . . . . . . . . . . . . . . . . . . . . . 1 . .
TMEM51 . . . . . . . . . . . . . . . . . . . . . . . . .
RP3-467K16.4 . . . . . . . . . . . . . . . . . . . . . . . . .
EFHD2 1 . . 1 . 1 . . . 1 . . . 1 . . . . 1 . . . 1 . .
CASP9 . . . . . . . . . . . . . . . . . . . . . . . . .
DNAJC16 . . . . . . . . . . . 1 1 . . . . . . . . . . . .
AGMAT . . . . . . . . . . . . . . . . . . . . . . . . .
DDI2 . . . . . . . . . . . . . . . . . . . . . . . . .
PLEKHM2 . . . . . . . . . . . . . . . . . . . . . . . . .
FBLIM1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-169K16.9 . . . . . . 1 . . . . . . . . . . . . . . . . . .
SPEN . 1 . . . . . . . . . . . . . . . . . . . . . . .
ZBTB17 . . . . . . . . . . . . . . . . . . . . . . . . .
ARHGEF19 . . . . . . . . . . . . . . . . . . . . . . . . .
RSG1 . 1 . . . . . . . . . . . . . . . . . . . . . . .
FBXO42 . . . . . . 1 . . . . . . . . . . . . . . . . . .
SZRD1 . . . 1 . . . . . . . . . . . . 1 . . . . . . . .
SPATA21 . . . . . . . . . . . . . . . . . . . . . . . . .
NECAP2 1 . . . . . . . . . . . . . . . . 2 . . 2 . 1 . .
RP4-798A10.2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP4-798A10.7 . . . . . . . . . . . . . . . . . . . . . . . . .
RP4-798A10.4 . . . . . . . . . . . . . . . . . . . . . . . . .
NBPF1 . . . . . . . . . . . . . . . . . . . . . . . . .
CROCCP2 . . . 1 . . . 1 . . . 1 . . . . . . . . . . . . .
CROCC . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-108M9.4 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-108M9.6 . . . . . . . . . . . . . . . . . . . . . . . . .
ATP13A2 . . . . . . . . . . . . . . . . . . . . . . . . 1
SDHB . 1 . . . . . . . . . . . . . . . . . 1 . . . . .
PADI2 . . . . . . . . . . . . . . . . . . . . . . . . .
PADI4 . . . . . . . . . . . . . . . . . . . . . . . . .
RCC2 . . . . . . . . . . . . . . . . . . . . . . . . .
ARHGEF10L . . . . . . . . . . . . . . . . . . . . . . . . .
ALDH4A1 . . . . . . . . . . . . . . . . . . . . . . . . .
IFFO2 . . . . . . . . . . . . . . . . . . . . . . . . .
UBR4 . . 1 . . . . . . . . . . . . . . . 1 . 1 . . . 1
RP1-43E13.2 . . . . . . . . . . . . . . . . . . . . . . . . .
EMC1 . . . . . . . . . . . . . . . . . . . . . 1 . . .
MRTO4 . . . . . . . . . . . . . . . . . . . . . . . . .
AKR7A2 1 . . . . . . . 1 . . 1 . . 1 . 3 . . . . . 1 . .
PQLC2 . . . . . . . . . . . . . . . . . . . . . . . . .
CAPZB 1 1 3 1 2 . 1 2 . 1 1 . . 1 . . . . 2 2 . . 3 1 .
MINOS1-NBL1 . . . . . . . . . . . . . . . . . . . . . . . . .
MINOS1 . . 1 . . . . . . . 1 1 2 . . . . . . . 1 . . . .
NBL1 . . . . . . . . . . . . . . . . . . . . . . . . .
TMCO4 . . . . . . . . . . 1 . . . . . . . . . . . . . .
OTUD3 . . . . . . . . . . . . . . . . . . . . . . . . .
UBXN10-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
UBXN10 . . . . . . . . . . . . . . . . . . . . . . . . .
CAMK2N1 . . . . . . . . . . . . . . . . . . . . . . . . .
MUL1 . . . . . . . . . . 1 . . . . . . . . . . . . . .
CDA . . . 3 . . . . . . . . . 1 . . 2 . . 1 . . . . .
PINK1 . . . . . . . . . . . 1 . . . . . . . . . . . . .
PINK1-AS . . . . . . . . . . . . . . . . . . . . . . . . .
DDOST . . . . . . . . . . . . . 2 . . . . . . . . . . .
HP1BP3 . 1 . . . . . . 1 1 . . . . 1 1 . . . . . . . . .
RP5-930J4.4 . . . . . . . . . . . . . . . . . . . . . . . . .
EIF4G3 . . . . . . . . . . . . . . . . . . . . . . . . .
ECE1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP3-329E20.2 . . . . . . . . . . . . . . . . . . . . . . . . .
NBPF3 . . . . . . . . . . . . . . . . . . . . . . . . .
ALPL . . . . . . . . . . . . . . . . . . . . . . . . .
USP48 . 1 . 1 . . . . . . . . . . . . . . . . . . . . 2
HSPG2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP1-224A6.3 . . . . . . . . . . . . . . . . . . . . . . . . .
LINC00339 . . . . . . . . . . . . . . . . . . . . . . . . .
CDC42 . . 1 2 1 1 1 . . . . . . 1 1 . 1 1 . . . . . . 1
ZBTB40 . . . . . . . . . . . . . . . . . . . 1 . . . . .
C1QA . . . . . . . . . . . . . . . . . . . . . . . . .
C1QC . . . . . . . . . . . . . . . . . . . . . . . . .
C1QB . . . . . . . . . . . . . . . . . . . . . . . . .
EPHB2 . . . . . . . . . . . . . . . . . . . . . . . . .
KDM1A . . . . . . . . . . . . . . . . . . . . . . . . .
LUZP1 . . . . . 1 . . . . . . . . . . . . . . . . . . .
RP5-1057J7.6 . . . . . . . . . . . . . . . . . . . . . . . . .
HNRNPR . . 1 . . . . . . . 1 . . . . . . . . . . . . . .
ZNF436 . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf213 . . . . . . . . . . . . . . . . . . . . . . . . .
TCEA3 . . . . . . . . . . . . . . . . . . . . . . . . 1
E2F2 . 1 . . . . . . . . . . . . . . . . . . . . . . .
ID3 . . . . . . . . . . . . . . . . . . . . . . . . .
MDS2 . . . . . . . . . . . . . . . . . . . . . . . . .
RPL11 41 39 24 19 3 14 17 20 4 6 43 33 14 16 7 10 11 10 64 23 47 7 26 9 16
TCEB3 . . . . . . . . . . . . . . . . . . . . . . . . .
RP5-886K2.3 1 . . . . . . . . . . . . . . . . . . . . . . . .
PITHD1 1 . . . . . . . . . . 1 . . . . . . . . . . . . .
LYPLA2 1 . . . . . . . . . . . . . . . . . . . . . . . .
GALE . . . . . . . . . . . . . . . . . . . . . . . . .
HMGCL . . . . . . . . . . . . . . . . . . . . . . . 1 1
FUCA1 . . . . . . . . . . . . . . . . . . . . . . . . .
CNR2 . . . . . . . . . . . . . . . . . . . . . . . . .
PNRC2 . 1 1 . . . . . . . . . . . . . . . . . . . . . .
SRSF10 . . . . . . . . . . . . . 1 . . . . 1 . . . . . .
IFNLR1 . . . . . . . . . . . . . . . . . . . . . . . . .
STPG1 . . . . . . . . . . . . . . . . . . . . . . . . .
NIPAL3 . 1 . . . . . . . . . . . . . . . . . . . . . . .
RCAN3 . . . . . . . . . . . 1 . . . . . . . 1 . . . . .
RP4-594I10.3 . . . . . . . . . . . . . . . . . . . . . . . . .
SRRM1 . . . . 1 . . . 1 . . . . . . . . . . . . . 1 . .
CLIC4 . . . . . . . . . . . . . . . . . . . . . . . . .
RUNX3 . . . . . . . . . . . . . . . . . . . . . . . 1 .
SYF2 . . . . 1 1 . . . . 3 1 . 1 . 1 2 . 3 . 1 . . . .
C1orf63 1 1 . . . . . . . . 1 2 . . . . . . . 2 . . . . .
RP3-465N24.6 . . . . . . . . . . . . . . . . . . . . . . . . .
TMEM50A . . 2 . 1 . . . . . . . 1 . . . . 2 . . . . . . .
TMEM57 . . . . . . . . . . . . . . . . . . . . . . . . .
LDLRAP1 . . . . . . . . . . . 2 1 . . . . . . . . . . . .
RP11-70P17.1 . . . . . . . . . . . . . . . . . . . . . . . . .
MAN1C1 . . . . . . . . . . . . . . . 1 . . . . . . . . .
RP1-187B23.1 . . . . . . . . . . . . . . . . . . . . . . . . .
SEPN1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP1-317E23.3 . . . . . . . . . . . . . . . . . . . . . . . . .
MTFR1L . . . 1 . . . . . . . . . . . . . . . . . . . . .
AL020996.1 . . . . . . . . . . . . . . . . . . . . . . . . .
PAQR7 . . . . . . . . . . . . . . . . . . . . . . . . .
STMN1 . . . . . . . . . . . . . . . . . . . . . . 15 . .
PAFAH2 . . . . . . . . . . . . . . . . . . . . . . . . .
PDIK1L . . . . . . . . . . . . . . . . . . . . . . . . .
ZNF593 . . . . . . . . 1 1 . . . . . . . . . . . . . . .
CNKSR1 . . . . . . . . . . . . . . . . . . . . . . . . .
CEP85 . . . . . . . . . . . . . . . . . . . . . . . . .
SH3BGRL3 2 5 6 12 3 3 2 . 2 1 1 1 1 6 2 . 2 3 17 3 6 1 1 1 10
UBXN11 . . . . . . . . . . . . . 1 . . . . . . . . 1 . .
CD52 6 8 13 4 . 2 . 2 1 2 8 3 3 3 3 . . 1 14 10 6 1 7 1 .
AIM1L . . . . . . . . . . . . . . . . . . . . . . . . .
ZNF683 . . . . . . . . . . . . . . . . . . . . 1 . . . .
DHDDS . . . . . . . . . . . . . 1 . . . . . . 1 . . . .
HMGN2 . . . . . . . . . 1 . . . . . . . . . . . . 1 . .
RPS6KA1 . . 1 . . . . . . . . . . . . . 1 . 1 . . . . . .
ARID1A . . 1 . . . . . . . 1 . . . . . . . . . . . . . .
PIGV . . . . . . . . . . . . . . . . . . . . . . . . .
ZDHHC18 . . . . . . . . . . . . . . . . . . . . . . . . .
GPN2 . . . . . . . . . . . . 1 . 1 . . . . . . . . . .
GPATCH3 . . . . . . . . . . . . . . . . . . . . . . . . .
NUDC . 2 2 . . . . . . . . . 1 . . . . . . . 1 . . . .
C1orf172 . . . . . . . . . . . . . . . . . . . . . . . . .
TRNP1 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC9A1 . . . . . . . . . . . . . . . . . . . . . . . . .
WDTC1 . . . . . . . . . . . . . . . . . . . . . . . . .
TMEM222 . . . . . . . . . . . . . . . . . . . . . . . . .
SYTL1 . . 2 . . . . . . 1 . . . . . . 1 . 1 1 . . . . .
MAP3K6 . . . . . . . . . . . . . . . . . . . . . . . . .
WASF2 . 2 . . . 1 . . . . . 1 1 . . . . . 1 . 1 . . 1 1
RP1-159A19.4 . . . . . . . . . . . . . . . . . . . . . . . . .
AHDC1 . . . . . . . . . . . . . . . . . . . . . . . . .
FGR . 1 . . . . . . . 2 . . . 3 . . 4 . 4 . . . . . .
IFI6 . 1 . 1 . . . . . 1 . 1 . 2 . 1 1 . 4 . 1 . 1 . .
FAM76A . . . . . . . . . . . . . . . . . . . . . . . . .
STX12 . . . . . 1 . 1 . . . . . . . 1 . . . . . . . . .
PPP1R8 . . . . . . . . . . . . . . . . . . . . . . . . .
THEMIS2 . . . . . . . . . . . . . . . . 2 . 2 . . . . . .
RPA2 . . . . . . . . . . 1 1 1 . . . . . . . . . 1 . .
XKR8 . . . . . . . . . . . . 1 . . . . . . . . . . . .
EYA3 . . . . . . . . . . . . . . . . . . . . . . . . .
PTAFR . . . . . . . . . . . . . 1 . . . . . . . . . . .
DNAJC8 . . . . . 1 . . . . . . . . . . . 1 2 . 1 . 2 . .
AL353354.2 . . . . . . . . . . . . . . . . . . . . . . . . .
ATPIF1 . . 2 . . . . . . . . . 1 . . . . . 1 . . . . 1 .
RP5-1092A3.4 . . . . . . . . . . . . . . . . . . . . . . . . .
SESN2 . . . . . . . . . . . . . . . . . . . . . . . . .
MED18 . . . . . . . . . . . . . . . . . . . . . . . . .
PHACTR4 . . . . . . . . . . . . . . . . . . . . . . . . .
RCC1 . 1 . . . . . . . . . . . . . . . . . . . . . . .
TRNAU1AP . . . . . . . . . 1 . . . . . . . . . . . . . . .
SNHG12 . . . . . . . . . . . . . . . . . . . 1 . 1 . . .
TAF12 . . 1 . . 1 . . . . . . . 1 . . . 1 . . . . . . .
RP11-442N24--B.1 . . . . . . . . . . . . . . . . . . . . . . . . .
RNU11 . . . . . . . . . . . . . . . . . . . . . . . . .
GMEB1 . . . . . . . . . . . . . . . . . . . . . . 1 . .
YTHDF2 . . . . . . . . . . . . . . . . . . 1 . . . . . .
EPB41 . . . . . . . . . . . . . 1 1 . . . . . . . . . .
TMEM200B . . . . . . . . . . . . . . . . . . . . . . . . .
SRSF4 . . . . . . . . . . . . . . . . . . . . 2 . . . .
MECR . . . . . . . . . . . . . . . . . . . . . . . . .
MATN1 . . . . . . . . . . . . . . . . . . . . . . . . .
MATN1-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
LAPTM5 . 9 16 2 . 2 . 3 1 2 . 2 3 4 . . 1 . 3 2 4 3 4 1 .
PUM1 . . 1 . . . . . . . . . . . . . . . . . . . . . .
SNRNP40 . . 1 . . . . . 1 . . . . . . . . . . . 1 . . . .
ZCCHC17 . . . . . . . . . . . . 1 . . . . 1 . . 1 . . . .
SERINC2 . . . . . . . . . . . . . . . . . . . . . . . . .
PEF1 . . . . . . . . . . . . . . . . . . 5 . . . . . .
SPOCD1 . . . . . . . . . . . . . . . . . . . . . . . . .
PTP4A2 1 1 1 . . 1 . . . . . 1 . . . . . . . . 1 1 . . .
KHDRBS1 . . . . . 1 . . 1 . . . . 1 . . . 1 1 . . . . . .
TMEM39B . . . . . . . . . . . . . . . . . . . . . . . . .
KPNA6 . . . . . . . . . . . . . . . . . . . . . . . . .
TXLNA . . . . . . . . . . . . . . . . . . . . . . . . .
CCDC28B . . . . . . . . . . . . . . . . . . . . . . . . .
TMEM234 . . . . . . . . . . . . . 1 . . . . . . . . . . .
EIF3I 1 1 1 1 . . 1 . . . 1 . 1 1 . . . . 1 1 1 . . . .
FAM167B . . . . . . . . . . . . . . . . . . . . . . . . .
LCK . . . . . . 1 . . . . 2 . . . . . . . . . . . 1 .
HDAC1 . . . . 1 . . . . . . . . . . . . . . . . . . . .
MARCKSL1 . . . . . . . . . 1 . . 1 . . . . . . 1 2 1 . . .
FAM229A . . . . . . . . . . . . . . . . . . . . . . . . .
BSDC1 . . . . . . . . . . . 1 . . . . . . . . . . . . .
ZBTB8A . . . . . . . . . . . . . . . . . . . . . . . . .
ZBTB8OS . 1 . . . . . . . . . . . . 1 . . . . . 1 . . . .
RBBP4 . 1 . . . . . . . . . . . . . . . . . . . . . . 1
SYNC . . . . . . . . . . . . . . . . . . . . . . . . .
YARS . . . . . . . . . . . . . . . . . . . . . . . . .
S100PBP . . . . . . . . . . . . . . . . . . . . . . . . .
RNF19B . . . . . . . . . . . . . . . . . . . . . . . . .
AK2 . . . . 1 . . . . . . . . . . . . . . . 1 . . . .
ADC . . . . . . . . . . . . . . . . . . . . . . . . .
TRIM62 . . . . . . . . . . . . . . . . . . . . . . . . .
ZNF362 . . . . . . . . . . . . . . . . . . . . . . . . .
PHC2 . . . . . . . . . . . . . . . . . . . . . . . . .
SMIM12 . . . 1 1 . . . . . . . . . . . . 1 . . . 1 . . .
DLGAP3 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-244H3.4 . . . . . . . . . . . . . . . . . . . . . . . . .
ZMYM6NB . . . . . . . . . . . . . . . . . . . . . . . . .
ZMYM6 . . . . . . . . . . . . . . . . . . . . . . . . .
ZMYM1 . . . . . . . . . . . . . . . . . . . . . . . . .
SFPQ 1 . . . . . . . . . 1 1 . . 1 . . . 1 . . . 2 . .
ZMYM4 . . 1 . . . . . . . . . . . . . . . . . . . . . .
KIAA0319L . . . . . . 1 . . . . . . . . . . . . . . . . . .
NCDN . . . . . . . . . . . . . . . . . . . . . . . . .
RP4-728D4.2 . . . . . . . . . . . . . . . . . . . . . 1 . . .
TFAP2E . . . . . . . . . . . . . . . . . . . . . . . . .
PSMB2 . 1 . . . . . . 1 . . . 1 . . . . . . . 1 . . . .
C1orf216 . . . . . . . 1 . . . . . . . . . . . . . . . . .
CLSPN . . . . . . . . . . . . . . . . . . 2 . . . . . .
AGO4 . . . . . . . . . . . . . . . . . . . . . . . . .
AGO1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP4-789D17.5 . . . . . . . . . . . . . . . . . . . . . . . . .
AGO3 . . . . . . . . . . . . . . . . . . . . . . . . .
ADPRHL2 . . . . . . . . . . . . . 1 . . . . . . . . . . .
TRAPPC3 . 1 . . 1 . . . . . 1 . . . . . . 1 1 . . . . . .
MAP7D1 . . . . . . . . . . . . . . . . . . . 1 . . . . 1
THRAP3 . . . . . . . . . . . 1 . . . . . . . . . . . . .
SH3D21 . . . . . . . . . . . . . . . . . . . . . . . . .
EVA1B . . . . . . . . . . . . . . . . . . . . . . . . .
STK40 . . . . . . . . . . . . . . . . . . 1 . . . . . .
LSM10 . 3 . . 1 . . . . . . . . 1 . . 2 1 . . 1 1 . . .
OSCP1 . . . . . . . . . . . . . . . . . . . . . . . . .
MRPS15 . 2 . . 1 . . . . . . . . 1 . 1 . . . . . . . . .
CSF3R . . . . . . . . . . . . . 5 . . . . . . . . . . .
LINC01137 . . . . . . . . . . . . . . . . . . . . . . . . .
ZC3H12A . . 1 . . . . . . . . . . . . . . . . . . . . . .
MEAF6 1 1 . . . 1 . . 1 . . . 1 . . . . . . . 3 . . . .
SNIP1 . . . . . . . . . . . . . . . . . . . . . . . . .
GNL2 . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf109 . . . . . . . . . . . . . . . . . . . . . . . . .
CDCA8 . . . . . . . . . . . . . . . . . . . . . . . . .
YRDC . . . . . . . . . . . . . . . . . . . 1 . . . . .
C1orf122 . . . . . . . . . . . . . . . . . . 1 . . . . . .
MTF1 . . . . . 1 . . . . . . . . . . . . . . . . . . 1
INPP5B . 1 . . . . . . . . . . . . . . . . . . . . 1 . .
SF3A3 . . . 1 . . . . . . . . . . . . . . 1 . . . . . .
FHL3 . . . . . . 2 . . . . . . . . . . . . . . . . . .
UTP11L . . . . . . . . . . 1 . . . . . . . . . . . . . .
RRAGC 1 1 . . . . 1 . . . . . 2 . . . . . . . . . . . .
RP5-864K19.4 . . . . . . . . . . . . . . . . . . . . . . . 1 1
MYCBP . 1 . . . . . . . . . . . . . . . 1 . . 1 . . . 1
AKIRIN1 . . . . . . 1 . 1 . . . . . . . . 1 . . . . 1 . .
NDUFS5 . 2 3 . 1 . . . . . . 2 1 . . 1 . . . 2 . . . . .
MACF1 . . . . . . . . . . . . . . . . . . 1 . . . . . .
KIAA0754 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-69E11.4 . . . . . . . . . . . . . . . . . . . . . . . . .
PABPC4 . . . . . . . . . . . . 1 . . . . . 1 . . . . . .
HPCAL4 . . . . . . . . . . . . . . . . . . . . . . . . .
PPIE . . 1 . . 1 1 . . . . . . . . . . . . . . . . . .
TRIT1 . . . . . . . . . . . 1 . . . . . . . . . . . . .
MYCL . . . . . . . . . . . . . . . . . . . . . . . . .
MFSD2A . . . . . . . . . . . . . . . . . . . . . . . . .
CAP1 . 2 . . . 2 . . 1 . . . . 1 . . . 2 4 . 2 1 . . .
PPT1 1 . 1 . . . . . . . . 1 . . . . 1 . . 1 . . . . .
RLF . . . . . . . . . . . . . . . . . . . . . . . . .
RP1-39G22.7 . . . 1 . . . . . . . . . . . . . . . . . . . . .
ZMPSTE24 . . . . . . . . . . . . . . . . . 1 . . . . . . .
COL9A2 . . . . . . . . . . . . . . . . . . . . . . . . .
SMAP2 . . . . . . . . . . 1 . . . . . 1 . . 1 1 . . . .
ZFP69B . . . . . . . . . . . . . . . . . . . . . . . . .
ZFP69 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-656D10.3 . . . . . . . . . . . . . . . . . . . . . . . . .
EXO5 . . . . . . . . . . . . . . . . . . . . . . . . .
ZNF684 . . . . . . . . . . . . . . . . . . . . . . . . .
RIMS3 . . . . . . . . . . . . . . . . . . . . . . . . .
NFYC-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
NFYC . . . . . . . . . . . . . . . . . . . . . . . . 1
CITED4 . . . . . . . . . . . . 1 . . . . . . . . . . . .
CTPS1 . . . . . . . . . . . . . . . . . . . . . . . . .
SLFNL1 . . . . . . . . . . . . . . . . . . . . . . . . .
SCMH1 . . . . . . . . . . . . . . . . . . . . . . . . .
HIVEP3 . . . . . . . . . . . . . . . . . . . . . . . . .
FOXJ3 . . . . . . . . . . . . . . . . . . . . . . . . .
PPCS . 3 . . . . 1 1 . . . . . . . . 1 . 1 . . . 1 . 1
CCDC30 . . . . . . . . . . . . . . . . . . . . . . . . .
PPIH . . . . . . . . . . . . . . . . . . . 1 . . . . .
YBX1 2 7 4 8 1 2 . 1 . 3 6 2 2 5 . . . 1 6 1 6 2 4 . 3
LEPRE1 . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf50 . . . . . . . . . . . . . . . . . 1 . . . . . . .
CCDC23 . . . . . . . . . . . 1 . 10 . . . . . 1 . . . . .
ERMAP . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-342M1.3 . . . . . . . . . . . . . . . . . . . . . . . . .
ZNF691 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC2A1 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC2A1-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
EBNA1BP2 1 . . . . . . . 1 . . . . . . . . . . . . . . . .
WDR65 . . . . . . . . . . . . . . . . . . . . . . . . .
RP1-92O14.3 . . . . . 1 . . . . . . . . . . . . . . . . . . .
CDC20 . . . . . . . . . . . . . . . . . . . . . . . . .
ELOVL1 . . 1 . . . . . . . . . . . . . . . . . 1 . 2 . .
MED8 . . . . 1 . . . . . 1 . . . . . . . . . . . . . .
SZT2 . . . . . . . . . . . . . . . . . . . . . . . . .
HYI . . . . . . . . . . . . . . . . . . . . . . . . .
KDM4A . . . . . . . . . . . . . . . . . . . . . . . . .
KDM4A-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
ST3GAL3 . . . . . . . . . . . . . . . . . . . . . . . . .
IPO13 . . . . . . . . . . . . . . . . . . . . . . . . .
DPH2 . . . . . . . . . . . . . . . . . . . . . . . . .
ATP6V0B . 2 . 1 1 . . . . 2 1 . . 1 . . 1 . 2 . 1 . 1 . .
CCDC24 . . . . . . . . . . . . . . . . . . . . . . . . .
DMAP1 . . . . . . . . . . . . . . . . . . 1 . . . 1 . .
ERI3 . . . . . . . . . . . . . . . . . . . . . . . . .
RNF220 . . . 1 1 . . . . . . . . . . . . . . . . . . . .
TMEM53 . 1 . . . . . . . . . . . . . . . . . . . . . . .
C1orf228 . . . . . . . . . . . . . . . . . . . . 1 . . . .
KIF2C . . . . . . . . . . . . . . . . . . . . . . . . .
RPS8 15 62 34 5 3 7 9 12 27 1 29 16 9 9 2 2 5 5 16 8 23 9 14 8 4
BEST4 . . . . . . . . . . . . . . . . . . . . . . . . .
PLK3 . . 1 . . . . 1 . . . . 1 1 . . . . . . . . . . .
BTBD19 . . . . . . . . . . . . . . . . . . . . . . . . .
PTCH2 . . . . . . . . . . . . . . . . . . . . . . . . .
EIF2B3 . . . . . . . . . . . . 1 . . . . . . . . . . . .
HECTD3 . . . . . . . . . . . . . . . . . . . . . . . . .
UROD . . . . . . . . . . . . . . . . . . . . . . . . .
HPDL . . . . . . . . . . . . . . . . . . . . . . . . .
MUTYH . . . . . . . . . . . . . . . . . . . . . . . 1 .
TOE1 . . . . . . . . . . . . . . . . . . . . . . . . .
TESK2 . . . . . . . . . . . . . . . . . . . . . . . . .
CCDC163P . . . . . . . . . . . . . . . . . . . . . . . . .
PRDX1 . . . 1 . 2 . . . 1 . 1 . . . . . . 1 . . . . . .
AKR1A1 . . . . . . . . . . 1 . 1 . . . 1 . 1 . . . 1 . .
NASP 1 . . 1 . . . . . . 1 . . . . . . . . . 1 1 . . .
GPBP1L1 . . 1 . . . . . . . . . . . . . . . . . 1 1 . . .
TMEM69 . . . . . . . . . . . . . . . . . . . . . . . . .
IPP . . . . . . . . . . . . . . . . . . . . . . . . .
MAST2 . . . . . . . . . . . . . . . . . . . . . . . . .
PIK3R3 . . . . . . . . . . . . . . . . . . . . . . . . .
POMGNT1 . . . . . . . . . . . . . . . . . . . . . . . . .
LRRC41 . . . . . . . . . . . . . . . . . . . . . . . . .
UQCRH 1 1 2 . . 1 1 . . . 4 . 1 1 . 1 . . 1 . 1 . . . 2
NSUN4 . . . . . . . . . . . . . . . . . . . . . . . . .
FAAH . . . . . . . . . . . . . . . . . . . . . . . . .
MKNK1 . . . . . . . . . . . . . . . . 1 . . . . . . . .
MOB3C . . 1 . . . . . . . . . . . . . . . . . . . . . .
ATPAF1 . . . . . . . . . . . . . . . . . . . . . . . . .
EFCAB14 . . . . . . . 1 . . . . . . . . . . . . 1 . . . .
PDZK1IP1 . . . . . . . . . . . . . . . . . . . . . . . . .
TAL1 . . . . . . . . . . . . . . . . . . . . . . . . .
CMPK1 1 . . . . . . 1 . . 1 1 1 1 . . . . . . 1 1 . 1 .
RP11-511I2.2 . . . . . . . . . . . . . . . . . . . . . . . . .
FOXD2-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
FOXD2 . . . . . . . . . . . . . . . . . . . . . . . . .
SPATA6 . . . . . . . . . . . . . . . . . . . . . . . . .
BEND5 . . . . . . . . . . . . . . . . . . . . . . . . .
FAF1 . . . . . . . . . . . . . . . . . . . . . . . . .
CDKN2C . . . . . . . . . . . . . . . . . . . . . . . . .
RNF11 1 1 . . . . . . . . . . 1 . . . . . . . . . . . .
EPS15 . . . . . . . . . . . . . . . . . . . 1 1 . . . .
OSBPL9 . . . . . . . . . 1 . . . . . . . . . 1 . . . . .
NRD1 . . . . . . . . . 1 . . . . . . . . 1 . . . . . .
TXNDC12 . . 1 . 1 . 1 . 1 2 . . . . 1 1 . . 1 . . . 1 . .
KTI12 . . . . . . . . . . . . . . . . . . . . 1 . . . .
BTF3L4 . . . . . . . . . . . . 1 . . . . . . . . . . . .
ZFYVE9 . . . . . . . . . . . . . . . . . . . . . . . . .
CC2D1B . . . . . . . . . . . . . . . . . . . . . . . . .
ORC1 . . . . . . . . . . . . . . . . . . . . . . . . .
PRPF38A . . . . . . . . . . . . . . . . . . . . . . . . .
ZCCHC11 . . 1 . . . . . . . . . . . . . . . . . . . 1 . .
GPX7 . . . . . . . . . . . . . . . . . . . . . . . . .
FAM159A . . . . . . . . . . 1 . 1 . . . . . . 1 . . . . .
COA7 . . . . . . . . . . . . . . . . . . . . . . . . .
ZYG11B . . . . . . 1 . . . . . . . . . . . . . . . . . .
ECHDC2 . . 1 . . 2 1 . . . 1 2 . . . . . . . . . . . . .
SCP2 1 1 . 1 . 1 . . 1 1 . . . . . . 1 . . . . . . . 1
PODN . . . . . . . . . . . . . . . . . . . . . . . . .
SLC1A7 . . . . . . . . . . . . . . . . . . . . . . . . .
CPT2 . . 1 . . . . . . . . . . . . . . . . . . . . . .
C1orf123 . 1 . . . . . . . . . . . . . . . . . 1 1 . . . .
RP5-1024G6.7 . . . . . . . . . . . . . . . . . . . . . . . . .
MAGOH . . . . . . . 1 . . . . . 2 . 1 . . 2 . 1 . . . .
RP4-784A16.5 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-117D22.2 . . . . . . . . . . . . . . . . . . . . . . . . .
NDC1 . . . . . . . . . . . . . . . . . . . . . . . . .
YIPF1 . . . . . . . . . . . . . . . . 1 . . . . . . . .
HSPB11 . . . . . . . . 1 . . . . 1 . . . . . . . . . . .
LRRC42 . . . . . . . . . . . . . . . . . . . . . . . . .
TMEM59 1 1 1 . . 1 1 . . . . . . 2 . . . . 1 1 1 . . . .
TCEANC2 . . . . . . . . . . 1 . . . . . . . . . . . . . .
CYB5RL . . . . . . . . . . . . . . . . . . . . . . . . .
MRPL37 . . . . . . . . . . . 1 . . . . . . . . . . . . .
SSBP3 . . . . . . . . . . . . . . . . . . 1 . . . . . .
ACOT11 . . . . . . . . . . . . . . . . . . . . . . . . .
TTC4 . . . . . . . . . . . . . . . . . . . . . . . . .
PARS2 . . . . . . . . . . . . . . . . . . . . . . . . .
TTC22 . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf177 . . . . . . . . . . . . . . . . . . . . . . . . .
DHCR24 . . . . . . . . . . . . . . . . . . . . . . . . .
USP24 . . . . . . . . . . . . . . . . . . . . . . . . .
DAB1 . . . . . . . . . . . . . . . . . . . . . . . . .
OMA1 . . . . . . . . . . . . . . . . . . . . . . 1 . .
MYSM1 . . . . . . . . . . . . . . . . . . . . . . . . .
JUN 11 . 13 1 . . . . . . 5 3 2 2 7 . 1 . 6 1 2 . 8 . 3
LINC01135 . . . . . . . . . . . . . . . . . . . . . . . . .
RP4-794H19.1 . . . . . . . . . . . . . . . . . . . . . . . . .
FGGY . . . . . . . . . . . . . . . . . . . . . . . . .
HOOK1 . . . . . . . . . . . 1 . . . . . . . . . . . . .
CYP2J2 . . . . . . . . . . . . . . . . . . . . . . . . .
NFIA . . . . . . . . . . . . . . . . . . . . . . . . .
TM2D1 . 1 1 . . 1 . 1 . . 1 . . . . . . . 1 . . . . . .
INADL . . . . . . . . . . . . . . . . . 1 . . . . . . .
L1TD1 . . . . . . . . . . . . . . . . . . . . . . . . .
USP1 . 1 . . . . . . . 1 1 . . . 1 . . . . . 1 . . . .
DOCK7 . . . . . . . . . . . . . . . . . . . . . . . . .
ATG4C . . . . . . . . . . . . . . . . . . . . . 1 . . .
ALG6 . . . . . . . . . . . . . . . . . . . . . . . . .
ITGB3BP . . . . . . . . . . . . . . . . . . . . . . . . .
EFCAB7 . . . . . . . . . . . . . . . . . . . . . . . . .
DLEU2L . . . . . . . . 1 . . . . . . . . . . . . . . . .
PGM1 . 1 . . . . . . . . . . . . . . . . . 1 . . . 1 .
RAVER2 . . . . . . . . . . . . . . . . . . . . . . . . .
JAK1 . 1 . . . . . . . 1 . . 1 . . . . . 1 1 . . . . 1
AK4 . . . . . . . . . . . . . . . . . . . . . . . . .
LEPR . . . . . . . . . . . . . . . . . . . . . . . . .
LEPROT . . . . . . . . . . 1 . . . . . 2 . 1 . . . . . .
PDE4B . . . . . . . . . . . . . . . . . . . . . . . . .
MIER1 . . . . . . . . . . . . . . . . . 1 . . . . . . .
SLC35D1 . . . . . . 1 . . . . . . . . . . . . . . . . . .
IL23R . . . . . . . . . . . . . . . . . . . . . . . . .
IL12RB2 . . . . . . . . . . . . . . . . . . . . . . . . .
SERBP1 . 1 1 1 . 1 . 1 . . 1 . . 1 . . . . 1 1 . . . . .
GADD45A . . . . . . . . . . . . . . . . . . . . . . . . .
WLS . . . . . . . . . . . . . . . . . . . . . . . . .
LRRC7 . . . . . . . . . . . . . . . . . . . . . . . . .
LRRC40 . . . . . . . . . . . . . . . . . . . . . . . . .
SRSF11 . . . 1 . . 1 . . . . . . . . . . . . . . . . . .
ANKRD13C . . . . . . . . . . . . . . . . . . . . . . . . .
HHLA3 . . . . . . . . . . . . . . . . . . . . . . . . .
CTH . . . . . . . . . . . . . . . . . . . . . . 1 . .
ZRANB2 . . 1 . . . 1 . . . . . . . . . . . . . . 1 . . .
LRRIQ3 . . . . . . . . . . . . . . . . . . . . . . . . .
FPGT . . . . . . . . . . . . . . . . . . . . . . . . .
CRYZ . . . . . . . . . . . . 1 . . . . . . . . . . . .
TYW3 . . . . . . . . . . . . . . . . . . . . . . . . .
ACADM . . . . . . . . . . . . . . 1 . . . . 1 . . . . .
RABGGTB . 1 . . . . . . 1 . . 1 . . . . . 1 . . . . . . .
ST6GALNAC3 . . . . . . . . . . . . . . . . . . . . . . . . .
PIGK . . . . . . . . . . . . . . . . . . . . . . . . 1
AK5 . . . . . . . . . . . 1 . . . . . . . . . . . . 1
ZZZ3 . . . . . . . . . . . . . . . . . . . . . . . . .
USP33 . . 2 . . . . . . . . . . . . . . . . . . . . . .
FAM73A . . . . . . . . . . . . . . . . . . . . . . . . .
NEXN . . . . . . . . . . . . . . . . . . . . . . . . .
FUBP1 . . . . . . . . . . 2 . . . . . . . . 1 1 . 2 . .
DNAJB4 . 1 . . . . . . . . . . . . . . . . . 1 . . . . .
RP11-386I14.4 . . . . . . . . . . . . . . . . . . . . . . . . .
IFI44L . . . . 1 . . . . . 1 . . . . . . . . . 1 . . . .
IFI44 . . . . . 1 . . . . . . . . . . . . . . . . 1 . .
RP5-887A10.1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-475O6.1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-486G15.2 . . . . . . . . . . . . . . . . . . . . . . . . .
PRKACB . 1 . . . . . . . . . . . . . . . . . . . . . . .
RPF1 . . . . . . . . . . . . . . . . . . . . . . . . .
GNG5 . 1 1 . . . . . . 1 . . . 1 . . . . . . 1 . . 3 .
CTBS . . . . . . . . . . . . . . . . . . . . . . . . .
SSX2IP . . . . . . . . . . . . . . . . . . . . . . . . .
MCOLN2 . 1 . . . . . . . . . . . . . . . . . . . . . . .
MCOLN3 . . . . . . . . . . . . . . . . . . . . . . . . .
SYDE2 . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf52 . . . . . . . . . . . 1 1 . . . . . . . . . . . .
BCL10 . . . . . . . . . . . . . . . . . . . . 1 . . . .
RP11-131L23.1 . . . . . . . . . . . . . . . . . . . . . . . . .
ZNHIT6 . . . . . . . . . . . . . . . . . . . 3 . . . . .
ODF2L . . . . . 1 1 . . . . . 1 . . . . . . . . . . 1 .
RP4-612B15.3 . . . . . . . . . . . . . . . . . . . . . . . . .
SH3GLB1 . 1 . . . . . . . . . . . . . . . 1 . . . . . . .
SEP15 1 1 2 . 1 . . . . . . . 1 1 . . . 1 . 2 . 1 . . 1
HS2ST1 . . . . . . . . . . . . . . . . . . . . . . . . .
LMO4 . . . 1 . . . . . . . . . . . . . . . 1 . . . . .
PKN2 . . . . . . . . . . . . . . . . . . . . . . . . .
GTF2B . . . . . 1 . . . . . . . . . . . . . . . . . . .
CCBL2 . . . . . . . . . . . . . . . . . . 1 . . . . . .
RBMXL1 . . . . . 1 . . . . . . 1 . . . . . . . . . . . .
GBP3 . . . . . . . 1 . . . . . . . . . . . . . . . . .
GBP1 . . . . . . . . . . . 1 1 . . . . . . . . . . . .
GBP2 . . 3 . . . 16 . . . . . . . . . . . . . 2 . . . .
GBP4 . . 1 . . . . . . . . . . . . . . . . . . 1 . . .
GBP5 1 1 . . . . . 1 . . . . . . . . . 1 . . . . . . .
LRRC8B . . . . . . . . . . . . . . . . . . . . . . . . .
RP5-1007M22.2 . . . . . . . . . . . . . . . . . . . . . . . . .
LRRC8C . . . . . . . . . . . . . . . . . . . . . . . . .
RP5-943J3.2 . . . . . . . . . . . . . . . . . . . . . . . . .
LRRC8D . . . . . . . . . . . . . . . . . . . . . . . . .
ZNF326 . 1 . 1 . . . . . . . . . . . . . . . . . . . . .
ZNF644 . . . . . . . . . . . . . . . . . 1 . 1 . . . . .
CDC7 . . . . . . . . . . . . . . . . . . . . . . . . .
TGFBR3 . . . . . . . . . . . . . . . . . . . . . . . . .
BTBD8 . . . . . . . . . . . . . . . . . . . . . . . . .
KIAA1107 . . . . . . . . . . . . . . . . . . . . . . . . .
GLMN . . 1 . . . . . . . . . . . . . . . . . . . . . .
RPAP2 . . . . . . . . . . . . . . . . . . . . 1 . . . .
GFI1 . . . . . . . 1 . . . . . . . . . . . . . . . . .
EVI5 . . . . . . . . . . . . . . . . . . . . . . . . .
RPL5 5 17 7 3 . 5 9 4 3 . 27 18 7 4 4 6 2 2 12 2 12 6 19 5 5
FAM69A . . . . . . . . . . . . . . . . . . . . . . . . .
MTF2 . . 1 . . . . . . . . . . . . 1 . . . . 2 . . . .
TMED5 . . . . . . . . . . . . . . . . . . 1 . 1 . . . .
CCDC18 . . . . . . . . . . . . . . . . . . 1 . . . . . .
DR1 . . . . . . . . 1 . . . . . . . . . . . . . 1 . 1
FNBP1L . . . . . . . . . . . . . . . . . . . . . . . . .
RP4-561L24.3 . . . . . . . . . . . . . . . . . . . . . . . . .
DNTTIP2 . 1 . . . . . . . . . . . . . . . . . . . . 1 . .
GCLM . . . . . 1 . . . . . . . . . . 1 . . . . . . . .
ARHGAP29 . . . . . . . . . . . . . . . . . . . . . . . . .
ABCD3 . . . . . . . . . . . . . . . . . . . . . . . . .
CNN3 . . . . . . . . . . . . . . . . . . . . . . . . .
ALG14 . . . . . . . . . . . . . . . . . . . . . . . . .
RWDD3 . . . . . . . . . . . . . . . . . . . . . . . . .
PTBP2 . . . . . . . . . . . . . . . . . . . . . . . . .
DPYD . 1 . . . . . . . . . . . . . . . . . . . . . . .
AGL . . . . . . . . 1 . . . . . . . . . . . . . . . .
RP5-884G6.2 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC35A3 . . . . . . . 1 . . . . . . . . . . . . . . . . .
HIAT1 1 . . . . . . . . . . . . . . . 1 . . . 1 . . . .
SASS6 . . . . . . . . . . . . . . . . . . . . . . . . .
TRMT13 . . . . . . . . . . . . . . . . . . . . . . . . .
DBT . . . . . . . . . . . . . . . . . . . . . . . . .
RTCA . . . . . . . . . . . . . . . . . . . . . . . 1 .
CDC14A . . . . . . . . . . . . . . . . . . . . . . . . .
EXTL2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP4-549L20.3 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC30A7 . . . . . . . . . . . . . . . . . . . . . . . . .
DPH5 . . . . . . . . . . 18 . . . . . . . . 1 . . . . .
AC093157.1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-421L21.2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-421L21.3 . . . . . . . . . . . . . . . . . . . . . . . . .
RP4-575N6.4 . . . . . . . . . . . . . . . . . . . . . 1 . . .
S1PR1 . . . . . . . . . . . . . . . . . . . . . . . . .
RNPC3 . . . . . . . . . . . . . . . . . . . . . . . . .
AMY2B . . . . . . . . . . . . . . . . . . . . . . . . .
PRMT6 . . . . . . . . . . . 1 . . . . . . . . . . . . .
VAV3 . . . . . . . . . . . . . . . . . . . . . . . . .
VAV3-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-356N1.2 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC25A24 . . . 1 . . . . . . . . . . . . . . . . . . . . .
RP11-483I13.5 . . . . . . . . . . . . . . . . . . . . . . . . .
FAM102B . . . . . . . . . . . . . . . . . . . . . . . . .
HENMT1 . . . . . . . 2 . . . . . . . . . . . . . . . . .
PRPF38B . . 2 . . 1 . . . . . 1 1 1 . . . . . . 1 . . . .
STXBP3 . . . . . . . . . . . . . . . . . . . . 1 . . . .
GPSM2 . . . . . . . . . . . . . . . . . . . . . . . . .
CLCC1 . . . . . . . . . . . . . . . . . . . . . . . . .
WDR47 . . . . . . . . . . . . 1 . . . . . . 1 . . . . .
TAF13 . . . . . . . . . . . . . . . . . . . . . . . . .
RP5-1065J22.8 . . . . . . . . . . . . . . . . . . . . . . . . .
TMEM167B . . . . . . . . . . . . . . . . . . . . . . . . .
KIAA1324 . . . . . . . . . . . . . . . . . . 1 . . . . . .
SARS . . . . . . . . . 1 . . . . . . . . . . . . . . .
CELSR2 . . . . . . . . . . . . . . . . . . . . . . . . .
PSRC1 . . . . . . . . . . . . . . . . . . . . . . . . .
SORT1 . . . . . . . . . . . . . . . . . . . . . . . . .
PSMA5 . . . . . . . . . . . 1 . 1 . 1 . . . . . . . . .
ATXN7L2 . . . . . . . . . . . . . . . . . . . . . . . . .
CYB561D1 . . . . . . . . . . . . . . . . . . . . . . . . .
AMIGO1 . . . . . . . . . . . . . . . . . . . . . . . . .
GPR61 . . . . . . . . . . . . . . . . . . . . . . . . .
GNAI3 . . . . . 1 . . . . . . 1 . . . . . 1 . . . . . .
AMPD2 . . 1 . . . . . . . . . . . . . . . 2 . . 1 . . .
GSTM4 . . . 1 . . . . . . . . . . . . . . . . . . . . .
GSTM2 . . . . . . . . . . . . . . . . . . . . . . . . .
GSTM1 . . . . . . . . . . . . . . . . . . . . . . . . .
GSTM3 . . . . . . . . . . . . . . . . . . . . . . . . .
CSF1 . . . . . . . . . . . . . . . . . . . . . . . . .
AHCYL1 . . . . . . . . . . . . . 1 . . . . . . . . . . .
STRIP1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP4-773N10.4 . . . . . . . . . . . . . . . . . . . . . . . . .
KCNC4 . . . . . . . . . . . . . . . . . . . . . . . . .
RBM15 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC16A4 . . . . . 1 . . . . . . . . . . . . . . . . . . .
RP5-1074L1.4 . . . . . . . . . . . . . . . . . . . . . . . . .
LAMTOR5-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
LAMTOR5 . 1 . . . . . . . . 2 . . . . . . . 1 . . 1 . . 1
RP11-284N8.3 . . . . . . . . . . . . . . . . . . . . . . . . .
KCNA3 . . . . . . . . . . . . . . . . . . . . . . . . .
CD53 1 2 1 1 . . . . 2 . 3 . 2 . . . . . 1 . 1 . 1 . .
RP11-96K19.2 . . . . . . . . . . . . . . . . . . . . . . . . .
LRIF1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-96K19.5 . . . . . . . . . . . . . . . . . . . . . . . . .
DRAM2 . 2 . . . . . . . . 1 . . . . . . . 1 . . . . . .
CEPT1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP5-1180E21.5 . . . . . . 1 . . . . . . . . . . . . . . . . . .
DENND2D . . . . . . . 1 . . . . . . . . . . . . 1 . . . .
CHI3L2 . . . . . . . . . . 1 . . . . . . . . . . . . . .
PIFO . . . . . . . . . . . . . . . . . . . . . . . . .
OVGP1 . . . . . . . . . . . . . . . . . . . . . . . . .
WDR77 . . . . . . . . . . . . . . . . . . . . . . . . .
ATP5F1 . 1 . . . . . . . 1 . . 1 . . . . . 5 1 . . . . 2
C1orf162 . . . 1 1 . . . . 1 . . . 1 . 1 2 . 7 . . . 1 . 1
ADORA3 . . . . . . . . . . . . . . . . . . . . . . . . .
RAP1A . . 1 . 1 . 1 . . . 1 2 . 1 1 . . . . . 2 1 1 1 .
FAM212B . . . . . . . . . . . . . . . . . . . . . . . . .
DDX20 . . . . . . . . . . . . . . . . . . . . . . . . .
CTTNBP2NL . . . . . . . . . . . . . . . . . . . . . . . . .
WNT2B . . . . . . . . . . . . . . . . . . . . . . . . .
ST7L . . . . . . . . . . . . . . . . . . . . . . . . .
CAPZA1 . . . . . . . . . . . . . . . . . . . 1 . . 1 . .
MOV10 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-426L16.3 . . . . . . . . . . . . . . . . . . . . . . . . .
RHOC . . . . . . . . . 1 . . 1 . . . . 1 2 . . . . . .
PPM1J . . . . . . . . . . . . . . . . . . . . . . . . .
SLC16A1 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC16A1-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-389O22.1 . . . . . . . . . . . . . . . . . . . . . . . . .
LRIG2 . . . . . . . . . . . . . . . . . . . . . . . . .
MAGI3 . . . . . . . . . . . . . . . . . . . . . . . . .
PHTF1 . . . . . . . . . . . . . . . . . . . . . . . . .
RSBN1 . . . . . . . . . . . . . 1 . . . . . . . . . . .
PTPN22 . . . . . . . . . . 1 . . . . . . . . . . . . . .
AP4B1-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
BCL2L15 . . . . . . . . . . . . . . . . . . . . . . . . .
AP4B1 . . . . . . . . . . . . . . . . . . . . . . . . .
DCLRE1B . . . . . . . . . . . . . . . . . . . . . . . . .
RP5-1073O3.7 . . . . . . . . . . . . . . . . . . . . . . . . .
HIPK1 . . . . . . . . . . . . . . . . . . . . . . . . .
TRIM33 . 1 . . . . . . . . . . . . . . . . . . . . . . .
BCAS2 . . . . . . . 1 1 . . . . 1 . . . . . . . . . . .
DENND2C . . . . . . . . . . . . . . . . . . . . . . . . .
NRAS . . . . . . . . . . . . . . . . . . . . . . . . .
CSDE1 1 1 2 . . . . 1 . . 2 1 1 . 1 . . . . 1 . . . . .
SIKE1 . 1 . . . . . 1 . . . . . . . . . . . . . . 1 1 .
TSPAN2 . . . . 1 . . . . . . . . . . . . . . . . . . . .
VANGL1 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC22A15 . . . . . . . . . . . . . . . . . . . . . . . . .
ATP1A1 . . . 2 . . . . . . . . 1 . . . . . . 1 1 . . . .
AL136376.1 . . . . . . . . . . . . . . . . . . . . . . . . .
CD58 . . . . . . . 1 . . 1 . . . . . . . . . 1 . . . .
CD2 . . 5 1 . . . 2 . . . 2 1 . . . . . . 1 . . . . .
CD101 . . . . . . . . . . . . . . . . . . . . . . . . .
TTF2 . . . . . . . . . . . . . . . . . . . . . . . . .
MAN1A2 . . . 1 . . . . . . 1 . . . . . . . . . . . . . .
FAM46C . . 2 . . . . . . . . . . . . . . . . 1 . . . . .
GDAP2 . . . . . . . . . . . . . . . . . . . . . . . 1 .
WDR3 . . . . . . . . . . . . . . . . . . 1 . . . . . .
WARS2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-418J17.1 . . . . . . . . . . . . . . . . . . . . . . . . .
PHGDH . . . . . . . . . . . . . . . . . . . . . . . . .
REG4 . . . . . . . . . . . . . . . . . . . . . . . . .
NOTCH2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-114O18.1 . . . . . . . . . . . . . . . . . . . . . . . . .
FAM72B . . . . . . . . . . . . . . . . . . . . . . . . .
FCGR1B . . . . . . . . . . . . . 1 . . . . . . . . . . .
RP11-343N15.5 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-344P13.6 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-423O2.5 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-782C8.1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP6-206I17.1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP6-206I17.2 . . . . . . . . . . . . . . . . . . . . . . . . .
LINC00623 . . . . . . . . . . . . . . . . . . . . . . . . .
AL592284.1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-640M9.1 . . . . . . . . . . . . . . . . . . . . . . . . .
NBPF9 . . . . . . . . . . . . . . . . . . . . . . . . .
PDE4DIP . . . . . . . . . . . . . . . . . 1 . . . . . . .
AL590452.1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-458D21.6 . . . . . . . . . . . . . . . . . . . . . . . . .
NOTCH2NL . . . . . . . . . 1 1 . . . . . . . 1 . . . . . .
NBPF10 . . . . . . . . . . 1 . . . . . . . . . . . . . .
TXNIP 1 . 1 2 2 . 1 3 1 . . 3 1 1 . . . 2 6 2 3 2 1 . 2
POLR3GL . . 1 . . . . . . . . . . . . . . . . 1 . . . . .
ANKRD34A . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-315I20.1 . . . . . . . . . . . . . . . . . . . . . . . . .
LIX1L . . . . . . . . . . . . . . . . . . . . . . . . .
RBM8A . 1 1 . 1 1 . . 1 1 1 . . 2 . . . . 1 . 2 . 1 1 .
PEX11B . . . . . . . . . . . . . . . . . . . . . . . . .
ITGA10 . . . . . . . . . . . . . . . . . . . . . . . . .
ANKRD35 . . . . . . . . . . . . . . . . . . . . . . . . .
PIAS3 . . . . . . . . . . . . . . . . . . . . . . . . .
NUDT17 . . . . . . . . . . . . . . . . . . . . . . . . .
POLR3C . . . . . . . . . . . . . . . . . . 1 . . . . . .
RNF115 1 . 1 . . . . . . . . . . . . . . . . . 2 . 1 . .
CD160 . . . . . . . . . . . . . . . . . . . . . . . . .
GPR89A . . . . . . . . . . . . . . . . . . . . . . . . .
NBPF12 . . . . . . . . . . 1 . . . . . . . . . . . . . .
PRKAB2 . . . . . . . . . . . . . . . . . . . . 1 . . . .
RP11-337C18.8 . . . . . . . . . . . . . . . . . . . . . . . . .
FMO5 . . . . . . . . . . . . . . . . . . . . . . . . .
CHD1L . . . . . . . . . . . . . . . . . . . . . . . . .
BCL9 . 1 . . . . 1 . . . . . . . . . . . . . . . . . .
ACP6 . . . . . . . . . . . . . . . . . . . . . . . . .
NBPF14 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-89F3.2 . . . . . . . . . . . . . . . . . . . . . . . . .
NBPF20 . . . . . . . . . . . . . . . . . . . . . . . . .
NBPF15 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-666A1.5 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-14N7.2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-403I13.4 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-403I13.8 . . . . . . . . . . . . . 1 . . . . . . . . . . .
RP11-277L2.3 . . . . . . . . . . . . . . . . . . . . . . 1 . .
RP11-353N4.4 . . . . . . . . . . . . . . . . . . . . . . . . .
FCGR1A . . . . . . . . . . . . . 1 . . . . 1 . . . . . .
HIST2H2BF . . . . . . . . . . . . . . . . . . . . . 1 . . .
RP11-196G18.24 . . . . . . . . . . . . . . . . . . . . . . . . .
HIST2H2BE . . . . . . 1 . . . . . . . . . . . . . . . . . .
HIST2H2AC . 1 . . . . . . . . . . . . . . . . . . . . . . .
HIST2H2AB 1 . . . . . . . . . . . . . . . . . . . . 1 . . .
BOLA1 . . . . . . . . . . . . . . . . . . 1 . . . . . .
SV2A . . . . . . . . . . . . . . . . . . . . . . . . .
SF3B4 . . . . 1 . . . . . . . . . . . . . . . . . . . .
MTMR11 . . . . . . . . . . . . . . . . . . 1 . . . . . .
OTUD7B . . . . . . . . . . . . . . . . . . . . . . . . .
VPS45 . . . . . . . . . . . . . . . . . . . . 1 . . . .
PLEKHO1 . . . . . . . . . . . . . . . . . . . . 1 . . . .
ANP32E . . . . . . . 1 . . . . . . . . . . . . . . . . .
APH1A . 1 . . . . . . . . . . . 1 . . 1 . . 1 . . . . .
C1orf54 . . . . . . . . . . . . . 1 . . . . . . . . . . .
C1orf51 . . . . . . . . . . . . . . . . . . . . . . . . .
MRPS21 1 . . 1 . . . . . . 1 . . . . . . . 3 2 . . . 1 .
PRPF3 . . . . . 1 . . . . . . . . . . . . . . . . . . .
RPRD2 . . . . . . . . . . . . . . . . . . . . . . 1 . .
TARS2 . . . . . . . . . . . . . . . . . . . . . . . . .
ADAMTSL4 . . . . . . . . . . . . . . . . . . . . . . . . .
MCL1 1 . . 1 . 1 . 1 . . . . . 3 1 . 1 . . 2 . . 1 . 5
ENSA . 1 . 1 . . . . . . 2 . 1 . . . . . . . . 1 . . .
GOLPH3L . . . . . . . . . . . . 1 . . . . . . . . . . . .
CTSS . 4 1 3 . 1 . . . 1 3 . . 7 . . . 1 11 . 1 . 2 1 3
CTSK . . . . . . . . . . . . . . . . . . . . . . . . .
ARNT . . . . . . . . . . . . . . . . 1 . . . . . . . .
SETDB1 . . . . . . . . . . . . . . . . . . . . . . . . .
CERS2 . . 1 . . . . . 1 . . . . . . . . . 1 . . . . . 1
FAM63A . . . . . . . . . . . . . . . . . . . . . . . . .
PRUNE . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf56 . . . . . . . . . . . . . . . . . . . . . . . . .
CDC42SE1 . . . 1 . . . 1 1 . . . 1 . . . . . . 1 . 1 . . .
MLLT11 . . . . . . . . . . . . 1 . . . . . . . . . . . .
GABPB2 . . . . . . . . . . . 1 . . . . . . . . . . . . .
TNFAIP8L2 . . . . . 2 . . . . . . . . . . . . . . . . . . 1
SCNM1 . . 1 . . . . 3 . . . . 1 . . . . . . . 1 . . . .
VPS72 . . . . . . . . . . . . . . . . . . 1 1 . . . . .
PIP5K1A . 1 . . . . . . . . . . . . . . . . . . . . . . .
PSMD4 1 1 1 . . 1 . . . 1 . . . . . . . . 1 1 . . 1 . .
RP11-126K1.2 . . . . . . . . . . . . . . . . . . . . . . . . .
ZNF687 . . . . . . . . . . . . . . . . . . . . . . . . .
PI4KB . . . . . . . . . 1 . 1 . . . . . . . . . . . . .
RFX5 . 1 . . . 1 . . . . . . . . . . . . . . . . . . .
RP11-126K1.6 . . . . . . . . . . . . . . . . . . . . . . . . .
PSMB4 1 . 1 . . 1 . . . . . . . . . . . . 1 1 . . . . .
POGZ . . . . . . . . . . . . . 1 . . . . . . . . . . .
TUFT1 . . . . . . . . . . . . . . . . . . . . . . . . .
SNX27 . . . . . . . . . 1 . . . . . . . . . . . . . . .
MRPL9 1 . . . . . . . . . . 1 . . 1 . . . . . . . . . .
OAZ3 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-98D18.3 . . . . . . . . . . . . . . . . . . . . . . . . .
TDRKH . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-98D18.9 . . . . . . . . . . . . . . . . . . . . . . . . .
RORC . . . . . . . . . . . . . . . . . . . . . . . . .
C2CD4D . . . . . . . . . . . . . . . . . . . . . . . . .
THEM4 1 . . . . . . . . . . . . . . . . . . . . . . . .
S100A10 . 3 . 2 . 3 2 . . 3 . . 4 6 1 . 3 . 2 2 1 . . 1 1
S100A11 . . 1 7 . 2 1 . . 4 1 . 2 5 . . 2 1 15 . 1 . . . 5
S100A9 . . . 12 . . . . . . . . . 34 1 . 20 . 26 . . . . 1 143
S100A12 . . . . . . . . . . . . . . . . 1 . . . . . . . .
S100A8 . . . 3 . . . . . 3 . . . 4 . . 8 . 5 . . . . . 79
S100A6 . 2 3 16 . 3 8 16 1 2 . 1 3 16 1 . 4 3 29 1 5 . . . 9
S100A5 . . . . . . . . . . . . . . . . . . . . . . . . .
S100A4 3 . 5 16 5 9 2 4 3 5 1 1 7 27 1 . 5 3 27 12 12 . . . 16
S100A13 . . . . . . . . . . . . . . . . . . 1 . . . . . .
CHTOP . . . . . . . . . . . . . 1 . . . . . 1 1 . . 1 .
SNAPIN . . . . . . . . . . . . . . . . . . 1 . . . 1 . .
ILF2 2 1 . . . . 2 . . . 1 . . . . . . . 1 1 . . 1 . .
INTS3 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC27A3 . 1 . . . . . . . . . . . 1 . . 1 . 1 . . . . . .
GATAD2B . . . . . . . 1 . . . . . . . . . . . . . . . . .
DENND4B . . . . . . . . . . . . . . . . 1 . . . . . . . 1
CRTC2 . . . . . . . . . . 1 . . . . . . . . . . . . . .
SLC39A1 . . . . . . . 1 . . . . . . . . . . . . . . . . .
CREB3L4 . . . . . . . . . . . . . . . . . . . . . . . . .
JTB 1 . 2 . . 1 . 1 . 1 . 1 1 . . . 1 . 3 1 1 . . . .
RP11-422P24.11 . . . . . . . . . . . . . . . . . . . . . . . . .
RAB13 . . . . . . . . . . . . . 1 . . . . . . . . . . .
RPS27 15 27 30 21 1 21 18 21 13 3 16 16 13 7 15 6 7 3 11 21 47 11 26 19 10
TPM3 . 2 1 2 2 . 1 . . 1 . . . . 1 . . . 2 . 5 . 2 . .
C1orf43 2 . 1 . . . . . . . . . . 1 1 . . . 1 . 1 . . . .
UBAP2L . . . 1 1 . 1 . . . . . . . . . . . . . . . . . .
HAX1 . 1 . . . . . . . . . . . . . . . . 1 . . . . . 1
AQP10 . . . . . . . . . . . . . . . . . . . . . . . . .
ATP8B2 . . . . . 1 . . . . . . . . . . . . . . . . . . .
RP11-350G8.5 . . . . . . . . . . . . . . . . . . . . . . . . .
IL6R . . . . . . . . . . . . . . . . . . . . . . . . .
UBE2Q1 . . . . . . . . . . . . . . . . . . . . . . . . .
ADAR . . . . . . 1 1 1 . . . . 1 . . . . . . . . . . .
PMVK 1 . . . . . . 1 . . 2 . . . . . . . 1 . . . . . .
RP11-307C12.13 . . . . . . . . . . . . . . . . . . . . . . . . .
PBXIP1 . . 2 . . 2 . . . . . . . . . . . 1 . 1 13 . . . .
PYGO2 . . . . . . . . . . . . . . . . . . . . . . 1 . .
RP11-307C12.12 . . . . . . . . . . . . . . . . . . . . . . . . .
SHC1 . . . . . . . . . . . . . . . . . . . . . . . . .
CKS1B . . . . 1 . . . . . . . . . . . . . . . . . . . .
FLAD1 . . . . . . . . . . . . . . . . . . . . . . 1 . .
ZBTB7B . . 1 . . . . . . . . . . 1 . . . . . . . . . . .
ADAM15 . . . . . . . . . . . . . . . . . . . . . . . . 1
EFNA4 . . . . . . . . . . . . . . . . . . . . . . . . .
EFNA3 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC50A1 . . . . . . . . . . . . . . . . . . 1 . . . . . .
DPM3 . . . . . . . . . . . . . . . . . . . . 1 . . . .
KRTCAP2 . . . 1 . . . 1 . . 1 . . . . . . . . 1 . . . . 1
TRIM46 . . . . . . . . . . . . . . . . . . . . . . . . .
THBS3 . . . . . . . . . . . . . . . . . . . . . . . . .
MTX1 . . . 1 . . . . . . . . . . . . . . . . 1 . . . .
GBA . . . . . . . . . . . . . . . . . . . . . . . . .
FAM189B . . . . . . . . . . . . . . . . . . . . . . . . .
SCAMP3 . . . . . . . . . . . . . . . . . . . . 2 . . . .
CLK2 . . . . . . . . . . . . . . . . . . . . . . . . .
FDPS . . . 1 . . . . . . 4 . . 1 . . . . . . . . . . .
RUSC1-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
RUSC1 . . . 1 . . . . . . . . . . . . . . . . . . . . .
ASH1L . . . . . . . 1 . . . . . . . . . . . . . . . . .
MSTO1 . . . . . . . . . . . . . . . . . . . . . . . . .
YY1AP1 . . . . . . . . . . . . . . . . . . . . 1 . . . .
DAP3 . . . . . . . . . . . . . . . . . 1 . 1 . . . . .
GON4L . . 1 . . . . . . . . . . . . . . . . . . . . . .
SYT11 . . . . . . . . . . . . . . . . . . . . . . . . .
RIT1 . . . . . . . . . . . . . . . . . . . . . . . . .
KIAA0907 . 1 . . . . . . . . . . . . 1 . . . . 1 2 . . . .
ARHGEF2 . . 1 . . . . . . . 1 . . . . . . . 1 . . . . . .
RP11-336K24.12 . . . . . . . . . . . . . . . . . . . . . . . . .
SSR2 1 1 1 . 2 1 . 1 1 1 . . 3 1 1 1 . . 4 . 4 . . 2 1
UBQLN4 . . . . . . . . . . . . . . . . . . . . . . . . .
LAMTOR2 . . . 1 . 1 . . . 1 . . . . . . 2 . 2 . . . . . .
RAB25 . . . . . . . . . . . . . . . . . . . . . . . . .
LMNA . . . . . . . . . . . . . . . . . . . . 2 . . . .
SEMA4A . . . . . . . . . . . . . . . . . . . . 1 . . . .
SLC25A44 . . . . . . . . . . . . . . . . . . . . . . . . .
PMF1 . . . . . . . . . . . . . 1 . . . . . . . . . . .
BGLAP . . . . . . . . . . . . . . . . . . . . . . . . .
SMG5 . . . . . . . . . . . . . . . . . . . . . . . . .
TMEM79 . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf85 . . . . . . . . . . . . . . . . . . . . . . . . .
CCT3 1 . . . . . . . . . . 2 . . . 1 . . 1 . . . 1 . .
C1orf61 . . . . . . . . . . . . . . . . . . . . . . . . .
MEF2D . . . . . . . . . . . . . . . . . . . . . . . . .
TTC24 . . . . . . . . . . . . . . . . . . . . . . . . .
APOA1BP . . . . . 1 . . . . 1 . . 1 . . . . 2 1 1 . . . .
GPATCH4 . . . . . . . . . . . . 1 . . . . . . . . . . . .
ISG20L2 . . . . . . . . . . . . . . . . . . . . . . . . .
RRNAD1 . . . . . 1 . . . . . . . . . . . . . . . . . . .
MRPL24 . . . . . . . . . . . . . . . . . . . . . . . . .
HDGF . . . . . . . . . . . . . . . . . . . . . . . . .
PRCC . . . . . . . . . . . . . . . . . . . . . . . . .
SH2D2A . . 1 . . 1 1 1 . . . . . . . . . . . . . . . . .
ARHGEF11 . . . . . . . . . . . . . . . . . . . . . . . . .
ETV3 . . . . . . . . . . . . . . . . . . . . . . . . .
FCRL5 . . . . . . . . . . . . . . . . . . . . 3 . . . .
FCRL3 . . . . . . . . . . . . . . . . . . . . . . . . .
FCRL2 . 1 . . . . . . . . . . . . . . . . . . . . . . .
FCRL1 . . . . . . . . . . . . . . . . . . . . . 2 . 1 .
CD1D . . . . . . . . . . 2 . . . . . . . . . . . . . .
CD1A . . . . . . . . . . . . . . . . . . . . . . . . .
CD1C . 2 . . . . . . . . 5 . . . . . . . . . 1 . 1 . .
CD1B . . . . . . . . . . . . . . . . . . . . . . . . .
CD1E . . . . . . . . . . . . . . . . . . . . . . . . .
MNDA . . . 2 . . . . . . . . . 1 . . . . 1 . . . . . 1
PYHIN1 1 . . . . . . 1 . . . . 1 . . . . . . 1 . . . . .
IFI16 . . . . . . . . . . . . . . . . . . . . . . . . .
AIM2 . 1 . . . . . . . . . . . . . . . . . . . . . . .
FCER1A . . . . . . . . . . . . . . . . . . . . . . . . .
DUSP23 . . . . . . . . . . . . . 2 . . . . . . . . . . 2
FCRL6 . . . . . . . . . . . . . . . . . . . . . . . . 1
SLAMF8 . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf204 . . . . . . . . . . . . . . . . . . . . . . . . .
VSIG8 . . . 1 . . . . . . . . . . . . . . . . . . . . .
TAGLN2 1 7 2 2 . 1 1 . . 2 . . 1 1 . 1 . 2 . . 2 2 . . .
PIGM . . . . . . . . . . . . . . . . . . . . . . . . .
IGSF8 . . . . . 1 . . . . . . 1 . . . . . . . . . . . .
RP11-536C5.7 . . . . . . . . . . . . . . . . . . . . . . . . .
PEA15 . . . . . . . . . . . . . . . . . . . . . . . . .
DCAF8 . 1 . . . . . . . . . . . . . . . . . . . . . . .
PEX19 . . . . . . . . . . . . . . . . . . . . . . 1 . .
COPA . . . . . 1 . . . . . . . . . . . . . . . . . . 1
NCSTN . . . 1 . . . . . . . . . . . . . . . . . . . . .
SLAMF6 . . . . . . . . . . 2 . . . . . . . . . . . . . .
CD84 . . 1 . . 1 . . . . 1 . . . . . . . . . . . . . .
SLAMF1 . . . . . . . . . . . . . 1 . . . . . 1 . . . . .
RP11-404F10.2 . . . . . . . . . . . . . . . . . . . . . . . . .
CD48 4 1 1 . . 1 . 1 2 . 1 1 3 . . 2 1 . 2 1 3 . 2 . 1
SLAMF7 . . . . . . . . . . . . . . . . . . . . . . . . .
LY9 . . . . . . . 1 . . 1 1 . . . . . . . . . . . . .
CD244 . . . . . . . . . . . . . . . . 1 . . . . . . . .
ITLN1 . . . . . . . . . . . . . . . . . . . . . . . . .
F11R . . . . . . . . . . . . . . . . . . . . . . . . .
TSTD1 . 1 . . . . . 1 . . . . 1 . . 1 . 1 . 1 . . . . .
USF1 . . . . . . . . . . . . . . . . . . . . . . . . .
ARHGAP30 . . . . . . . 1 . . . . . 1 . . . . . . . . . . .
KLHDC9 . . . . . . . . . . . . . . . . . . . . . . . . .
PFDN2 . . 1 . . . . . . . . . . 1 . . . . 1 . 1 . . . 1
NIT1 . . . . . . . . . . . . . . . . . . . . . . . . .
DEDD . . . . . . . . . . . . . . . . . . . . . 1 . . 1
UFC1 1 1 . 1 . . 1 . . . 1 2 . 2 . . 1 . . 1 1 . 1 1 .
USP21 . . . . . . . . . . . . . . . . . . . . . . . . .
PPOX . . . . . . . . . . . . . . . . . . . . . . . . .
B4GALT3 . . 1 . 1 . . . . . . . . . . . . . . . . . . . .
NDUFS2 . . 2 1 . . . . . . . 2 . . . . . . . 1 . . . . 2
FCER1G . . . 7 1 . . . . 5 . . . 2 . . . . 9 2 . . . . 2
TOMM40L . . . 1 . . . . . . . . . . . . . . . . . . . . .
MPZ . . . . . . . . . . . . . . . . . . . . . . . . .
SDHC . . 1 . . . . . . . . 1 . . . . 1 . . . 1 . . . .
FCGR2A . . . 1 . . . . . . . . . 2 . . . . . . 1 . . . .
HSPA6 . . . . . . . . . . . . . . . . . . . . . . . . .
FCGR3A . . . 1 . . . . . 1 . . . . . . . . 3 . . . . . .
FCGR2B . . . . . . . . . . . . . . . . . . . . . . . . .
FCGR3B . . . . . . . . . 1 . . . . . . . . . . . . . . .
FCRLA . . . . . . . . . . 2 . . . . . . . . . 1 . 2 . .
FCRLB . . . . . . . . . . . . . . . . . . . . . . . . .
DUSP12 . . . . . . . . . . . . . . . . . . . . . . . . .
ATF6 . . . . . . . . . . . 1 . . . . . . . . . . . . .
SH2D1B . . . . . . . . . . . . . . . . . . . . . . . . .
UHMK1 . . . . . . . . . . . . . . . . . . . . . . 1 . .
RP11-359K18.3 . . . . . . . . . . . . . . . . . . . . . . . . .
UAP1 . 1 . . . . . 1 . . . . . . . . . . . . 1 . . . .
HSD17B7 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-267N12.3 . . . . . . . . . . . . . . . . . . . . . . . . .
NUF2 . . . . . . . . . . . . . . . . . . . . . . . . .
PBX1 . . . . . . . . . . . . . . . . . . . . . . . . .
MGST3 . . . . . . . . 1 . . . 1 1 . 1 . 1 . . . . . . 1
ALDH9A1 . . 1 . . 1 . . . . 1 . . . . . . . 1 . . . . . .
TMCO1 . 1 1 1 . . . . . . . . . . . . . . 1 . . 1 1 . .
RP11-466F5.8 . . . . . . . . . . . . . . . . . . . . . . . . .
UCK2 . . . . . . . . . . . . . . . . . . . . . . . . .
POGK . . . . . . . . . . . . . . . . . . . . . . . . .
TADA1 . . . . . . . . . . . . . . . . . . . . 1 . . . .
GPA33 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-277B15.3 . . . . . . . . . . . . . . . . . . . . . . . . .
POU2F1 . . . . . . . . . . . . . . . . . . . . . . . . .
CD247 . . 1 . . . 1 . . . . . 1 . . 1 . . . . 2 . . . .
RP11-104L21.3 . . . . . . . . . . . . . . . . . . . . . . . . .
CREG1 . . . . . . . . . . . . . . . . . . . . . . . . 1
RP3-455J7.4 . . . . . . . . . . . . . . . . . . . . . . . . .
RCSD1 . 1 . . . . . . . . . . 1 . . . 1 . . 1 2 1 2 . .
MPZL1 . . . . . . . . . . . . . . . . . . . . . . . . .
MPC2 1 1 . . . . . . . . . . . . . . . . . . . . . . 1
DCAF6 . . . . . . . . . . . . . . . . . . . 1 . . . . .
TIPRL . . . . . . . . . . . . . . . . . . . . . . . . .
SFT2D2 . . . . . 1 . . . . . . . . . . . . . . . . . . .
TBX19 . . . . . . . . . . . . . . . . . . . . . . . . .
XCL2 . . . . . . . . . . . . . . . . . . . . . . . . .
XCL1 . . . . . . . . . . . . . . . . . . . . . . . . .
ATP1B1 . . . . . . . . . . . . . . . . . . . . . . . . .
NME7 . . . . . . . . . . . . . . . . . . . . . . . . .
BLZF1 . . . . . . . . . . . . . . . . . . . . . 1 . . .
SLC19A2 . . . . . . . . . . . . . . . . . . . . . . . . .
F5 . . . . . . . . . . . . . . . . . . . . . . . . .
SELP . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf112 . . . . . . . . . . . . . . . . . . . . . . . . .
SELL . 4 2 . . 1 . . . . . 1 1 . . . . . . . 2 1 . . .
METTL18 . . . . . . . . . . . . . . . . . . . . . . . . .
SCYL3 . . . . . . . . . . . . . . . . . . . . . . . . .
KIFAP3 . . . . . . . . . . . . . . . . . . . . . . . . .
GORAB . . . . . . . . . . . . . . . . . . . . . . . . .
FMO4 . . . . . . . . . . . . . . . . . . . . . . . . .
PRRC2C . 1 . 1 . . . . . . . . . . . . . . 1 1 2 . . . .
VAMP4 . . . . . . . . . . . . . . . . . . . . . . . . .
METTL13 . . . . . . . . . . . . . . . . . . . . . . . . .
DNM3 . . . . . . . . . . . . . . . . . . . . . . . . .
PIGC . . . . . . . . . . . . . . . . . . . . 1 . . . 1
SUCO . . . . . . . . . . . . . . . . . . . . . . . . .
FASLG . . . . 1 . . . . . . . . . . . . . . . . . . . .
TNFSF4 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-296O14.3 . . . . . . . . . . . . . . . . . . . . 1 . . . .
PRDX6 . . . . 2 . . . . . . . . . . . . . . . . . . . 1
KLHL20 . . . . . . . . . . . . . . . . . . . . . . . . .
CENPL . . . . . . . . . . . . . . . . . . . . . . . . .
DARS2 . . . . . . . . . . . . . . . . . . . . . . . . .
ZBTB37 . . . . . . . . . . . . . . . . . . . . . . . . .
RC3H1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-160H22.5 . . . . . . . . . . . . . . . . . . . . . . . . .
RABGAP1L . . . . . . . . . 1 . . . 1 . . 1 . . . . . . . .
CACYBP . 1 1 . . . . . . . . . . . . 1 . . . . . . . . .
MRPS14 . . . . . . . . . . . . . 1 . . . . . 1 . . . . .
KIAA0040 . 2 . 1 . . . . . . 1 . . . . . . . . . . . 2 . .
RP11-318C24.2 . . . . . . . . . . . . . . . . . . . . . . . . .
RFWD2 . 1 . . . . . . . . . . . . . . . . . . . . . . .
RP5-1098D14.1 . . . . . . . . . . . . . . . . . . . . . . . . .
RALGPS2 . . . . . . . . . . . . . . . . . . . . . . 1 . .
FAM20B . . . . . . . . . . . . . . . . . . . . . . . . .
TOR3A . . . 1 . . . . . . . . . . . . 1 . . . . . . . .
ABL2 . . . . . . . . . . . 1 . . . . . . . . . . . . .
SOAT1 . . . . . . . . . . . . . . . . . . . . . . . . .
TOR1AIP2 . . . . . . . . . . . . 1 . . . . . . . . . . . .
RP11-533E19.7 . . . . . . . . . . . . . . . . . . . . . . . . .
TOR1AIP1 . . . . . . . . . . . . 1 . . . . . . . . . . . .
RP11-533E19.5 . . . . . . . . . . . . . . . . . . . . . . . . .
CEP350 . . 1 . . . . . . . . . . . . . . . . . . . . . .
QSOX1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP5-1180C10.2 . . . . . . . . . . . . . . . . . . . . . . . . .
ACBD6 . . . . . . . . . . 1 . . . . . . . . . . . . . .
XPR1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-46A10.5 . . . . . . . . . . . . . . . . . . . . . . . . .
STX6 . . . . . . . . . . . . . . . . . . . . . . . . .
MR1 . . . . . . . . 1 . . . . . . . . . . . . . . . .
IER5 . . . 1 . . . . . . . . . . . . . 1 . . . . . . .
RP11-309G3.3 . . . . . . . . . . . . . . . . . . . . . . . . .
GLUL . . . . . 2 . . . . . . . . . . . . . 1 1 . 1 . .
RNASEL . . . . . . . . . . . . . . . . . . . . . . . . .
RGS16 . . . . . . . . . . . . . . . . . . . . . . . . .
NPL . . . . . . . . . . . . . . . . . . . . . . . . .
DHX9 . . . . . . 1 . . . . . . . . . . . . . . . . . .
LAMC1 . . . . . . . . . . . . . . . . . . . . . . . . .
SMG7 . . . . . . . . . . . . . . . . . . . . . . . . .
NCF2 . . . . . . . 1 . . . . . 1 . . 1 . . . 1 . . . .
ARPC5 . 1 2 2 1 . . 1 . 2 . 2 1 . . . . . 1 . 1 . 2 . 1
RGL1 . . . . . . . . . . . . . . . . . . . . . . . . .
COLGALT2 . . . . . . . . . . . . . . . . . . . . . . . . .
TSEN15 . . . . . . . . . . . . . 1 . . . . . . . . . . .
C1orf21 . . . . . . . . . . . . . . . . . . . . . . . . .
EDEM3 . . . . . . . . . . . . . . . . . . . . . . . . .
FAM129A . . . . . . . . . . . . . . . . . . . 1 . . . . .
RNF2 . 1 . . . . . . . . . . . . . . . . . . . . . . .
TRMT1L . . . . . . . . . . . . . 1 . . . . . . . . . . .
SWT1 . . . . . . . . . . . . . . . . . . . . . . . . .
IVNS1ABP . . . . . . . . . . 1 . . . . . . 1 . . . . 1 . .
TPR . 2 1 . . . . . 1 1 . 2 1 . . . . 1 . . 1 . . . .
C1orf27 . . . . . . . . . . . . . . . 1 . . . . . . . . .
PTGS2 . . . . . . . . . . . . . . . . . . . . . . . . .
RGS18 . . . . . . . . . . . . . . . . . . 1 . . . . . .
RGS1 . . . . . . . . . . 1 . . . . . . . 1 1 . . . . .
RGS2 . . . 1 . . . . . 2 . . . 5 . . . . 3 1 1 . . . 1
UCHL5 . . . . . . . . . . . . 1 . . . . . . . . . . . .
TROVE2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-101E13.5 . . . . . . . . . . . . . . . . . . . . . . . . .
GLRX2 . . . . . . . . . . . . . . . . . . . . . . . . .
CDC73 . . . . . . . . . . . . . . . . . . . . . . . . .
B3GALT2 . . . . . . . . . . . . . . . . . . . . . . . . .
CFH . . . . . . . . . . . . . . . . . . . 1 . . . . .
ASPM . . . . . . . . . . . . . . . . . . . . . . . . .
ZBTB41 . . . . . . . . . . . . . . . . . . . . . . . . .
CRB1 . . . . . . . . . . . . . . . . . . . . . . . . .
DENND1B . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf53 . . . . . . . . . . . . . . . . . . . . . . . . .
NEK7 . . . . . . . . . . . . . . 1 . . . . . . . . . .
RP11-553K8.2 . . . . . . . . . . . . . . . . . . . . . . . . .
PTPRC 1 2 2 1 1 . 1 . . . . . . 1 . . . 1 1 . 1 . . . 1
MIR181A1HG . . . . . . . . . . . . . . . . . . . . . . . . .
ZNF281 . . . . . 1 . . . . . . . . . . . . . . . . . . .
DDX59 . . . . . . . . . . . . . . . . . . . . . . . . .
CAMSAP2 . . . . . . . . . . . . . . . . . . . . . . . . .
KIF21B . . . . . . . . . . . . . . . . . . . . . . . . .
TMEM9 . . . . . . . . . . . . . . . . . . . . . . . . .
PHLDA3 . . . . . . . . . . . . . . . . . . . . . . . . .
CSRP1 . . . . . . . . . . . . . . 1 1 . . . . . . . . .
RP11-134G8.7 . . . . . . . . . . . . . . . . . . . . . . . . .
RPS10P7 . . . . . . . . . . . . . . . . . . . . . . . . .
NAV1 . . . . . . . . . . . . . . . . . . . . . . . . .
IPO9-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
IPO9 . . . . . . . . . . 1 . . . . . . . . . 1 . . . .
SHISA4 . . . . . . . . . . . . . . . . . . . . . . . . .
TIMM17A . . . . . . . . . . . . 1 1 . . . . . . . . . . 1
RNPEP . . . . . . . . . . . . . . . . . . 1 . . . . . .
ARL8A . . . . . . . . . . . . . . . . 1 . . . . . . . .
PTPN7 . . 1 . . . . . . . . . . . . . . . . . 1 . . . .
LGR6 . . . . . . . . . . . . . . . . . . . . . . . . .
UBE2T . . . . . . . . . . . . . . . . . . . . . . . . .
PPP1R12B . . . . . . . . . . . . . . . . . . . . . . . . .
KDM5B . . . . . . . . . . . . . . . . . . . . . . . . .
RABIF . . . . . . . . . . . . . 1 . . . . . 1 . . . . 1
KLHL12 . . . . . . . . . . . . . . . . . . . . . . . . .
ADIPOR1 . . . 1 . . . . . . . . . . . . . 1 1 . . . . . .
CYB5R1 . . 1 . . . . . . . . . . . . . . . . . . . . . .
TMEM183A . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-134P9.3 . . . . . . . . . . . . . . . . . . . . . . . . .
LINC01136 . . . . . . . . . . . . . . . . . . . . . . . . .
BTG2 1 . . 1 . . 1 . . . . 1 3 . 1 1 . . 3 . 1 . 1 . .
ATP2B4 . . . . . . . . . . . . . . . . . . . . . . . . .
LAX1 . . . . . . . . . . . . . . . . . . . . . . . . .
ZC3H11A . . . . . . . . . . . . . . . . . . . . . . . . .
ZBED6 . . . . . . . . . . . . . . . . . . . . . . . . .
SNRPE 1 . . . 1 . 1 . . . . 1 . . . . . . . . 2 . 2 . .
SOX13 . . . . . . . . . . . . . . . . . . . . . . . . .
PPP1R15B . . . . . . . . . . . . . . . . . . . . . . . . .
PIK3C2B . . . . . . . . 1 . . . . . . . . . . . . . . . .
MDM4 . 1 . . . . . . . . . . . 1 . . . . . . 1 . 1 . .
TMEM81 . . . . . . . . . . . . . . . . . . . . . . . . .
RBBP5 . 1 . . . . . . . . . . . . . . . . . . . . . . .
DSTYK . . . . . . . . . . . . . . . . . . . . . . . . .
TMCC2 . . . . . . . . . . . . . . . . . . . . . . . . .
NUAK2 . . . . . . . . . . . . . . . . . . . . . . . . .
CDK18 . . . . . . . . . . . . . . . . . . . . . . . . .
MFSD4 . . . . . . . . . . . . . . . . . . . . . . . . .
ELK4 . . . . . . . . . . . . . . . . . . . 1 . . . . .
SLC45A3 . . . . . . . . 1 . . . . . . . . . . . . . . . .
NUCKS1 . . . 2 . . . 1 1 . . . . . . . . . . . . . . . .
RAB7L1 1 . . . . . . . . . . . . . 2 . . . . . 1 . . . .
SLC41A1 . . . . . . . . . . . . . . . . . . . . . . . . .
PM20D1 . . . . . . . . . . . . . . . . . . . . . . . . .
FAM72A . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf186 . . . . . . . . . . . . . . . . . . . . . . . . .
SRGAP2 . . . . . . . . . . . . . . . . . . . . . . . . .
IKBKE . . 1 . . . . . . . . . . . . . . . . . 1 . . . .
RASSF5 . 1 . . 1 . . . . . . . . . . . . . . 1 . . . 1 .
EIF2D . . . . . . . . . . . . . . . . 1 . . 1 . . . . .
RP11-343H5.6 . . . . . . . . . . . . . . . . . . . . . . . . .
MAPKAPK2 1 . . 1 . . . . . . . . . . . . . . . . . . . . .
IL10 . . . . . . . . . . . . . . . . . . . . . . . . .
IL24 . . . . . . . . . . . . . . . . . . . . . . . . .
FAIM3 . 2 2 . . 1 . 1 . . . . . . 1 1 . . . 2 2 . 1 1 .
YOD1 . . . . . . . . . . . . . . . . . . . . . . . . .
PFKFB2 . . . . . . . . . . . . . . . . . . . . . . . . .
C4BPB . . . . . . . . . . . . . . . . . . . . . . . . .
CD55 . 1 . . . . . . . . . 1 . . . . . . 1 . . 1 . . .
CR2 . . . . . . . . . . . . . . . . . . . . . . . . .
CR1 . . . . . . . . . . . . . . . . . . . . . . . . .
CD46 . . 1 . . . . . . . . . . . . . . . 1 . . . . . .
RP1-28O10.1 . . . . . . . . . . . . . . . . . . . . . . . . .
G0S2 . 1 . . . . . . . . . . . . . . . . . . . . . . .
HSD11B1 . . . . . . . . . . . . . . . . . . . . . . . . .
TRAF3IP3 2 1 . . 2 4 1 . . 1 . 1 1 . 1 . . . 1 1 . . . . .
DIEXF . . . . . . . . . . . . . . . . . . . . . . . . .
HHAT . . . . . . . . . . . . . . . . . . . . . . . . .
RCOR3 . . . . . . . . . . . . . . . . . . . 1 . . . . .
TRAF5 . . . . . . . . . . 1 1 . . . . . . . . 1 . . . .
SLC30A1 . . . . . . . . . . . . . . . . . . . . . . . . .
NEK2 . . . . . . . . . . . . . . . . . . . . . . . . .
LPGAT1 . . . . . . . . . . . 1 . . . . . . . . . . . . .
INTS7 . . . . . . . . . . . . . . . . . . . . . . . . .
PPP2R5A 1 . . . . 1 . . . . . . . 1 1 . . . . . 1 . . . .
TMEM206 . . . . . . . . . . . . . . . . . . . . . . . . .
NENF . . . . . . . . . . . . . . . . . . 1 . . . . . .
RP11-61J19.4 . . . . . . . . . . . . . . . . . . . . . . . . .
ATF3 . . . . . . . . 1 . . . . 1 . . . . 1 . . . . . .
RP11-338C15.3 . . . . . . . . . . . . . . . . . . . . . . . . .
BATF3 . . . . . . . . . . . . . . . . . . . . . . . . .
NSL1 . . . . . . . . . . . . . . . . . . . . . . . . .
TATDN3 . . . . . . . . . . . . . . . . . . . 1 . . . . .
FLVCR1-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
FLVCR1 . . . . . . . . . . . . . . . . . . . . . . . . .
ANGEL2 . . . . . . . . . . . . . . 1 . . . . . . . . . .
RPS6KC1 . . . . . . . . . . . . . . . . . . . . . . . . .
SMYD2 . . . . . . . . . . . . . . . . . . . . . . . . .
CENPF . . . . . . . . . . . . . . . . . . . . . . . . .
KCTD3 . . . . . . . . . . . . . . . . . . . . . . . . .
GPATCH2 . . . . . . . . . . . . . . . . . . . . . . . . .
RRP15 . . 1 . . . . . . . . . . . . . . . . . . . . . .
LYPLAL1 . . . . . . . . . . . . . . . . . . . . . . . . .
EPRS . . . . . . 1 . . . . . . 1 . . . . . . 1 . . . .
BPNT1 . . . . . . . . . . . 2 . . . . . . . . . . . . .
IARS2 . . . . . . . . . . . . . . . . . . . . . . . . .
RAB3GAP2 . . . . . . . . . . . . . . . . . . . . . . . . 1
C1orf115 . . . . . . . . . . . . . . . . . . . . . . . . .
MARC2 . . . . . . . . . . . . . . . . . . . . . . . . .
MARC1 . . . . . . . . . . . . . . . . . . . . . . . . .
HLX . . . . . . . . . . . . . . . . . . . . . . . . .
DUSP10 . . . . . . . . . 1 . . . . . . . . . . . . . . .
TAF1A . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-378J18.3 . . . . . . . . . . . . . . . . . . . . . . . . .
MIA3 . . . . . . 1 . . . 1 . . . . . . . . . . . . . .
AIDA . . . . . . . . . . 1 . . . . . . . . . . . . . .
BROX . . . . . . . . . . . . . . . . . . 1 . . . . . .
FAM177B . . . . . . . . . . 1 . . . . . . . . . . . . . .
RP11-452F19.3 . . . . . . . . . . . . . . . . . . . . . . . . .
DISP1 . . . . . . . . . . . . . . . . . . . . . . . . .
TLR5 . . . . . . . . . . . . . . . . . . . . . . . . .
SUSD4 . . . . . . . . . . . . . . . . . . . . . . . . .
CAPN8 . . . . . . . . . . . . . . . . . . . . . . . . .
CAPN2 . . . . . . . . . . . . . . 1 . . . 2 1 . . . . .
TP53BP2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-504P24.8 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-504P24.6 . . . . . . . . . . . . . . . . . . . . . . . . .
FBXO28 . . . . . . . . . . . . . . . . . . . . . . . . .
DEGS1 1 . 2 . . . . . . . . . . . . . . . . . . 1 . . .
NVL . . . . . . . . . . . . . . . . . . . . . . . . .
CNIH4 . . . . . . . . . . . . . . . . . 1 . . . . . . .
WDR26 . . . . . . . . . . . . . . . . . . . . . . . . .
CNIH3 . . . . . . . . . . . . . . . . . . . . . . . . .
LBR . . . . . . . . . . . . . . . . 1 . . . 1 . . . .
ENAH . . . . . . . . . . . . . . . . . . . . . . . . .
SRP9 1 . . . . . . . 1 . . . . . . . 1 . 2 3 . . . . .
EPHX1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-285F7.2 . . . . . . . . . . . . . . . . . . . . . . . . .
TMEM63A . . . . . . . . . . . . . . . . . . . . . . . . .
PYCR2 . 2 . . . . . . . . . . . . . . . . . . . . . 1 .
SDE2 . . . . . . . . . . . . . . . . . . . . . . . . .
H3F3A . 1 2 . . 1 . . . 1 . 1 . . . . . . . . . . 2 . 1
ACBD3 . . . . . . . . . . . . . . . . . . . . . . . . .
LIN9 . . . . . . . . . . . . . . . . . . . . . . . . .
PARP1 . 2 . 1 . . . . . . . . . . . . . . . . . . . . .
ITPKB . . 1 . . . . . . . . . . . . . . . . . . . . . .
ITPKB-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
PSEN2 . . . . . . . . . . . . . . . . . . . . . . . . .
ADCK3 . . . . . . . . 1 . . . . . . . . . 1 . . . . . .
ZNF678 . . . . . . . . . . . . . . . . . . . . . . . . .
SNAP47 . . . . . . . . 1 . . . . . . . . . 1 . . . . . .
JMJD4 . . . . . . . . . . . . . . . . . . . . 1 . . . .
ARF1 . . . 2 . 2 . . . . . 1 . 2 1 . . 1 . . . . . . .
C1orf35 . 1 . . . . . . . . . . . . . . . . . . . . . . .
MRPL55 . . 2 1 . . . . . . . . . . 1 . 1 . 1 . . 1 . . 1
GUK1 . . 1 . . 16 . 1 . 1 . . 1 1 . . 1 1 1 4 1 1 2 . 1
IBA57 . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf145 . . . . . . . . . . . . . . . . . . . . . . . . .
OBSCN . . . . . . . . . . . . . . . . . . . 1 . . . . .
TRIM11 . . . . . . . . . . . . . . . . . . . . . . . . .
HIST3H2A . . . . . . . . . . . . . . . . . . . . . . . . .
RNF187 . . . . . . . 7 . . . . . . . . . . . . 1 . . . .
RHOU . . . . . . . . . . . . . . . . . . . . . . . . .
RAB4A . . . . . . . . . . . . . 1 . . . . . 1 2 1 . . .
RP4-803J11.2 . . . . . . . . . . . . . . . . . . . . . . . . .
CCSAP . . . . . . . . . . . . . . . . . . . . . . . . .
NUP133 . . . . . . . . . . . . . . . . . . . . . . . . .
ABCB10 . . . . . . . . . . . . . . . . . . . . . . . . .
TAF5L . . . . . . . . . . . . . . . . . . . . . . . . .
URB2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-552D4.1 . . . . . . . . . . . . . . . . . . . . . . . . .
GALNT2 . . . . . . . . . . . . . . . . 1 . . . . . . . .
COG2 . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf198 . . . . . . . . . . . . . . . . . . . . . . . . .
TTC13 . . . . . . . . . . . . . . . . . . . . . . . . .
ARV1 . . . 1 . . . . . . . . . . . . . . . . . . 1 . .
FAM89A . . . . . . . . . . . . . . . . . . . . . . . . .
C1orf131 . . . . . . . . . . . . . . . . . . . . 1 . . . .
GNPAT . . . 1 . . . . . . . . . . . . . . . 1 . . . . .
EXOC8 . . . . . . . . . . . . . . . . . . . . . . . . .
SPRTN . . . . . . . . . . . . . . . . . . . . . . . . .
EGLN1 1 . . . . . . . . . . . 1 . . . . . 1 . 1 . . . .
RP11-295G20.2 . . . . . . . . . . . . . . . . . . . . . . . . 1
TSNAX . . . . . . . . . . 1 . . . . . . . . 1 . . . . 1
DISC1 . . . . . . . . . . . . . . . . . . 1 . . . . . .
SIPA1L2 . . . . . . . . . . . . . . . . . . . . . . . . .
MAP10 . . . . . . . . . . . . . . . . . . . . . . . . .
NTPCR . . . . . . . 1 . . . . . . . . . . . . . . . . .
PCNXL2 . . . . . . . . . . . . . . . . . . . . . . . . .
SLC35F3 . . . . . . . . . . . . . . . . . . . . . . . . .
RP5-827C21.4 . . . . . . . . . . . . . . . . . . . . . . . . .
COA6 . . . . . . . . . . . . . 1 . . . . . . 1 . . . .
TARBP1 . . . . . . . . . . 1 . . . . . . . . . . . . . .
IRF2BP2 1 . . . . . 1 . . . 1 . . . . . . 1 . . . . . . .
RP4-781K5.2 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-443B7.1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-443B7.3 . . . . . . . . . . . . . . . . . . . . . . . . .
TOMM20 . . . . . . . 1 . . 1 . 1 1 . . . . 6 1 . . 1 . .
RBM34 . . . . . . . . . . . . . . 1 . . . . . . . . . .
ARID4B . . . . . . . 1 . . . . . . . . . . . . 1 . . . .
GGPS1 . . . . . . . . . . . . . 1 . . . . . . 1 . . . .
TBCE . . . . . . . . . . . . . . . . . . . . . 1 . . .
B3GALNT2 . . . . . . . . . . . . . . . . . . . . . . . . .
LYST . . . . . . . 1 . . . . . . . . . . 2 . . . . . 1
NID1 . . . . . . . . . . . . . . . . . . . . . . . . .
GPR137B . . . . . . . . . . . . . . . . . . . . . . . . .
ERO1LB . . . . . . . 1 . . . . . . . . . . . . . . . . .
EDARADD . . . . . . . . . . . . . . . . . . . . . . . . .
LGALS8 . . . . . . . . . . . . . 1 . . . . . . . . . . 1
LGALS8-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-385F5.5 . . . . . . . . . . . . . . . . . . . . . . . . .
HEATR1 . . . . . . . . . . . . . . . . . . . . . . . . .
MTR . . . . . . . . . . . . . . . . . . . . . . . . .
GREM2 . . . . . . . . . . . . . . . . . . . . . . . . .
FH . . . . . . . . . . . . . 1 . . . . . . . . . . .
KMO . . . . . . . . . . . . . . . . . . . . . . . . .
OPN3 . . . . . . . . . . . . . . . . . . 2 . . . . . .
CHML . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-261C10.3 . . . . . . . . . . . . . . . . . . . . . . . . .
CEP170 . . . . . . . . . . . . . . . . . . . . . . . . .
SDCCAG8 . . . 1 . . . . . . . . . . . . . . . . . . . . .
AKT3 . . . . . . . . . . . . . . . . . . . . . . . . .
ZBTB18 . . . . . . . . . . . . . 1 . . . . . . . . . . .
ADSS . . . 1 . . . 1 . . . . 1 . . . . . . . 1 . . . .
C1orf101 . . . . . . . . . . . . . . . . . . . . . . . . .
DESI2 . . . . . . 1 . . . . . . . . . . 1 . . . . . . .
COX20 . . 1 . . . . . . . . . . . . . 1 . . . . . 1 . .
HNRNPU-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
HNRNPU 1 1 1 . . 1 . . . . 1 . . . . . . . . . . . . . .
RP11-11N7.4 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-156E8.1 1 . . . . . . . . . . . . . . . . . . . . . . . .
EFCAB2 . . . . . . . . . . . . . . . . . . 2 . . . . . 2
SMYD3 . . . . . . . . . . . . . . . . . . . . . . . . .
TFB2M . . . . . . . . . . . . . . . . . . . . . . . . .
CNST . 1 . . . . . . . . . . . . . . . . . . . . . . .
SCCPDH . 1 . . . . . . . . 1 . . . . . . . . . . . . . .
AHCTF1 . . . . . . . . . . . . . . . . . . . . . . . . .
ZNF670 . . . . . . . . . . . . . . . . . . . . 1 . . . .
ZNF669 . . . . . . . . . . . . . 1 . . . . . . . . . . .
ZNF124 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-488L18.10 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-488L18.8 . . . . . . . . . . . . . . . . . . . . . . . . .
ZNF496 . . . . . . . . . . . . . . . . . . . . . . . . .
NLRP3 . . . . . . . . . . . . . . . . . . . . . . . . .
TRIM58 . . . . . . . . . . . . . . . . . . . . . . . . .
SH3BP5L . . . . . . . . . . . . . . . . . . . . . . . . .
ZNF672 . . . . . . . . . . . . . . . . . . . . . . . . .
ZNF692 . 1 . . . . . . . . 1 . . . . . . . . . . . . . .
PGBD2 . . . . . . . . . . . . . . . . . . . . . . . . .
SH3YL1 . . . . . . . . . . . . . . . . . 1 . . . . 1 . .
ACP1 . . 1 . . 1 . . . . 1 . . . . . . . 1 . 6 . . . 1
TMEM18 . . . 1 . . . . . . . . . . . . . . 1 . . . . . .
AC116614.1 . . . . . . . . . . . . . . . . . . . . . . . . .
TPO . . . . . . . . . . . . . . . . . . . . . . . . .
TSSC1 . . 1 . . . . . . . 1 . . . . . . . 2 . . . . . .
TRAPPC12 . . . 1 . . . . . . . . . . . . . . . . 1 . . . .
TRAPPC12-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
ADI1 . . . . . . . . . 1 1 . . . . . . . . . . . . 2 1
AC142528.1 . . . . . . . . . . . . . . . . . . . . . . . . .
AC108488.4 . . . . . . . . . . . . . . . . . . . . . . . . .
RNASEH1 . . . . . . . . . . . . . . . . . . . . . . . . .
RNASEH1-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
RPS7 13 31 8 10 1 8 5 7 6 4 15 12 11 10 3 2 2 3 12 15 24 7 18 4 3
CMPK2 . . . 1 . . . . . . . . . . . . . . 3 . . . . . .
RSAD2 . . . . . . . . . . . 1 . . . . . . . . . . . . .
RNF144A . . . . . . . . . . . . . . . . . . . . . . . . .
AC092580.4 . . . . . . 2 . 1 . . . . . . . . . . . . . . . .
LINC00299 . . . . . . . . . . . . . . . . . . . . . . . . .
AC011747.4 . . . . . . . . . . . . . . . . . . . . . . . . .
ID2 1 . . 3 2 . . . . . . 1 1 4 . 1 1 . 2 . 1 . . . 2
KIDINS220 . . . . . . . . . . . . . . . . . . . . . . . . .
MBOAT2 . . . . . . . . . . . . . . . . . . . . . . . . .
ASAP2 . . . . . . . . . . . . . . . . . . . . . . . . .
ITGB1BP1 . . 1 . . . . . 1 . . . . . . . . . . 1 . . . . 1
CPSF3 . . . . . . . . . . . . . . . . . . . . . . . . .
IAH1 1 . . 2 . . 5 . 1 . . . . . 1 . . 1 1 . 2 . . . .
ADAM17 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-214N9.1 . . . . . . . . . . . . . . . . . . . . . . . . .
YWHAQ . 2 2 . 1 2 2 1 . 2 . . 1 1 . . . . . . 3 . . . .
TAF1B . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-95D17.1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-254F7.2 . . . . . . . . . . . . . . . . . . . . . . . . .
KLF11 . . . . . . . . . . . . . . . . . . . . . . . . .
RRM2 . . . . . . . . . . . . . . . . . . . . . . . . .
HPCAL1 . 1 . . . . . . . . . . . . . . . . . . . . . . .
ODC1 . 2 . . . . . . . . . . . 2 . . . . . . . . . . 1
NOL10 . . . . . . . . . . . . . . . . . . . . 1 . . . .
RN7SL832P . . . . . . . . . . . . . . . . . . . . . . . . .
ATP6V1C2 . . . . . . . . . . . . . . 1 . . . . . . . . . .
RP11-791G15.2 . . . . . . . . . . . . . . . . . . . . . . . . .
PDIA6 . . . . . . . . . 1 . . . . . 1 1 1 . 1 . . . . 1
PQLC3 . . . 1 . 1 . . . . . . . . . . . . . . . . . . .
ROCK2 . . . . . . . . . . . . . . . . . . . . . . . . .
E2F6 . . . . . . . . . . . . . . . . . . . . . . . . .
LPIN1 . 2 . . . . . . . . . . . . . . 1 . . . . . . . .
TRIB2 . . . . . . . . . . 1 . . . . . . . . . . . . . .
NBAS . . . . . . . . . . . . . . . . 1 . . . . . . . .
DDX1 . . . . . . . . . . . . . . . . . . . . . . . . .
FAM49A . . . . . . . . . . . . . . . . . . . . . . . . .
SMC6 . . . . . . . . . . . . . . . . . . . . . . . . .
GEN1 . . . . . . . . . . . . . . . . . . . . . . . . .
RP11-111J6.2 . . . . . . . . . . . . . . . . . . . . . . . . .
RDH14 . . . . . . . . . . . . . . . . . . . . 1 . . . .
LINC00954 . . . . . . . . . . . 1 . . . . . . . . . . . . .
TTC32 . . . . . . . . . . . . . . . . . . . . . . . . .
WDR35 . . . . . . . . . . . . . . . . . . . . . . . . .
LAPTM4A . . . . . . . 1 . . 1 . . 1 . . . 1 1 . . 1 1 . .
PUM2 . . . 1 . 1 . . . . . . . . . . . . . . . . . . .
RHOB . . . 4 . . . . . 1 . . . . . . . 2 1 . . . 3 . .
HS1BP3 . . . . . . . . . . . . . . . . . . . . . . . . .
C2orf43 . . . . . . . . . . . . . . . . . . . . . . . . .
KLHL29 . . . . . . . . . . . . . . . . . . . . . . . . .
ATAD2B . . . . . . . . . . . . . . . . . . . . 1 . . . .
UBXN2A . . . . . . . . . . . 1 . . . . . . . . . . . . .
MFSD2B . . . . . . . . . . . . . . . . . . . . . . . . .
C2orf44 . . . . . . . . . . . . . . . . . . . . . . . . .
FKBP1B . . . . . . . . . . . . . . . . . . . . . . . . .
SF3B14 . . . 1 . . . . . . . . 1 . . . . . . 2 . . . . .
FAM228B . . . . . . . . . . . 1 . . . . . . . . . . . . .
TP53I3 . . . . . . . . . . . . . . . . . . . . . . . . .
FAM228A . . . . . . . . . . . . . . . . . . . . . . . . .
ITSN2 . . 1 . . . . . . . 1 . . . . . . . 2 . 1 . . . .
NCOA1 . . . . . . . . . . . . . . . . . . . . . . . . .
PTRHD1 . 1 . . . 1 . . . . . . . . . . . . . . . . . . .
CENPO . . . . . . . . . . . . . . . . . . . . . . . . .
ADCY3 . . . . . . . . . . . . . . . . . . . . 1 . . . .
DNAJC27 . . . . . . . . . . . . . . . . . . . . . . . . .
DNAJC27-AS1 . . . . . . . . . . . . . . . . . . . . . . . . .
POMC 1 . . . . . . . . . . . . . . . . . . . . . . . 1
DNMT3A 1 . . . . . . . . . . . . . . . . . . . . . . . .
AC012074.2 . . . . . . . . . . . . . . . . . . . . . . . . .
DTNB . . . . . . . . . . . . . . . . . . . . . . . . .
AC104699.1 . . . . . . . . . . . . . . . . . . . . . . . . .
ASXL2 . . . . . . . . . . . . . . . . . . . . . . . . .
KIF3C . . . . . . . . . . . . . . . . . . . . . . . . .
AC013449.1 . . . . . . . . . . . . . . . . . . . . . . . . .
RAB10 . . . . . . . . . . . . . . . . . . 1 . . . . . 1
GAREML . . . . . . . . . . . . . . . . . . . . . . . . .
HADHA . 1 . . . . . . . . . . . . . . . . 1 2 . . 2 . .
HADHB . . . 1 . 1 . . . . . . . . . . 1 . . 1 . . . . .
EPT1 . . . . . . . . . . . . . . . . . . . . . . . . .
OTOF . . . . . . . . . . . . . . . . . . . . . . . . .
SLC35F6 . . . . . . . . . 1 . . . . . . . . . . . . . . .
CENPA . . . . . . . . . . . . . . . . . . . . . . . . .
MAPRE3 . . . . . . . . . . . . . . . . . . . . . . . . .
TMEM214 . . . . . . . . . . . . . . . . . . . 1 . . . . .
AGBL5 . . . . . . . . . . . . . . . . . . . . . . . . 1
OST4 . . 2 3 . . . . . 2 . . 2 2 2 . . 1 1 . 1 1 2 1 2
KHK . . . . . . . . . . . . . . . . . . . . . . . . .
ABHD1 . . . . . . . . . . . . . . . . . . . . . . 1 . .
PREB . . . . . 2 . . . . . . . . . 1 . . . . . . . . 1
SLC5A6 . . . . . . . . . . . . . . . . . . . . 1 . . . .
ATRAID . 1 . . . . 1 1 . . . 1 1 1 . . . . . . . . . . .
CAD . . . . . . . . . . . . . . . . . . . . . . . . .
TRIM54 . . . . . . . . . . . . . . . . . . . . . . . . .
MPV17 . . 1 . . . . . . . . . . . . . . . . . . . . . .
GTF3C2 . . . . . . . . . . . . . . . . . . . . . . . . .
AC074117.10 . . . . . . . . . . . . . . . . . . . . . . . . .
EIF2B4 . . . . . . . . . . . . . . . . . . . . . . . . .
SNX17 . . . 1 . . . . . . 1 . . . . . . . . 1 1 . . . .
ZNF513 . . . . . . . . . . . . . . . . . . . . . . . . .
PPM1G . 1 . 1 . . . 1 . . 1 . . 1 . . . . . . 1 . . 1 .
NRBP1 . 1 . . . . . . . . . . . . . . . 1 1 . 1 . 1 . .
KRTCAP3 . . . . . . . . . . . . . . . . . . . . . . . . .
IFT172 . . . . . . . . . . . . . . . . . . . . 1 . . . .
ZNF512 . . . . . 1 . . . . . . . . . . . . . . . . . . .
CCDC121 . . . . . . . . . . . . . . . . . . . . . . . . .
GPN1 . . . . . . . . . . . . . . . . . . . . . . . 1 .
SUPT7L . . . . . . . . . . . . . . . . . . . . . . . . .
SLC4A1AP . . . . . . . . . . . . . . . . . . . . . . . . .
MRPL33 . . . . . . . 1 . . . . . . . . . . 1 . . . . . .
RBKS . . . . . . . . . . . . . . . . . . . . . . . . .
BRE . . . . . . 1 . . . . . . . . . . . . . 1 . . . .
FOSL2 . . . . . . . . . . . . . . . . . . . . . . . . .
PLB1 . . . . . . . . . . . . . . . . . . . . . . . . .
PPP1CB . . . . . . . 1 . . . . . . . . . . . . . . . . .
AL627309.1 . . . . . . . ......
AP006222.2 . . . . . . . ......
RP11-206L10.2 . . . . . . . ......
RP11-206L10.9 . . . . . . . ......
LINC00115 . . . . . . . ......
NOC2L . . . . . . . ......
KLHL17 . . . . . . . ......
PLEKHN1 . . . . . . . ......
RP11-54O7.17 . . . 1 . . . ......
HES4 . . . . . . 2 ......
RP11-54O7.11 . . . . . . . ......
ISG15 1 . . 2 . 12 4 ......
AGRN . . . . . . . ......
C1orf159 . . . . . . . ......
TNFRSF18 . . . . . . . ......
TNFRSF4 . . . . . . . ......
SDF4 1 . . . . . 2 ......
B3GALT6 . . . . . . . ......
FAM132A . . . . . . . ......
UBE2J2 . . . 1 . . . ......
ACAP3 . . . . . . . ......
PUSL1 . . . . . . . ......
CPSF3L . . . . . . . ......
GLTPD1 . . . . . . . ......
DVL1 . . . . . . . ......
MXRA8 . . . . . . . ......
AURKAIP1 . . . 1 . . . ......
CCNL2 . . . . . . . ......
RP4-758J18.2 . . . . . . . ......
MRPL20 . . . . . . . ......
ATAD3C . . . . . . . ......
ATAD3B . . . . . . . ......
ATAD3A . . . . 1 . . ......
SSU72 . 1 . . . . 1 ......
AL645728.1 . . . . . . . ......
C1orf233 . . . 1 . . . ......
RP11-345P4.9 . . . . . . . ......
MIB2 . . . . . . . ......
MMP23B . . . . . . . ......
CDK11B . . . . . . . ......
SLC35E2B . . . . . . . ......
CDK11A . . . 1 . . . ......
SLC35E2 . . . . . . . ......
NADK . . . . . . 2 ......
GNB1 1 . . . . . . ......
RP1-140A9.1 . . . . . . . ......
TMEM52 . . . . . . . ......
PRKCZ . . . . . . . ......
RP5-892K4.1 . . . . . . . ......
C1orf86 . . . 3 . . . ......
AL590822.2 . . . . . . . ......
SKI . . . . . . . ......
RER1 . . . . 1 . 1 ......
PEX10 . . . . . . . ......
PLCH2 . . . . . . . ......
PANK4 . . . . . . . ......
RP3-395M20.12 . . . . . . . ......
TNFRSF14 1 . . 1 . . 1 ......
RP3-395M20.9 . . . . . . . ......
FAM213B . . . . . . . ......
MMEL1 . . . . . . . ......
TTC34 . . . . . . . ......
MEGF6 . . . . . . . ......
TPRG1L . . . . . . . ......
WRAP73 . . . . . . . ......
TP73-AS1 . . . . . . . ......
SMIM1 . . . . . . . ......
LRRC47 . . . . . . 1 ......
CEP104 . . . . . . . ......
DFFB . . . . . . . ......
C1orf174 . 1 . . . . . ......
NPHP4 . . . . . . . ......
KCNAB2 . 2 . . . . . ......
RPL22 2 1 4 2 7 3 2 ......
RNF207 . . . . . . . ......
ICMT . . . . . . 1 ......
GPR153 . . . . . . . ......
ACOT7 . . . . . . . ......
RP1-202O8.3 . . . . . . . ......
ESPN . . . . . . . ......
TNFRSF25 . . . . . 3 . ......
PLEKHG5 . . . . . . . ......
NOL9 . . . . . . . ......
ZBTB48 . . . . . . . ......
KLHL21 . . . . . . . ......
PHF13 . . . . . . . ......
THAP3 . . . . . . . ......
DNAJC11 . . . . . . . ......
RP11-312B8.1 . . . . . . . ......
CAMTA1 . 13 . . 2 . . ......
VAMP3 . . . . . . . ......
PER3 . . . . . . . ......
UTS2 . . . . . . . ......
TNFRSF9 . . . . . . . ......
PARK7 . . . 3 1 1 3 ......
SLC45A1 . . . . . . . ......
RERE . . . . . . . ......
RP5-1115A15.1 . . . . . . . ......
ENO1 1 . . 2 1 1 6 ......
ENO1-AS1 . . . . . . . ......
CA6 . . . . . . . ......
SLC2A5 . . . . . . . ......
GPR157 . . . . . . . ......
H6PD . . . . . . . ......
SPSB1 . . . . . . . ......
SLC25A33 . . . . . . . ......
TMEM201 . . . . . . . ......
PIK3CD . . . . . . . ......
C1orf200 . . . . . . . ......
RP11-558F24.4 . . . . . . . ......
CLSTN1 . . . . . . . ......
CTNNBIP1 . . . 1 . . . ......
LZIC . . . . 1 . . ......
NMNAT1 . . . . . . . ......
RBP7 . . . . . . . ......
UBE4B . . . . . . . ......
KIF1B . . . . . . . ......
PGD . . . 1 . . . ......
APITD1 . . . . . . . ......
DFFA . . . . . . . ......
PEX14 . . . . . . . ......
CASZ1 . . . . . . . ......
TARDBP . . . . . 1 . ......
RP4-635E18.8 . . . . . . . ......
SRM . . . 1 1 . . ......
EXOSC10 . . . . . . . ......
MTOR . . . . . . . ......
UBIAD1 . . . . . . . ......
FBXO2 . . . . . . . ......
FBXO44 . . . . . . . ......
FBXO6 . . . . . . 1 ......
MAD2L2 . . . . . . . ......
DRAXIN . . . . . . . ......
AGTRAP 1 . . 1 . . 1 ......
MTHFR . . . 2 . . . ......
CLCN6 . . . . . . . ......
NPPA-AS1 . . . . . . . ......
KIAA2013 . . . . . . . ......
PLOD1 . . . . . . . ......
MFN2 . . . . . . . ......
MIIP . . . . 1 . . ......
TNFRSF8 . . . . . . 1 ......
TNFRSF1B . . . 1 . . . ......
VPS13D . . . . . . . ......
DHRS3 . . . . . . . ......
PRDM2 . . . 1 . . . ......
TMEM51 . . . . . . . ......
RP3-467K16.4 . . . . . . . ......
EFHD2 . . . 1 . . 1 ......
CASP9 . . . . . . . ......
DNAJC16 . . . . . . . ......
AGMAT . . . . . . . ......
DDI2 . . . . . . . ......
PLEKHM2 . . . . . . . ......
FBLIM1 . . . . . . . ......
RP11-169K16.9 . . . . . . . ......
SPEN . . 1 . . . . ......
ZBTB17 . . . . . . . ......
ARHGEF19 . . . . . . . ......
RSG1 . . . . . . . ......
FBXO42 . . . . . . . ......
SZRD1 1 . 1 . . . . ......
SPATA21 . . . . . . . ......
NECAP2 . . 1 1 . . 1 ......
RP4-798A10.2 . . . . . . . ......
RP4-798A10.7 . . . . . . . ......
RP4-798A10.4 . . . . . . . ......
NBPF1 . . 1 . . . . ......
CROCCP2 . . . . . . . ......
CROCC . . . . . . . ......
RP11-108M9.4 . . . . . . . ......
RP11-108M9.6 . . . . . . . ......
ATP13A2 . . . . . . . ......
SDHB . . . 1 . . . ......
PADI2 . . . . . . . ......
PADI4 . . . . . . . ......
RCC2 . . . . . . . ......
ARHGEF10L . . . . . . . ......
ALDH4A1 . . . . . . . ......
IFFO2 . . . . . . . ......
UBR4 . . . 1 . . . ......
RP1-43E13.2 . . . . . . . ......
EMC1 . . . . . . . ......
MRTO4 . . . . . . . ......
AKR7A2 . . . . . . . ......
PQLC2 . . . . . . . ......
CAPZB 2 . . 3 . 2 5 ......
MINOS1-NBL1 . . . . . . . ......
MINOS1 . . 1 2 . . . ......
NBL1 . . . . . . . ......
TMCO4 . . . . . . . ......
OTUD3 . . . . . . . ......
UBXN10-AS1 . . . . . . . ......
UBXN10 . . . . . . . ......
CAMK2N1 . . . . . . . ......
MUL1 . . . . . . . ......
CDA . . . 1 1 . 1 ......
PINK1 . . . . . . . ......
PINK1-AS . . . . . . . ......
DDOST . . 2 1 . . . ......
HP1BP3 . . . . . . 1 ......
RP5-930J4.4 . . . . . . . ......
EIF4G3 . . . . . . . ......
ECE1 . . . . . . . ......
RP3-329E20.2 . . . . . . . ......
NBPF3 . . . . . . . ......
ALPL . . . . . . . ......
USP48 . . . . . . . ......
HSPG2 . . . . . . . ......
RP1-224A6.3 . . . . . . . ......
LINC00339 . . . . . . . ......
CDC42 . . . 2 . . 1 ......
ZBTB40 . . . . . . . ......
C1QA . . . 3 . . . ......
C1QC . . . . . . . ......
C1QB . . . 3 . . . ......
EPHB2 . . . . . . . ......
KDM1A . . . 2 . . . ......
LUZP1 . . . . . . . ......
RP5-1057J7.6 . . . . . . . ......
HNRNPR . . . . . . . ......
ZNF436 . . . . . . . ......
C1orf213 . . . . . . . ......
TCEA3 . . . . . . . ......
E2F2 . . . . . . . ......
ID3 . . 1 . . . . ......
MDS2 . . . . . . . ......
RPL11 13 37 32 44 36 22 12 ......
TCEB3 . . . . . . . ......
RP5-886K2.3 . . . . . . . ......
PITHD1 . . . . 1 . . ......
LYPLA2 . . . . 2 . . ......
GALE . . . . . . . ......
HMGCL . . . . 1 . 1 ......
FUCA1 . . . . . . . ......
CNR2 . . . . . . . ......
PNRC2 . . . . . . . ......
SRSF10 . . . 1 . . . ......
IFNLR1 . . . . . . . ......
STPG1 . . . . . . . ......
NIPAL3 . . . . . . 1 ......
RCAN3 . . . 1 . . . ......
RP4-594I10.3 . . . . . . . ......
SRRM1 . . . . . 1 . ......
CLIC4 . . . . . . . ......
RUNX3 . . 1 . . . . ......
SYF2 1 . . 2 1 . 3 ......
C1orf63 . . 2 1 . . 2 ......
RP3-465N24.6 . . . . . . . ......
TMEM50A . . . . . 1 1 ......
TMEM57 . . . . . . . ......
LDLRAP1 1 . 2 1 3 . . ......
RP11-70P17.1 . . . . . . . ......
MAN1C1 . . . . . . . ......
RP1-187B23.1 . . . . . . . ......
SEPN1 . . . . . . . ......
RP1-317E23.3 . . . . . . . ......
MTFR1L . 1 . . 1 . . ......
AL020996.1 . . . . . . . ......
PAQR7 . . . . . . . ......
STMN1 . . . . . . . ......
PAFAH2 . . . . . . . ......
PDIK1L . . . . 1 . . ......
ZNF593 . . 1 . . . . ......
CNKSR1 . . . . . . . ......
CEP85 . . . . . . . ......
SH3BGRL3 1 1 1 9 2 1 11 ......
UBXN11 . . . . . . . ......
CD52 11 1 2 9 3 1 5 ......
AIM1L . . . . . . . ......
ZNF683 . . . . . . . ......
DHDDS . . . . . . . ......
HMGN2 . . . . . . . ......
RPS6KA1 . . . . . 1 . ......
ARID1A . . . . . . . ......
PIGV . . . . . . . ......
ZDHHC18 . . . 1 . . . ......
GPN2 . . . . . . . ......
GPATCH3 . . . . . . . ......
NUDC . . . 1 . . . ......
C1orf172 . . . . . . . ......
TRNP1 . . . . . . . ......
SLC9A1 . . . . . . . ......
WDTC1 1 . . . . . . ......
TMEM222 . . . . . . . ......
SYTL1 . . . . . . . ......
MAP3K6 . . . . . . 1 ......
WASF2 . . . 1 . . 2 ......
RP1-159A19.4 . . . . . . . ......
AHDC1 . . . . . . . ......
FGR . . . . . . 1 ......
IFI6 . . . . . 10 3 ......
FAM76A . . . . . . . ......
STX12 . 1 . . . . . ......
PPP1R8 . . . . . . . ......
THEMIS2 . . . . . . 1 ......
RPA2 . . . . . . . ......
XKR8 . . . . . . . ......
EYA3 . . . . . . . ......
PTAFR . . . 1 . . . ......
DNAJC8 . . . . . . . ......
AL353354.2 . . . . . . . ......
ATPIF1 . . 2 1 . . . ......
RP5-1092A3.4 1 . . . . . . ......
SESN2 . . . . . . . ......
MED18 . . . . . . . ......
PHACTR4 . . . . . . . ......
RCC1 . . . . . . . ......
TRNAU1AP . . . . . . . ......
SNHG12 . . . . . . . ......
TAF12 . . . . . . . ......
RP11-442N24--B.1 . . . . . . . ......
RNU11 . . . . . . . ......
GMEB1 . . . . . . . ......
YTHDF2 . . . . 1 . . ......
EPB41 . . . . . . . ......
TMEM200B . . . . . . . ......
SRSF4 . . . . . . . ......
MECR . . . 1 . . . ......
MATN1 . . . . . . . ......
MATN1-AS1 . . . . . . . ......
LAPTM5 3 2 1 3 2 . 5 ......
PUM1 . . . . . . . ......
SNRNP40 . . . . . . 1 ......
ZCCHC17 . . . 1 . . . ......
SERINC2 . . . . . . . ......
PEF1 . . . . . . . ......
SPOCD1 . . . . . . . ......
PTP4A2 . 1 . 1 1 . 1 ......
KHDRBS1 . . . . 1 . . ......
TMEM39B . 1 . . . . . ......
KPNA6 . . . . . . . ......
TXLNA . . . . . . . ......
CCDC28B . . . . . . . ......
TMEM234 . . . . . . . ......
EIF3I . . . . 1 . . ......
FAM167B . . . . . . . ......
LCK . . 1 . 2 . . ......
HDAC1 . . . . . . . ......
MARCKSL1 . . . 1 . . . ......
FAM229A . . . . . . . ......
BSDC1 . . . . . . 1 ......
ZBTB8A . . . . . . . ......
ZBTB8OS . . . . . . . ......
RBBP4 . . . . . . . ......
SYNC . . . . . . . ......
YARS . . . . . . . ......
S100PBP . . . . 1 . . ......
RNF19B . . . . . . . ......
AK2 . 1 . 1 . . . ......
ADC . . . . . . . ......
TRIM62 . . . . . . . ......
ZNF362 . . . . . . . ......
PHC2 . . . . . . . ......
SMIM12 . . . 1 . . . ......
DLGAP3 . . . . . . . ......
RP11-244H3.4 . . . . . . . ......
ZMYM6NB . . . . . . . ......
ZMYM6 . . . . . . . ......
ZMYM1 . . . . . . . ......
SFPQ . . . . . . . ......
ZMYM4 . . . 1 . . . ......
KIAA0319L . . . . . 1 . ......
NCDN . . . . . . . ......
RP4-728D4.2 . . . . . . . ......
TFAP2E . . . . . . . ......
PSMB2 . . . 3 1 . . ......
C1orf216 . . . . . . . ......
CLSPN . . . . . . . ......
AGO4 . . . . . . . ......
AGO1 . . . . . . . ......
RP4-789D17.5 . . . . . . . ......
AGO3 . . . . . . . ......
ADPRHL2 . . . . . . . ......
TRAPPC3 . . . . 1 . . ......
MAP7D1 . . . . . . . ......
THRAP3 . . . 1 . . . ......
SH3D21 . . . . . . . ......
EVA1B . . . . . . . ......
STK40 . . . . . . . ......
LSM10 . 1 . . . . 1 ......
OSCP1 . . . . . . . ......
MRPS15 . . 1 . . . . ......
CSF3R . . . 1 . . . ......
LINC01137 . . . . . . . ......
ZC3H12A . . . . . . . ......
MEAF6 . 1 . . . . . ......
SNIP1 . . . . . . . ......
GNL2 . . . . . . 1 ......
C1orf109 . . . . . . . ......
CDCA8 . . . . . . . ......
YRDC . . . 1 . . . ......
C1orf122 1 . . . . . 1 ......
MTF1 . . . . . . . ......
INPP5B . . . . . . . ......
SF3A3 . . . . . . 1 ......
FHL3 . . . . . . . ......
UTP11L . . . . . . . ......
RRAGC . . 1 . 1 . . ......
RP5-864K19.4 . . . . . . . ......
MYCBP . . . . . . . ......
AKIRIN1 1 . . 1 . . 1 ......
NDUFS5 . . 1 . 2 . . ......
MACF1 . . . . 13 . . ......
KIAA0754 . . . . . . . ......
RP11-69E11.4 . . . . . . . ......
PABPC4 . . . . . . . ......
HPCAL4 . . . . . . . ......
PPIE . . . . . . . ......
TRIT1 . . . . . . . ......
MYCL . . . . . . . ......
MFSD2A . . . . . . . ......
CAP1 . . . . . . 5 ......
PPT1 . . 2 . . . 1 ......
RLF . . . . . . . ......
RP1-39G22.7 . . . . . . . ......
ZMPSTE24 . . . 1 . . . ......
COL9A2 . . . . . . . ......
SMAP2 . . . . . . 2 ......
ZFP69B . . . . . . . ......
ZFP69 . . . 1 . . . ......
RP11-656D10.3 . . . . . . . ......
EXO5 . . . . . . . ......
ZNF684 . . . . . . . ......
RIMS3 . . . . . . . ......
NFYC-AS1 . . . . . . . ......
NFYC . . . . . . . ......
CITED4 . . . . 1 . . ......
CTPS1 . . . . . . . ......
SLFNL1 . . . . . . . ......
SCMH1 . . . . . . . ......
HIVEP3 . . . . . . . ......
FOXJ3 . . . . . . . ......
PPCS 1 . . 1 1 . . ......
CCDC30 . . . . . . . ......
PPIH . . . . 1 . . ......
YBX1 1 . 1 13 3 2 10 ......
LEPRE1 . . . . . . . ......
C1orf50 . . . . . . . ......
CCDC23 . . . . 1 . 1 ......
ERMAP . . . . . . . ......
RP11-342M1.3 . . . . . . . ......
ZNF691 . . . . . . . ......
SLC2A1 . . . . . . . ......
SLC2A1-AS1 . . . . . . . ......
EBNA1BP2 . . 1 . . 1 . ......
WDR65 . . . . . . . ......
RP1-92O14.3 . . . . . . . ......
CDC20 . . . . . . . ......
ELOVL1 . . . . . . . ......
MED8 . . . 1 1 . . ......
SZT2 . . . . . . . ......
HYI . . . . . . . ......
KDM4A . . . . . . . ......
KDM4A-AS1 . . . . . . . ......
ST3GAL3 . . . . . . . ......
IPO13 . . . . . . . ......
DPH2 . . . . . . . ......
ATP6V0B . . . 3 . . 2 ......
CCDC24 . . . . . . . ......
DMAP1 . . . . . . . ......
ERI3 . . . . . . . ......
RNF220 . . . . 1 . . ......
TMEM53 . . . . . . . ......
C1orf228 . . . 1 . . . ......
KIF2C . . . . . . . ......
RPS8 18 16 25 32 14 9 4 ......
BEST4 . . . . . . . ......
PLK3 . . . 1 . . . ......
BTBD19 . . . . . . . ......
PTCH2 . . . . . . . ......
EIF2B3 . . . 1 . . 1 ......
HECTD3 . . . . . . . ......
UROD . . . . . . . ......
HPDL . . . . . . . ......
MUTYH . . . . . . . ......
TOE1 . . . . . . . ......
TESK2 . . . . . . . ......
CCDC163P . . . . . . . ......
PRDX1 2 . . 2 . . . ......
AKR1A1 . . . . . . . ......
NASP . . . . 1 . . ......
GPBP1L1 . . . 1 . 1 . ......
TMEM69 . . . . . . . ......
IPP . . . . . . . ......
MAST2 . . . . . . . ......
PIK3R3 . . . . . . . ......
POMGNT1 . . . . . . . ......
LRRC41 1 . . . . . . ......
UQCRH . . . 1 . . 1 ......
NSUN4 . . . . . . . ......
FAAH . . . . 20 . . ......
MKNK1 . . . . . . . ......
MOB3C . . . . . . . ......
ATPAF1 . . . . . . . ......
EFCAB14 . . . . . . . ......
PDZK1IP1 . . . . . . . ......
TAL1 . . . . . . . ......
CMPK1 . . 1 . . 1 . ......
RP11-511I2.2 . . . . . . . ......
FOXD2-AS1 . . . . . . . ......
FOXD2 . . . . . . . ......
SPATA6 . . . 1 . . . ......
BEND5 . . . 1 . . . ......
FAF1 . . . . . . . ......
CDKN2C . . . . . . . ......
RNF11 . . . . . . . ......
EPS15 . . 1 . . . . ......
OSBPL9 . . . . . . 1 ......
NRD1 . 1 . . . . . ......
TXNDC12 . . 1 . . . . ......
KTI12 . . . . . . . ......
BTF3L4 . . . . . 9 . ......
ZFYVE9 . . . . . . . ......
CC2D1B . . . . . . . ......
ORC1 . . . . . . . ......
PRPF38A . . . . . . 1 ......
ZCCHC11 1 . . . . . . ......
GPX7 . . . . . . . ......
FAM159A . . . . 2 . . ......
COA7 . . . . . . . ......
ZYG11B . . . . . . . ......
ECHDC2 . . . . . . . ......
SCP2 1 . . 1 1 . . ......
PODN . . . . . . . ......
SLC1A7 . . . . . . . ......
CPT2 . 1 . . . . . ......
C1orf123 . . . . . . . ......
RP5-1024G6.7 . . . . . . . ......
MAGOH . 1 1 . . 1 1 ......
RP4-784A16.5 . . . . . . . ......
RP11-117D22.2 . . . . . . . ......
NDC1 . . . . . . . ......
YIPF1 . . . . 1 . . ......
HSPB11 . . . . . . . ......
LRRC42 . . . . . . . ......
TMEM59 3 1 1 . . 1 1 ......
TCEANC2 . . . . . . . ......
CYB5RL . . . . . . . ......
MRPL37 . . . . . . 1 ......
SSBP3 . . 1 . . . . ......
ACOT11 . . . . . . . ......
TTC4 . . . . . . . ......
PARS2 . . . . . . . ......
TTC22 . . . . 1 . . ......
C1orf177 . . . . . . . ......
DHCR24 . . . . . . . ......
USP24 . . . . 1 . . ......
DAB1 . . . . . . . ......
OMA1 . . . . . . . ......
MYSM1 . . . . . . . ......
JUN 4 4 1 5 1 . 1 ......
LINC01135 . . . . . . . ......
RP4-794H19.1 . . . . . . . ......
FGGY . . . . . . . ......
HOOK1 . . . . . . . ......
CYP2J2 . . . . . . . ......
NFIA . . . . . . . ......
TM2D1 . 1 . . . . . ......
INADL . . . . . . . ......
L1TD1 . . . . . . . ......
USP1 . . . . . . . ......
DOCK7 . . . . . . . ......
ATG4C . . . . . . . ......
ALG6 . . . . . . . ......
ITGB3BP . . . . . . . ......
EFCAB7 . . . . . . . ......
DLEU2L . . . . . . . ......
PGM1 . . . . . . . ......
RAVER2 . . . . . . . ......
JAK1 . 1 1 6 1 1 . ......
AK4 . . . . . . . ......
LEPR . . . . . . . ......
LEPROT . . . . . . 1 ......
PDE4B . . . . . . . ......
MIER1 . . . . . 1 . ......
SLC35D1 . . . . . . . ......
IL23R . . . . . . . ......
IL12RB2 . . . . . . . ......
SERBP1 1 . . . . 2 . ......
GADD45A . . . . . . . ......
WLS . . . . . . . ......
LRRC7 . . . . . . . ......
LRRC40 . . . . . . . ......
SRSF11 . 1 . 1 . 1 . ......
ANKRD13C . . . . . . . ......
HHLA3 . . . . . . . ......
CTH . . . . . . . ......
ZRANB2 . . . . . . . ......
LRRIQ3 . . . . . . . ......
FPGT . . . . . . . ......
CRYZ . . 1 . 1 . . ......
TYW3 . . . . . . . ......
ACADM . . . . . . . ......
RABGGTB . . . . . 1 . ......
ST6GALNAC3 . . . . . . . ......
PIGK 1 1 . . . . . ......
AK5 . . . . . . . ......
ZZZ3 . . . . . . . ......
USP33 . . . . . . . ......
FAM73A . . . . . . . ......
NEXN . . . . . . . ......
FUBP1 . . . 1 . . . ......
DNAJB4 . . . . . . . ......
RP11-386I14.4 . . . . . . . ......
IFI44L . . . 9 1 8 2 ......
IFI44 . . . . . 2 . ......
RP5-887A10.1 1 . . . . . . ......
RP11-475O6.1 . . . . . . . ......
RP11-486G15.2 . . . . . . . ......
PRKACB . . . . . . . ......
RPF1 . 1 . . . . . ......
GNG5 . . . 1 . 1 1 ......
CTBS . . 1 . . 1 1 ......
SSX2IP . . . . . . . ......
MCOLN2 . . . . . . . ......
MCOLN3 . . . . . . . ......
SYDE2 . . . . . . . ......
C1orf52 . . . 1 . . . ......
BCL10 . . . . . . . ......
RP11-131L23.1 . . . . . . . ......
ZNHIT6 . . . . . . . ......
ODF2L . . . 1 . 2 . ......
RP4-612B15.3 . . . . . . . ......
SH3GLB1 . . . . . . . ......
SEP15 1 . . 1 . . 3 ......
HS2ST1 . . . . . . . ......
LMO4 . . . . . . 1 ......
PKN2 . . . . . . . ......
GTF2B . . . . . 1 1 ......
CCBL2 . . . . . . . ......
RBMXL1 . . . . . . . ......
GBP3 . . . . . . . ......
GBP1 . . . 1 . 2 2 ......
GBP2 . . . 1 1 1 3 ......
GBP4 . . . . . . . ......
GBP5 . . . . . 2 . ......
LRRC8B . . . . . . . ......
RP5-1007M22.2 . . . . . . . ......
LRRC8C . . . . 1 . . ......
RP5-943J3.2 . . . . . . . ......
LRRC8D . . . . . . . ......
ZNF326 . . . . . . . ......
ZNF644 . . . . . . . ......
CDC7 . . . . . . . ......
TGFBR3 . . . . . . . ......
BTBD8 . . . . . . . ......
KIAA1107 . . . . . . . ......
GLMN . . . . . . . ......
RPAP2 . . . . . . . ......
GFI1 . . . . . . . ......
EVI5 . . . . . . . ......
RPL5 12 6 16 7 10 8 19 ......
FAM69A . 1 . . . . . ......
MTF2 . . . . . . . ......
TMED5 . . . . . 1 . ......
CCDC18 . . . . . . . ......
DR1 1 . . . . . . ......
FNBP1L . . . . . . . ......
RP4-561L24.3 . . . . . . . ......
DNTTIP2 . . . . . . 1 ......
GCLM . . . . . . . ......
ARHGAP29 . . . . . . . ......
ABCD3 . . . . . . . ......
CNN3 . . . . . . . ......
ALG14 . . . . . . . ......
RWDD3 . . . . . . . ......
PTBP2 . . . . . . . ......
DPYD . . . . . . . ......
AGL . . . . . . . ......
RP5-884G6.2 . . . . . . . ......
SLC35A3 . . . . . . . ......
HIAT1 . . . . . . . ......
SASS6 . . 1 . . . . ......
TRMT13 . . . . . . . ......
DBT . . . . . . . ......
RTCA . . . . . . . ......
CDC14A . . . . . . . ......
EXTL2 . . . . . . . ......
RP4-549L20.3 . . . . . . . ......
SLC30A7 . . 1 . . . . ......
DPH5 . . . . . . . ......
AC093157.1 . . . . . . . ......
RP11-421L21.2 . . . . . . . ......
RP11-421L21.3 . . . . . . . ......
RP4-575N6.4 . . . . . . . ......
S1PR1 . . . . . . . ......
RNPC3 1 . . . 1 . . ......
AMY2B . . . . . . . ......
PRMT6 . . . . . . . ......
VAV3 . . . . . . . ......
VAV3-AS1 . . . . . . . ......
RP11-356N1.2 . . . . . . . ......
SLC25A24 . . . . . . . ......
RP11-483I13.5 . . . . . . . ......
FAM102B . . . . . . . ......
HENMT1 . . . . . . . ......
PRPF38B 1 . 1 . . . 1 ......
STXBP3 . . . . . . . ......
GPSM2 . . . . . . . ......
CLCC1 . . . . . . . ......
WDR47 . . . . . . . ......
TAF13 . . . . . . . ......
RP5-1065J22.8 . . . . . . . ......
TMEM167B . . . . 1 . 1 ......
KIAA1324 . . . . . . . ......
SARS . . . . . . 1 ......
CELSR2 . . . . . . . ......
PSRC1 . . . . . . . ......
SORT1 . . . . . . . ......
PSMA5 . . 1 . . . . ......
ATXN7L2 . . . . . . . ......
CYB561D1 . . . . . . . ......
AMIGO1 . . . . . . . ......
GPR61 . . . . . . . ......
GNAI3 . . . . . 1 1 ......
AMPD2 . . . 1 1 . . ......
GSTM4 . . . . . . . ......
GSTM2 . . . . . . . ......
GSTM1 . . . . . . . ......
GSTM3 . . . . . . . ......
CSF1 . . . . . . . ......
AHCYL1 . . . . . . . ......
STRIP1 . . . . . . . ......
RP4-773N10.4 . . . . . . . ......
KCNC4 . . . . . . . ......
RBM15 . . . . . . . ......
SLC16A4 . . . . . . . ......
RP5-1074L1.4 . . . . . . . ......
LAMTOR5-AS1 . . . . . . . ......
LAMTOR5 . . . . . . 1 ......
RP11-284N8.3 . . . . . . . ......
KCNA3 . . . . . . . ......
CD53 3 . 4 . 1 2 2 ......
RP11-96K19.2 . . . . . . . ......
LRIF1 . . . . . . . ......
RP11-96K19.5 . . . . . . . ......
DRAM2 1 . . . . . . ......
CEPT1 . . . . . . . ......
RP5-1180E21.5 . . . . . . . ......
DENND2D . . . . 1 . . ......
CHI3L2 . . . . . . . ......
PIFO . . . . . . . ......
OVGP1 . . . . . . . ......
WDR77 . . . . . . . ......
ATP5F1 . . 1 1 . . 1 ......
C1orf162 1 . . 1 . . 2 ......
ADORA3 . . . . . . . ......
RAP1A . . . 2 1 . 1 ......
FAM212B . . . . . . . ......
DDX20 . . . . 1 . . ......
CTTNBP2NL . . . . . . . ......
WNT2B . . . . . . . ......
ST7L . . . . . . . ......
CAPZA1 . 1 . . . 1 . ......
MOV10 . . . . . . . ......
RP11-426L16.3 . . . . . . . ......
RHOC . . . 3 . . 17 ......
PPM1J . . . . . . . ......
SLC16A1 . . . . . . . ......
SLC16A1-AS1 . . . . . . . ......
RP11-389O22.1 . . . . . . . ......
LRIG2 . . . . . . . ......
MAGI3 . . . . . . . ......
PHTF1 . . . . . . . ......
RSBN1 . 1 . . . . . ......
PTPN22 . . . . . . . ......
AP4B1-AS1 . . . . . . . ......
BCL2L15 . . . . . . . ......
AP4B1 . . . . . . . ......
DCLRE1B . . . . 1 . . ......
RP5-1073O3.7 . . . . . . . ......
HIPK1 . . . . . . . ......
TRIM33 . . . . 1 . . ......
BCAS2 . . . . . 1 . ......
DENND2C . . . . . . . ......
NRAS . . . . . . . ......
CSDE1 1 . 1 . 4 1 2 ......
SIKE1 . . . . 1 . . ......
TSPAN2 . . . . . . . ......
VANGL1 . . . . . . . ......
SLC22A15 . . . . . . . ......
ATP1A1 . . . . . 1 . ......
AL136376.1 . . . . . . . ......
CD58 . . . . . . . ......
CD2 . 1 . . 2 2 . ......
CD101 . . . . . . . ......
TTF2 . . . . . . . ......
MAN1A2 . . . . . . . ......
FAM46C . . . 1 . . . ......
GDAP2 . . . . . . . ......
WDR3 . . . . . . . ......
WARS2 1 . . . . . . ......
RP11-418J17.1 . . . . . . . ......
PHGDH . . . . . . . ......
REG4 . . . . . . . ......
NOTCH2 . . . 1 . . 1 ......
RP11-114O18.1 . . . . . . . ......
FAM72B . . . . . . . ......
FCGR1B . . . . . . . ......
RP11-343N15.5 . . . . . . . ......
RP11-344P13.6 . . . . . . . ......
RP11-423O2.5 . . . . . . . ......
RP11-782C8.1 . . . . . . . ......
RP6-206I17.1 . . . . . . . ......
RP6-206I17.2 . . . . . . . ......
LINC00623 . 1 . . . . . ......
AL592284.1 . . . . . . . ......
RP11-640M9.1 . . . . . . . ......
NBPF9 . . . . . . . ......
PDE4DIP . . . . . . . ......
AL590452.1 . . . . . . . ......
RP11-458D21.6 . . . . . . . ......
NOTCH2NL . . . 1 . 1 . ......
NBPF10 . . . 1 . . . ......
TXNIP . . 2 2 5 1 5 ......
POLR3GL . . . 1 . . . ......
ANKRD34A . . . . . 1 . ......
RP11-315I20.1 . . . . . . . ......
LIX1L . . . . . . . ......
RBM8A . . . . . 1 1 ......
PEX11B . . . . . . . ......
ITGA10 . . . . . . . ......
ANKRD35 . . . . . . . ......
PIAS3 . . . . . . . ......
NUDT17 . . . . . . . ......
POLR3C . . . . . . . ......
RNF115 . . . . . . . ......
CD160 . . . . . . . ......
GPR89A . . . . . . . ......
NBPF12 . . 3 . . . . ......
PRKAB2 . . . . . . . ......
RP11-337C18.8 . . . . . . . ......
FMO5 . . . . . . . ......
CHD1L . . . . . 1 . ......
BCL9 . . . . . . . ......
ACP6 . . . . . . . ......
NBPF14 . . . . . . . ......
RP11-89F3.2 . . . . . . . ......
NBPF20 . . . . . . . ......
NBPF15 . . . . . . . ......
RP11-666A1.5 . . . . . . . ......
RP11-14N7.2 . . . . . . . ......
RP11-403I13.4 . . . . . . . ......
RP11-403I13.8 . . . . . . . ......
RP11-277L2.3 . . . . . . . ......
RP11-353N4.4 . . . . . . . ......
FCGR1A . . . . . . . ......
HIST2H2BF . . . . . . . ......
RP11-196G18.24 . . . . . . . ......
HIST2H2BE . . . . . . . ......
HIST2H2AC . . . . . . . ......
HIST2H2AB . . . . . . . ......
BOLA1 . . . . . . . ......
SV2A . . . . . . . ......
SF3B4 . . 1 . 1 . . ......
MTMR11 . . . . . . . ......
OTUD7B . . . . . . . ......
VPS45 . . . . . . . ......
PLEKHO1 . . . . . . . ......
ANP32E . . . . . . . ......
APH1A 1 . . . 1 . . ......
C1orf54 . . . . . . . ......
C1orf51 . . . . . . . ......
MRPS21 . . . . 2 . . ......
PRPF3 1 . . . . . . ......
RPRD2 1 . . . . . . ......
TARS2 . . . . . . . ......
ADAMTSL4 . . . . . . . ......
MCL1 . 1 . . 1 1 . ......
ENSA 2 . . . . . 2 ......
GOLPH3L . . . . . . . ......
CTSS 1 1 . 7 2 2 10 ......
CTSK . . . . . . . ......
ARNT . 1 . . . . . ......
SETDB1 . . . . . . . ......
CERS2 . . . . . 1 . ......
FAM63A . . . . . . . ......
PRUNE . . . . . . . ......
C1orf56 . . . . . . . ......
CDC42SE1 . . 1 . . . . ......
MLLT11 . . . . . . . ......
GABPB2 . . . . . . . ......
TNFAIP8L2 . . . 1 . . . ......
SCNM1 1 . . . . . . ......
VPS72 . . . 1 . . . ......
PIP5K1A . . . . . . . ......
PSMD4 . . . . . 1 2 ......
RP11-126K1.2 . . . . . . . ......
ZNF687 . . . . . 1 . ......
PI4KB . 1 . . . . . ......
RFX5 . . . . . . 1 ......
RP11-126K1.6 . . . . . . . ......
PSMB4 . . . . . . . ......
POGZ . . 1 . . . . ......
TUFT1 . . . . . . . ......
SNX27 . . . . . . . ......
MRPL9 1 . . 1 . . . ......
OAZ3 . . . . . . . ......
RP11-98D18.3 . . . . . . . ......
TDRKH . . . . . . . ......
RP11-98D18.9 . . . . . . . ......
RORC . . . . . . . ......
C2CD4D . . . . . . . ......
THEM4 . . . . . . . ......
S100A10 . . 2 4 . . 2 ......
S100A11 . . . 5 . 1 5 ......
S100A9 . . . . 1 . 2 ......
S100A12 . . . . . . . ......
S100A8 . . 1 . . . . ......
S100A6 1 . 3 11 . 1 10 ......
S100A5 . . . . . . . ......
S100A4 3 . 2 15 . 3 21 ......
S100A13 . . . . 1 . . ......
CHTOP . . . 1 . . . ......
SNAPIN . . . . . 1 . ......
ILF2 . . . . . 1 . ......
INTS3 . . . . . . . ......
SLC27A3 . . . . . . . ......
GATAD2B . . . . 1 . . ......
DENND4B . . . . . . 1 ......
CRTC2 . . . . . . 1 ......
SLC39A1 . . . . . . . ......
CREB3L4 . . . . . . . ......
JTB 1 . . . 2 1 . ......
RP11-422P24.11 . . . . . . . ......
RAB13 . . . . . . . ......
RPS27 19 13 25 44 37 25 17 ......
TPM3 . . 1 2 . . 2 ......
C1orf43 . 1 . . . 1 13 ......
UBAP2L . . . . . . . ......
HAX1 . . . 1 . 1 . ......
AQP10 . . . . . . . ......
ATP8B2 . . . . . . . ......
RP11-350G8.5 . . . . . . . ......
IL6R . . . . . . . ......
UBE2Q1 . . . . . . . ......
ADAR . . . . . 1 . ......
PMVK . . 1 . . . . ......
RP11-307C12.13 . . . . . . . ......
PBXIP1 . . 1 . . . . ......
PYGO2 . . . . . . . ......
RP11-307C12.12 . . . . . . . ......
SHC1 . . . . . . . ......
CKS1B . 1 . . . . 1 ......
FLAD1 . . . . . . . ......
ZBTB7B . . . . . . . ......
ADAM15 . . . . . . . ......
EFNA4 . . . . . . . ......
EFNA3 . . . . . . . ......
SLC50A1 . . . . . . 1 ......
DPM3 . . . . . . . ......
KRTCAP2 . . . 1 . 1 . ......
TRIM46 . . . . . . . ......
THBS3 . . . 1 . . . ......
MTX1 . . . . . . . ......
GBA . . . . . . . ......
FAM189B . . . . 1 . . ......
SCAMP3 . . . . . 1 . ......
CLK2 . . . . . . . ......
FDPS . . . . . . . ......
RUSC1-AS1 . . . . . . . ......
RUSC1 . . . . . . . ......
ASH1L . . . . . . . ......
MSTO1 . . . . . . . ......
YY1AP1 . . . . . . . ......
DAP3 . . . 1 . . . ......
GON4L . . . . . . . ......
SYT11 . . . . . . . ......
RIT1 . . . . . . . ......
KIAA0907 . . 1 . . . . ......
ARHGEF2 1 . . . . 1 . ......
RP11-336K24.12 . . . . . . . ......
SSR2 . . 1 2 1 . . ......
UBQLN4 . . . . . . . ......
LAMTOR2 . . . . . . . ......
RAB25 . . . . . . . ......
LMNA . . . . . . 1 ......
SEMA4A . . . . . . . ......
SLC25A44 . . . . . . . ......
PMF1 . . . . . . . ......
BGLAP . . . . . . . ......
SMG5 . . . . . . . ......
TMEM79 . . . . . . . ......
C1orf85 . . . . 1 . . ......
CCT3 . . 1 1 . 1 2 ......
C1orf61 . . . . . . . ......
MEF2D . . . . . . . ......
TTC24 . . . . . . . ......
APOA1BP . . 1 . . . 1 ......
GPATCH4 . . . . . . . ......
ISG20L2 . 1 . . . . . ......
RRNAD1 . . . . . . . ......
MRPL24 . . . . . . . ......
HDGF . . . . . . . ......
PRCC . . . . . . 1 ......
SH2D2A . . . . . . . ......
ARHGEF11 . . . . . . . ......
ETV3 . . . . . . . ......
FCRL5 . . . . . . . ......
FCRL3 . . . . . . . ......
FCRL2 . . . . . . . ......
FCRL1 . . . . . . . ......
CD1D . . . . . . . ......
CD1A . . . . . . . ......
CD1C . . . . . . . ......
CD1B . . . . . . . ......
CD1E . . . . . . . ......
MNDA . . . . . . . ......
PYHIN1 . . . . . . . ......
IFI16 . . . . . 3 1 ......
AIM2 . . . . . 1 . ......
FCER1A . . . . . . . ......
DUSP23 1 . . 2 . . . ......
FCRL6 . . . . . . . ......
SLAMF8 . . . . . . . ......
C1orf204 . . . . . . . ......
VSIG8 . . . . . . . ......
TAGLN2 2 1 . 3 . 1 . ......
PIGM . . . . . 1 . ......
IGSF8 . . . . . . . ......
RP11-536C5.7 . . . . . 1 . ......
PEA15 . . . . . . . ......
DCAF8 . . . . . . . ......
PEX19 . . . . . . . ......
COPA . . . 1 1 . . ......
NCSTN . . . . . . . ......
SLAMF6 . . . . 1 1 . ......
CD84 . 1 . . . . . ......
SLAMF1 . . . . . . . ......
RP11-404F10.2 . . . . . . . ......
CD48 1 . . 5 2 2 2 ......
SLAMF7 . . 1 . . . . ......
LY9 . . . 1 1 . . ......
CD244 . . . . . . . ......
ITLN1 . . . . . . . ......
F11R . . . . . . . ......
TSTD1 . . . . 1 . . ......
USF1 . . . . . . . ......
ARHGAP30 . . . . 1 . 1 ......
KLHDC9 . . . . . . . ......
PFDN2 . . . 2 . . . ......
NIT1 . . . . . 1 . ......
DEDD . . . . . . . ......
UFC1 . . . 1 1 . . ......
USP21 . . . . . . . ......
PPOX . . . . . . . ......
B4GALT3 . . . . . . . ......
NDUFS2 1 . . 1 1 . . ......
FCER1G . 1 . 13 . . 13 ......
TOMM40L . . . . . . . ......
MPZ . . . . . . . ......
SDHC . . . 2 . . . ......
FCGR2A . . . . . . 4 ......
HSPA6 . . . . . . 2 ......
FCGR3A . . . 4 1 . 23 ......
FCGR2B . . . . . . . ......
FCGR3B . . . . . 1 . ......
FCRLA . . . . . . . ......
FCRLB . . . . . . . ......
DUSP12 . . . . . . . ......
ATF6 . . . . . . 1 ......
SH2D1B . . . . . . . ......
UHMK1 . . . . . . 1 ......
RP11-359K18.3 . . . . . . . ......
UAP1 . . . . . . . ......
HSD17B7 . 1 . . . . . ......
RP11-267N12.3 . 1 . . . . . ......
NUF2 . . . . . . . ......
PBX1 . . . . . . . ......
MGST3 . . . . 1 1 1 ......
ALDH9A1 . . . . . . 1 ......
TMCO1 . . . . . . . ......
RP11-466F5.8 . . . . . . . ......
UCK2 . . . . . . . ......
POGK . . . . . . . ......
TADA1 . . . . . . . ......
GPA33 . . . . . . . ......
RP11-277B15.3 . . . . . . . ......
POU2F1 . . . . . . . ......
CD247 . . 1 . . 1 . ......
RP11-104L21.3 . . . . . . . ......
CREG1 . . . . . . . ......
RP3-455J7.4 . . . . . . . ......
RCSD1 . . . 2 . 1 . ......
MPZL1 . . . . . . . ......
MPC2 . . . . . . . ......
DCAF6 . . . . . . . ......
TIPRL . . . . . . . ......
SFT2D2 . . . . . . . ......
TBX19 . . . . . . . ......
XCL2 . . . . . . . ......
XCL1 . . . . . . . ......
ATP1B1 . . . . . . . ......
NME7 . . . . . . . ......
BLZF1 . . . . . . . ......
SLC19A2 . 1 . . . . . ......
F5 . . . . . . . ......
SELP . . . . . . . ......
C1orf112 . . . . . . . ......
SELL 1 1 2 . 1 . 2 ......
METTL18 1 . . . . . . ......
SCYL3 . . . . . . . ......
KIFAP3 . . . . 1 . . ......
GORAB . . . . . . 1 ......
FMO4 . . . . . . . ......
PRRC2C . 1 . 1 1 . . ......
VAMP4 . . . . . . . ......
METTL13 . . . . . . . ......
DNM3 . . . . . . . ......
PIGC . . . . . . 1 ......
SUCO . . . . . . . ......
FASLG . . . . . . . ......
TNFSF4 . . . . . . . ......
RP11-296O14.3 . . . . . . . ......
PRDX6 2 . 2 1 . 1 1 ......
KLHL20 . . . . . . . ......
CENPL . . . . . . . ......
DARS2 . . . . . . . ......
ZBTB37 . 1 . . . . . ......
RC3H1 . . . . . . . ......
RP11-160H22.5 . . . . . . . ......
RABGAP1L . . . . . . 1 ......
CACYBP 1 . . . . . . ......
MRPS14 . . . . . . . ......
KIAA0040 . . . . . . . ......
RP11-318C24.2 . . . . . . . ......
RFWD2 . . . . . . . ......
RP5-1098D14.1 . . . . . . . ......
RALGPS2 . . . . . . . ......
FAM20B . . . . . . . ......
TOR3A . . . 1 . . . ......
ABL2 . . . . . . . ......
SOAT1 . . . . . . . ......
TOR1AIP2 1 . . . . . . ......
RP11-533E19.7 . . . . . . . ......
TOR1AIP1 . . . . . 1 1 ......
RP11-533E19.5 . . . . . . . ......
CEP350 . . . . . . . ......
QSOX1 . . . . . . . ......
RP5-1180C10.2 . . . . . . . ......
ACBD6 . . 1 . . . . ......
XPR1 . . . . . . . ......
RP11-46A10.5 . . . . . . . ......
STX6 . . . . . . . ......
MR1 . . . . . . . ......
IER5 1 . . . . . . ......
RP11-309G3.3 . . . . . . . ......
GLUL . . . . . . . ......
RNASEL . . . . . . . ......
RGS16 . . . . . . . ......
NPL . . . . . . 1 ......
DHX9 . . . . . . . ......
LAMC1 . . . . . . . ......
SMG7 . . . . . . . ......
NCF2 . . . . . . . ......
ARPC5 1 1 . . . . 3 ......
RGL1 . . . . . . . ......
COLGALT2 . . . . . . . ......
TSEN15 . . . . . . . ......
C1orf21 . . . . . . . ......
EDEM3 . . . . . 1 . ......
FAM129A . . . . . . . ......
RNF2 . . 1 . . . . ......
TRMT1L . . . . . . . ......
SWT1 . . . . . . . ......
IVNS1ABP . . . . . . . ......
TPR . . . . . . . ......
C1orf27 . . . . . . . ......
PTGS2 . . . . . . . ......
RGS18 . . . 1 . . . ......
RGS1 . . . . . . . ......
RGS2 . . . 2 . 1 3 ......
UCHL5 . . . . . . . ......
TROVE2 . . 1 . . . . ......
RP11-101E13.5 . . . . . . . ......
GLRX2 . . . . . . . ......
CDC73 . . . . . . . ......
B3GALT2 . . . . . . . ......
CFH . . . . . . . ......
ASPM . . . . . . . ......
ZBTB41 . . . . . . . ......
CRB1 . . . . . . . ......
DENND1B . . . . . . . ......
C1orf53 . . . . . . . ......
NEK7 . . . . . . . ......
RP11-553K8.2 . . . . . . . ......
PTPRC 1 . 2 2 . 1 . ......
MIR181A1HG . . . . . . . ......
ZNF281 . . . . . . . ......
DDX59 . . . . . . . ......
CAMSAP2 . . . . . . . ......
KIF21B . . . . . . . ......
TMEM9 . . . . . . . ......
PHLDA3 . . . . . . . ......
CSRP1 . . . . . . 1 ......
RP11-134G8.7 . . . . . . . ......
RPS10P7 . . . . . . . ......
NAV1 . . . . . . . ......
IPO9-AS1 . . . . . . . ......
IPO9 . . . . . . . ......
SHISA4 . . . 1 . . . ......
TIMM17A . . . 1 . 1 . ......
RNPEP . . . . . . 1 ......
ARL8A . . . . . . . ......
PTPN7 1 . . . . . . ......
LGR6 . . . . . . . ......
UBE2T . . . . . . . ......
PPP1R12B . . . . . . . ......
KDM5B . . . 1 . . . ......
RABIF . . . . . . . ......
KLHL12 . . . . . . . ......
ADIPOR1 . . . 1 1 1 . ......
CYB5R1 . . . . . . . ......
TMEM183A 2 . . . . 1 . ......
RP11-134P9.3 . . . . . . . ......
LINC01136 . . . . . . . ......
BTG2 . . 1 2 2 . 1 ......
ATP2B4 . . . 1 . . . ......
LAX1 . . . . . . . ......
ZC3H11A . . . . . . . ......
ZBED6 . . . . . 1 . ......
SNRPE . . . . . . 1 ......
SOX13 . . . . . . . ......
PPP1R15B . . . . . . . ......
PIK3C2B . . . . . . . ......
MDM4 . . . . . . . ......
TMEM81 . . . . . . . ......
RBBP5 . . . 1 . . . ......
DSTYK . . . . 1 . . ......
TMCC2 . . . . . . . ......
NUAK2 . . . . . . . ......
CDK18 . . . . . . . ......
MFSD4 . . . . . . . ......
ELK4 . . . . . . . ......
SLC45A3 . . . . . . . ......
NUCKS1 1 . . . . . . ......
RAB7L1 . . . . . . . ......
SLC41A1 . . . . . . . ......
PM20D1 . . . . . . . ......
FAM72A . . . . . . . ......
C1orf186 . . . . . . . ......
SRGAP2 . . . 1 . . . ......
IKBKE . . . . . . . ......
RASSF5 . . . 3 . . . ......
EIF2D . . . . 1 . . ......
RP11-343H5.6 . . . . . . . ......
MAPKAPK2 . . . . . . . ......
IL10 . . . . . . . ......
IL24 . . . . . . . ......
FAIM3 . . . . . . . ......
YOD1 . . . . . . . ......
PFKFB2 . . . . . . . ......
C4BPB . . . . . . . ......
CD55 . . . 1 . 2 2 ......
CR2 . . . . . . . ......
CR1 . . . . . . . ......
CD46 . . . . 1 . . ......
RP1-28O10.1 . . . . . . . ......
G0S2 . . . 4 . . . ......
HSD11B1 . . . . . . . ......
TRAF3IP3 . . . . 2 1 1 ......
DIEXF . . . . . . . ......
HHAT . . . . . . . ......
RCOR3 . . . . . . 1 ......
TRAF5 . . . . . . . ......
SLC30A1 . . . 1 . . . ......
NEK2 . . . . . . . ......
LPGAT1 . . . . . . . ......
INTS7 . . . . . . . ......
PPP2R5A 2 . . . . . . ......
TMEM206 . . . . . . . ......
NENF 1 . . . . . . ......
RP11-61J19.4 . . . . . . . ......
ATF3 . . . . . . . ......
RP11-338C15.3 . . . . . . . ......
BATF3 . . . . . . . ......
NSL1 . . . . . . . ......
TATDN3 . . . 1 . . . ......
FLVCR1-AS1 . . . . . . . ......
FLVCR1 . . . . . . . ......
ANGEL2 . . . . . . . ......
RPS6KC1 . . . . . . . ......
SMYD2 . 1 . . . . . ......
CENPF . . . . . . . ......
KCTD3 . . . . . . . ......
GPATCH2 . . . . . . . ......
RRP15 . . . . . . . ......
LYPLAL1 . . . . . . . ......
EPRS . . . 1 1 . 1 ......
BPNT1 . . . . . . . ......
IARS2 . . . . 1 . . ......
RAB3GAP2 . . . . . . . ......
C1orf115 . . . . . . . ......
MARC2 . . . . . . . ......
MARC1 . . . . . . . ......
HLX . . . . . . . ......
DUSP10 . . . . . . . ......
TAF1A . . . . . . . ......
RP11-378J18.3 . . . . . . . ......
MIA3 . . . . . 2 . ......
AIDA . . . . . . . ......
BROX . . . . . . . ......
FAM177B . . . . . . . ......
RP11-452F19.3 . . . 1 . . . ......
DISP1 . . . . . . . ......
TLR5 . . . . . . . ......
SUSD4 . . . . . . . ......
CAPN8 . . . . . . . ......
CAPN2 . . . 1 . . . ......
TP53BP2 . . . . . . . ......
RP11-504P24.8 . . . . . . . ......
RP11-504P24.6 . . . . . . . ......
FBXO28 . . . . . . . ......
DEGS1 . . . . . . . ......
NVL . . . 1 . . . ......
CNIH4 . . . . 1 . . ......
WDR26 . . . . . . 1 ......
CNIH3 . . . . . . . ......
LBR . . . . 1 . 1 ......
ENAH . . . . . . . ......
SRP9 . . . 1 1 . 1 ......
EPHX1 . . . . . . . ......
RP11-285F7.2 . . . . . . . ......
TMEM63A 1 . . . 2 . . ......
PYCR2 . . . . . . . ......
SDE2 . . . . . . . ......
H3F3A 2 . . . . 1 2 ......
ACBD3 1 . . . . . . ......
LIN9 . . . . . . . ......
PARP1 . . . . . . . ......
ITPKB . . . . . . . ......
ITPKB-AS1 . . . . . . . ......
PSEN2 . . . . . . . ......
ADCK3 . 1 . . . . . ......
ZNF678 . . . . . . . ......
SNAP47 . . . . . . . ......
JMJD4 . . . . . . . ......
ARF1 1 . . . . 2 1 ......
C1orf35 . . . 1 1 . . ......
MRPL55 . . 1 . 2 . . ......
GUK1 1 1 1 2 . 2 2 ......
IBA57 . . . . . . . ......
C1orf145 . . . . . . . ......
OBSCN . . . . . . . ......
TRIM11 . . . . . . . ......
HIST3H2A . . . . . . . ......
RNF187 . . 1 1 . . . ......
RHOU . . . . . . . ......
RAB4A . 1 . . . . . ......
RP4-803J11.2 . . . . . . . ......
CCSAP . . . . . . . ......
NUP133 . . . . . . . ......
ABCB10 . . . . . . . ......
TAF5L . . . . . . . ......
URB2 . . . . . . . ......
RP11-552D4.1 . . . . . . . ......
GALNT2 . . . . . . 1 ......
COG2 . . 1 . . . . ......
C1orf198 . . . . . . . ......
TTC13 . . . . . . . ......
ARV1 . . . . . . . ......
FAM89A . . . . . . . ......
C1orf131 . . . . . . . ......
GNPAT 1 . . . . 1 . ......
EXOC8 . . . . . . . ......
SPRTN . . . . . . . ......
EGLN1 . . . . . . . ......
RP11-295G20.2 . . . . . . . ......
TSNAX . . . . . . . ......
DISC1 . . . . . . . ......
SIPA1L2 . . . . . . . ......
MAP10 . . . . . . . ......
NTPCR . . . . . . . ......
PCNXL2 . . . . . . . ......
SLC35F3 . . . . . . . ......
RP5-827C21.4 . . . . . 1 . ......
COA6 . . . . . 28 . ......
TARBP1 . . . . . . . ......
IRF2BP2 . . . . . . . ......
RP4-781K5.2 . . . . . . . ......
RP11-443B7.1 . . . . . . . ......
RP11-443B7.3 . . . . . . . ......
TOMM20 1 . . . 2 1 3 ......
RBM34 . . . . . 1 . ......
ARID4B . . . . 1 . . ......
GGPS1 . . . . . . . ......
TBCE . . . . . . . ......
B3GALNT2 . . . . . . . ......
LYST . . . 1 . . 1 ......
NID1 . . . . . . . ......
GPR137B . . . . . . . ......
ERO1LB . . . . . . . ......
EDARADD . . . . . . . ......
LGALS8 . . . . . . . ......
LGALS8-AS1 . . . . . . . ......
RP11-385F5.5 . . . . . . . ......
HEATR1 . . . . . . . ......
MTR . . . . . . . ......
GREM2 . . . . . . . ......
FH . . . . . . . ......
KMO . . . . . . . ......
OPN3 . . . . . . . ......
CHML . . . . . . . ......
RP11-261C10.3 . . . . . . . ......
CEP170 . . . . . . . ......
SDCCAG8 . . . . . . . ......
AKT3 . . . . . . . ......
ZBTB18 . . . . . . . ......
ADSS . . . . . 1 . ......
C1orf101 . . . . . . . ......
DESI2 . . . . . . . ......
COX20 . . . . . 4 . ......
HNRNPU-AS1 . . . . . . . ......
HNRNPU 1 . . . . . . ......
RP11-11N7.4 . . . . . . . ......
RP11-156E8.1 . . . . . . . ......
EFCAB2 . . . . . . . ......
SMYD3 . . . . . . . ......
TFB2M . . . . . . . ......
CNST . . . . . . . ......
SCCPDH . . . . 1 . . ......
AHCTF1 . . . . 1 . . ......
ZNF670 . . . . . . . ......
ZNF669 . . . . . . . ......
ZNF124 . . . . 1 . . ......
RP11-488L18.10 . . . . . . . ......
RP11-488L18.8 . . . . . . . ......
ZNF496 . . . . . . . ......
NLRP3 . . . . . . . ......
TRIM58 . . . . . . . ......
SH3BP5L . . . . . . . ......
ZNF672 . . . . . . . ......
ZNF692 . . . . . . . ......
PGBD2 . . . . . . . ......
SH3YL1 . . . . 1 . . ......
ACP1 . . 1 . . . . ......
TMEM18 . . . . . . . ......
AC116614.1 . . . . . . . ......
TPO . . . . . . . ......
TSSC1 . . . . . . . ......
TRAPPC12 . . . . . . . ......
TRAPPC12-AS1 . . . . . . . ......
ADI1 . . . . 1 . . ......
AC142528.1 . . . . . . . ......
AC108488.4 . . . . . . . ......
RNASEH1 . . . . . 1 1 ......
RNASEH1-AS1 . . . . . . . ......
RPS7 13 11 12 14 9 15 11 ......
CMPK2 . . . . . . . ......
RSAD2 . . . . . 1 . ......
RNF144A . . . . . . 1 ......
AC092580.4 . . 1 . . . . ......
LINC00299 . . . . . . . ......
AC011747.4 . . . . . . . ......
ID2 . . . 3 . . . ......
KIDINS220 . . . . . . . ......
MBOAT2 . . . . . . . ......
ASAP2 . . . . . . . ......
ITGB1BP1 . . 1 1 . . . ......
CPSF3 . . . . . . . ......
IAH1 1 . . 1 . . 1 ......
ADAM17 . . . . . . . ......
RP11-214N9.1 . . . . . . . ......
YWHAQ . . 1 2 . . 2 ......
TAF1B . . . . . 1 . ......
RP11-95D17.1 . . . . . . . ......
RP11-254F7.2 . . . . . . . ......
KLF11 . . . . . . . ......
RRM2 . . . . . . . ......
HPCAL1 1 . . . . . . ......
ODC1 . . . . . . . ......
NOL10 1 . . . 1 . . ......
RN7SL832P . . . . . . . ......
ATP6V1C2 . . . . . . . ......
RP11-791G15.2 . . . . . . . ......
PDIA6 1 . . 2 . . 3 ......
PQLC3 . . . 1 . . . ......
ROCK2 . . . . . . . ......
E2F6 . . . . . . . ......
LPIN1 . . . . 1 . . ......
TRIB2 . . . . . . . ......
NBAS . . . . . . . ......
DDX1 . . . . . . . ......
FAM49A . . . 1 . . . ......
SMC6 . . . . . . . ......
GEN1 . . . . . . . ......
RP11-111J6.2 . . . . . . . ......
RDH14 . . . . . . . ......
LINC00954 . . . . . . . ......
TTC32 . . . 1 . . . ......
WDR35 . . . . . . . ......
LAPTM4A . . . 1 . . 2 ......
PUM2 . . . . 1 . . ......
RHOB 1 . . . . . 1 ......
HS1BP3 . . . . . . . ......
C2orf43 . . . . . 1 . ......
KLHL29 . . . . . . . ......
ATAD2B . . . . . . . ......
UBXN2A . . . . 1 . . ......
MFSD2B . . . . . . . ......
C2orf44 . . . . . . . ......
FKBP1B . . . . . . . ......
SF3B14 . . . . . . . ......
FAM228B . . . . . . . ......
TP53I3 . . . . . . . ......
FAM228A . . . . . . . ......
ITSN2 . . . . . . . ......
NCOA1 . . . . . . . ......
PTRHD1 . . . . . . . ......
CENPO . . . . . . . ......
ADCY3 . . . . . . . ......
DNAJC27 . . . . . . . ......
DNAJC27-AS1 . . . . . . . ......
POMC . . . . . 1 . ......
DNMT3A . . . . . . . ......
AC012074.2 . . . . . . . ......
DTNB . . . . . . . ......
AC104699.1 . . . . . . . ......
ASXL2 . . . . . . 1 ......
KIF3C . . . . . . . ......
AC013449.1 . . . . . . . ......
RAB10 . . . . . . 2 ......
GAREML . . . . . . . ......
HADHA . . . 1 . . . ......
HADHB . . . . . . . ......
EPT1 . . . . . . . ......
OTOF . . . . . . . ......
SLC35F6 . . . . . . . ......
CENPA . . . . . . . ......
MAPRE3 . . . . . . . ......
TMEM214 . . . . . . . ......
AGBL5 . . . . . . 1 ......
OST4 2 . . . . 3 1 ......
KHK . . . . . . . ......
ABHD1 . . . . . . . ......
PREB . . . . . . . ......
SLC5A6 . . . . . . . ......
ATRAID . . . 2 . 1 2 ......
CAD . . . . . . . ......
TRIM54 . . . . . . . ......
MPV17 . . 1 . . . . ......
GTF3C2 . . . . . . . ......
AC074117.10 . . . . . . . ......
EIF2B4 . . . . . . . ......
SNX17 . . . . . . . ......
ZNF513 . . . . . . . ......
PPM1G . . . . . . 1 ......
NRBP1 . . . . . . . ......
KRTCAP3 . . . . . . . ......
IFT172 . . . . . . . ......
ZNF512 . . . . . . . ......
CCDC121 . . . . . . . ......
GPN1 . . . . . 1 . ......
SUPT7L . . . . . . . ......
SLC4A1AP . . . . . . . ......
MRPL33 . . . . . 1 . ......
RBKS . . . . . . . ......
BRE . . . . . . . ......
FOSL2 . . . . . . . ......
PLB1 . . . . . . . ......
PPP1CB . . . . . . . ......
..............................
........suppressing 2666 columns and 10590 rows in show(); maybe adjust options(max.print=, width=)
.............................. [[ suppressing 32 column names 'AAACATACAACCAC-1', 'AAACATTGAGCTAC-1', 'AAACATTGATCAGC-1' ... ]]
HMHA1 . 2 1 . . . 1 1 . . . . . . . . . . 1 . 2 . . 1
POLR2E . . . 1 . . . . 1 . . . . . . . . . . 1 1 . . .
GPX4 . 1 2 1 . 1 1 . . 1 . 1 1 1 1 . 3 . 3 1 2 1 2 .
SBNO2 . . . . . . . . . . . . . . . . . . . . . . . .
STK11 . . . . . . . . . . . . . . . . . . . . . . . .
ATP5D 1 1 1 1 . . 1 2 . . 1 1 . 1 . . . . 4 . 1 . 1 .
MIDN . . . . . . . . . . . . . 1 . . . . . . . . 3 .
CIRBP 1 1 2 . 2 . 1 2 1 . 3 5 2 . 2 . . . 2 1 4 1 . 2
C19orf24 1 . . . . . 1 . . . . . 1 1 . . . . . 1 1 . . .
MUM1 . . . . . . . . . . . . . . . . . . . . . . . .
NDUFS7 . 2 . 1 . 1 . . . 1 . . 1 . . . . . 2 . . . 2 .
GAMT . . . . . . . 1 . . . . . . . . . . . . . . . .
DAZAP1 . . . . . . . . 1 . . . . . 1 . . . . . . . 1 .
RPS15 22 19 15 9 1 11 11 13 2 3 18 12 19 7 4 2 8 2 34 17 41 7 16 6
AC027307.3 . . . . . . . . . . . . . . . . . . . . . . . .
C19orf25 . . . . . 1 . . . . 1 . . . . . . . 1 . . . . .
PCSK4 . . . . . . . . . . . . . . . . . . . . . . . .
REEP6 . . . . . . . . . . . . . . . . . . . . . . . .
ADAMTSL5 . . . . . . . . . . . . . . . . . . . . . . . .
MBD3 . . . . . . . . . . . . . . . . . . 1 . . . . .
UQCR11.1 . 1 4 2 2 1 1 . . 2 2 . . 1 . 1 . . 1 1 2 1 . .
TCF3 . . . . . . . 1 . . . . . . . . . . . . . . . .
ATP8B3 . . . . . . . . . . . . . . . . . . . . . . . .
REXO1 . . . . . . . . . . . . . . . . . . . . . . . .
KLF16 . . . . . . . . . . . . . . . . . . . . . . . .
CTB-31O20.2 . . . . . . 1 . . . . . . . . . . . . . . . . .
ABHD17A . . . . . . . . . . . . . . . . . . . . . . . .
SCAMP4 . 1 . . . . . . . . . . 1 . . . . . . . . . . .
ADAT3 . . . . . . . . . . . . . . . . . . . . . . . .
CSNK1G2 . . . . . . . . . . . . . . . . . . . . . . . .
BTBD2 . . . . . . . . . . . . . . . . . . . . . . . .
AC005306.3 . . . . . . . . . . . . . . . . . . . . . . . .
MKNK2 . . 1 . . . . . . . 1 . . 1 . . . . 2 . . . . .
MOB3A . . . . . . . . . . . . . . . . . . 2 . . . . .
IZUMO4 1 . . . . . . . . . . . . . . . . . . . . . . .
AP3D1 . . . . . . . . . . . . . . . . . . . . . . . .
DOT1L . . . . . . . . . . . . . . . . . . . . . . . .
PLEKHJ1 . . 1 . . . . . . . . . 1 . . . . . . . . . . .
SF3A2 . . . . . . . . 1 . . . . . . . . . . . . . . .
JSRP1 . . . . . . . . . . . . . . . . . . . . . . . .
OAZ1 5 9 4 13 2 2 4 3 1 3 10 1 8 8 1 2 4 3 30 1 4 5 3 2
C19orf35 . . . . . . . . . . . . . . . . . . . . . . . .
LINGO3 . . . . . . . . . . . . . . . . . . . . . . . .
LSM7 . . 3 1 . 1 . . . . 1 1 1 . . . . 1 8 1 7 . 1 .
TIMM13 . 1 . . . 6 . . . . . . . . . . . 1 2 . 1 . . .
LMNB2 . . . . . . . . . . . . . . . . . . . . . . . .
GADD45B 1 . 2 . . . . . . . 1 . . . . . . . 2 1 . . . .
GNG7 . . . . . . . . . . 2 . . . . . . . . . . 1 . .
DIRAS1 . . . . . . . . . . . . . . . . . . . . . . . .
SLC39A3 . . . . . . . 1 . . . . . . . . . . . . . . 1 .
SGTA . . . . . . . . . . . . . . . . . . . . . . . .
THOP1 . . . . . . . . . . . . . . . . . . 1 . . . . .
ZNF554 . . . . . . . . . . . . . . . . . 1 . . . . . .
ZNF555 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF57 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF77 . . . . . . . . . . . . . . . . . . . . . . . .
TLE2 . . 1 . . . . . . . . . 1 . . . . . . . . . . .
AC005944.2 . . . . . . . . . . . . . . . . . . . . . . . .
AES . 2 1 . 1 2 1 1 . . 1 . . . . 1 . . . 2 1 . . 1
GNA11 . . . . . . . . . . . . . . . . . . . . . . . .
GNA15 . . . 2 . . . . . . . . . . . . . . 1 . . . . .
S1PR4 . . 1 1 . 1 3 1 . . . . . . . . . 1 . 1 . . . .
NCLN . . . . . . . . . . . . . . . . . . . . . . . .
NFIC . . . . . . . . . . . . . . . . . . . . . . . .
C19orf77 . . . . . . . . . . . . . . . . . . . . . . . .
DOHH . . . . . . . . . . . . . . . . . . . . . . . .
FZR1 . . . . . . . . . 1 . . . . . . . . . . . . . .
MFSD12 . . . . . . . . . . . . . 1 . . . . . . . . . .
HMG20B . . . . . . . . . . . . . 1 . . . . . . . . 1 .
TBXA2R . . . . . . . . . . . . . . . . . . . . . . . .
CACTIN . . . . . . . . . . . . . . . . . . . . . . . .
PIP5K1C . . . . . . . . . . . . . . . . . . . . . . . .
TJP3 . . . . . . . . . . . . . . . . . . . . . . . .
APBA3 . . . . . . . . . . . . . . . . . . . . . . . .
MRPL54 . . . . . . . 1 . 1 2 . 1 1 . . . . . . . . . .
MATK . . . . . . 1 1 . . . . . . . . . . 1 . . . . .
ZFR2 . . . . . . . . . . . . . . . . . . . . . . . .
DAPK3 . . . . . . . . . . . . . . . . . . . . . . . .
EEF2 1 6 1 1 1 1 1 4 1 . 3 1 1 2 . 1 2 2 1 2 5 1 . 2
PIAS4 1 . . . . . . . . . . . . . . . . . . . . . . .
AC016586.1 . . . . . . . . . . . . . . . . . . . . . . . .
ZBTB7A . . 1 1 . . . . 1 . . . . . . . . . . . . . . .
MAP2K2 . 2 1 . . . . . . . . . . . . . . . . . . . . .
SIRT6 . . . . . 1 . . . . . . . 1 . . . . . . . . . .
EBI3 . 1 . . . . . . . . . . . . . . . . . . 1 . 1 .
CCDC94 . . . . . . . . . . . . . 2 . . . . . . . . . .
TMIGD2 . . . . . . . . . . . . . . . . . . . . . . . .
FSD1 . . . . . . . . . . . . . . . . . . . . . . . .
STAP2 . . . . . . . . . . . . . . . . . . . . . . . .
MPND . . . . . . . . . . . . . . . . . . . . . . . .
SH3GL1 . . . . . . . . . . . . . . . . . . . . . . . .
CHAF1A . . . . . . . 1 . . . . . . . . . . . . . . . .
UBXN6 . 1 . . . . . . . . . 1 . 1 . . . . . . . . . .
HDGFRP2 . . . . . . . . . . . . . . . . . . . . . . . .
PLIN5 . . . . . . . . . . . . . . . . . . . . . . . .
LRG1 . . . . . . . . . . . . . . . . . . . . . . . .
SEMA6B . . . . . . . . . . . . . . . . . . . . . . . .
TNFAIP8L1 . . . . . . . . . . . . . . . . 1 . . . . . . .
C19orf10 1 1 . 1 1 . 1 . . . . . . 1 . . 1 1 . 1 . . 1 .
DPP9 . . . . . . . . . . . . . . . . . . . . . . . .
FEM1A . . . . . . . . . . . . . . . . . . . 1 . . . .
AC005523.2 . . . . . . . . . . . . . . . . . . . . . . . .
TICAM1 . . . . . . . . . . . . . . . . . . . 1 . . . .
CTC-518P12.6 . . . . . . . . . . . . . . . . . . . . . . . .
PLIN3 . . . . . . . 1 . . . . . . . . . . . . . . . .
ARRDC5 . . . . . . . . . . . . . . . . . . . . . . . .
KDM4B . . . . . . . . . . . . . . . . . . . . . . . .
SAFB2 . . . . . . . . . . 1 . . . . . . . . 1 . . 2 .
SAFB . . 1 . . . . . . . . . . . . . . . . 1 . . . .
RPL36 8 8 6 6 1 8 3 1 . 3 12 11 4 4 . 1 2 . 8 8 12 6 8 3
C19orf70 1 . . 2 . . . . . . . . . . . . . . . . . . 1 .
HSD11B1L . . . . . . . . . . . . . . . . . . . . . . . .
LONP1 . . . . . . . . . . . . . . . . . . . . . . . .
PRR22 . . . . . . . . . . . . . . . . . . . . . . . .
DUS3L . . . . . . 1 . . . . . . . . . . . . . . . . .
AC024592.12 . . . . . . . . . . . . . . . . . . . . . . . .
NDUFA11 1 1 . 1 . 1 . 1 . . . 1 . 2 . 5 . . 2 . 4 . . .
VMAC . . . . . . . . . . . . . . . . . . . . . . . .
AC104532.4 . . . . . . . . . . . . . . . . . . . . . . . .
CAPS . 1 . . . . . . . . . . . . . . . . 1 . . . . .
RANBP3 . . . . . . . . . . . . . . . . . . . . . . . .
RFX2 . . . . . . . . . . . . . . . . . . . . . . 1 .
CTC-503J8.6 . . . . . . . . . . . . . . . . . . . . . . . .
CLPP . . 1 . . . . . 1 . . 1 . . . . . . . . . . . .
ALKBH7 1 . 1 1 . 2 . . . . . 1 . . . . 1 . 1 1 . . . .
PSPN . . . . . . . . . . . . . . . . . . . . . . . .
GTF2F1 . . . . . . . . . . . 1 . . . . . . . . . . . .
KHSRP . . . . . . . . . . . . . . . . . . . . . . . .
SLC25A23 . . . . . . . . . . . . . . . . . . . . . . . .
CRB3 . . . . . . . . . . . . . . . . . . . . . . . .
DENND1C . . . . . . . . . . . . . . . . . . . 1 . . . .
TUBB4A . . . . . . . . . . . . . . . . . . . . . . . .
CD70 . . . . . . . . . . . . . . . . . . . . . . . .
TNFSF14 . . . . . . . . . . . . . . . . . . . . . . . .
C3 . . . . . . . . . . . . . . . . . . . . . . . .
GPR108 . . . . . . . . . . . . . . . . . . . . . . . .
TRIP10 . . . . . . . . . . . . . . . . . . . . . . . .
SH2D3A . . . . . . . . . . . . . . 1 . . . . . 1 . . .
VAV1 . . . . . . . . . . . . . . . . . . 1 . . . . .
EMR1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF557 . . . . . . . . . . . . . . . . . . . . . . . .
INSR . . . . . . . . . . . . . . . . . . . . . . . .
CTB-133G6.1 . . . . . . . . . . . . . . . . . . . . . . . .
CTB-133G6.2 . . . . . . . . . . . . . . . . . . . . . . . .
ARHGEF18 . . . . . . . . . . . . 1 . . . . . . . . . . .
PEX11G . . . . . . . . . . . . . . . . . . . . . . . .
ZNF358 . . . . . . . . . . . . . . . . . . . . . . . .
MCOLN1 . . . . . . . . . . . . . . . . . . 1 . . . . .
PNPLA6 . . . . . . . . . . . . . 1 . . . . . . . . . .
XAB2 . . . . . 1 . . . . . . . . . . . . . . . . . .
PET100 . . . . . . . . . . . 1 . . . . . . . . . 1 . .
CTD-3214H19.4 . . . . . . . . . . . . . . . . . . . . . . . .
PCP2 . . . . . . . . . . . . . . . . . . . . . . . .
STXBP2 . . . 1 . . . 1 1 1 . . . . . . . . 3 . . . . .
RETN . . . . . . . . . . . . . . . . . . 1 . . . . .
C19orf59 . . . . . . . . . . . . . 1 . . 1 . . . . . . .
TRAPPC5 . . . . . . . . . . . . . . . . . . . . . . . .
FCER2 . . . . . . . . . . . . . 1 . . . . . . . . . .
CLEC4G . . . . . . . . . . . . . . . . . . . . . . . .
EVI5L . . . . . . . . . . . . . . . . . . . . . . . .
MAP2K7 . . . . . . . . . . . . . . . . . . . . . . . .
TGFBR3L . . . . . . . . . . . . . . . . . . . . . . . .
SNAPC2 . 1 . . . . . . . . . . . . . . . . 1 . . . . .
TIMM44 . . . . . . . . . . . . . . . . . . . . . . . .
ELAVL1 . . . . . . . . . . . . . . . . . . . . 1 . . .
CTD-2325M2.1 . . . . . . . . . . . . . . . . . . . . . . . .
CERS4 . . . . . . . . . . . . . . . . . . . . . . . .
CD320 . . . . . . . . . . . . . . . . . . . . . . . .
NDUFA7.1 . . . . . . . . . . . 1 . . . . . . . . . . . 1
RPS28 4 12 5 5 . 5 4 4 1 2 13 9 4 7 1 3 5 1 8 9 13 2 3 4
KANK3 . . . . . . . . . . . . . . . . . . . . . . . .
RAB11B-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
RAB11B . . . . . . . . . . . 1 . . . . . . . . . . . .
MARCH2 . 1 . . . . . . . 1 . . . . . . . . 1 . . . . .
HNRNPM . . . 1 . . . 2 1 1 . 1 2 . . . . . 1 2 1 . 1 .
PRAM1 . . . . . . . . . . . . . 1 . . . . . . . . . .
ZNF414 . . . . . . . . . . . . . . . . . . . . . . . .
MYO1F 1 1 . . . 3 . 1 . . 1 . . . 1 . . . 1 1 . . . .
ADAMTS10 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF558 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF317 . 1 . . . . 1 . . . . . . . . . . . . . . . . .
ZNF699 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF559 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF266 . . . 1 . . . . . . . . . . . . . . . . . . . .
ZNF426 . . . . . . . . . . . . . . . 1 . . . . . . . .
ZNF121 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF561 . . 1 . . . . . . . . . . . . . . . . . . . . .
C19orf82 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF562 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF846 . . . . . . . . . . . . . . . . . . 1 . . . . .
FBXL12 . . . . . . . . . . . . . . . . . . . . . . . .
UBL5 1 . . 2 . . 1 . 1 1 . . 1 2 . . . 2 . 1 2 . . .
CTD-2623N2.11 . . . . . . . . . . . . . . . . . . . . . . . .
PIN1 . 1 2 . . . . . . 1 1 . 1 . . . . . . . . . 1 .
OLFM2 . . 1 . . . . . . . . . . . . . . . . . . . . .
C19orf66 1 . . . . . . . 1 . . . . . . . . 2 . 1 . . 1 .
ANGPTL6 . . . . . . . . . . . . . . . . . . . . . . . .
PPAN . . . . . . . . . . . . . . . . . . . . . . . .
P2RY11 . . 1 . . . . . . . . . . . . . . . . . . . . .
EIF3G 1 3 . 1 1 . 1 1 1 1 2 3 1 1 . . . . . 1 3 1 . 1
DNMT1 . . . . . . . . . . . . . . . . . . . . 1 . . .
CTD-2369P2.2 . . . . . . . . . . . . . . . . . . . . . . . .
MRPL4 . 1 . . . . . . 1 . 2 . 1 . . . . . . . . . . .
ICAM1 . . . . . . . . . . . . . . . . . . . . . . . .
ICAM4 . . . . . . . . . . . . . . . . . . . . . . . .
ZGLP1 . . . . . . . . . . . . . . . . . . . . . . . .
FDX1L . . . . . . . . . . . . . . . . . . . . . . . .
RAVER1 . . . . . . . . . . . . . . . . . . . . . . . .
ICAM3 . 1 . . . . . . . . . . 2 1 . 1 . . 2 . . . 1 .
TYK2 . . . . . . . . . . . . . . . . . . . . . . . .
CDC37 . . 2 . . . . . . . . . . . . . . . . . 1 . . .
PDE4A . . . . . . . . . . . . . . . . . . . . . . . .
KEAP1 . . . . . . . . . . . . . . . . . . 1 . . . . .
S1PR5 . . . . . . . 1 . . . . . . . . . . . . . . . .
ATG4D . 1 . . . . . . . . . . . . 1 . . . . . . . . .
KRI1 . . . . . . . . . . 1 . . . . . . . . . . . . .
CDKN2D . . . . . . . . . 1 . . . . . . . . . . . . . .
SLC44A2 . . . 1 . 1 . . . 1 1 . . . . . . . . . 1 . . .
ILF3-AS1 . . . . . . . . . . . 1 . . . . . 1 . 2 1 . . .
ILF3 2 . . . . . . . . . . . . . . . . . . 1 . . . .
QTRT1 . . . . . . . . . . . . . . . . . . 1 . . . . .
DNM2 . . . . . . . . . . . . . . . . . . . . 1 . . .
TMED1 . . . . . . . . . . . . . . . . . . . . . . . .
C19orf38 . . . 1 . . . . . 1 . . . 1 . . . . 1 . . . 1 .
CARM1 . 1 . . . . . . . . . . . . . . . . . . . . . .
YIPF2 . . . . . . . . . . . . . . . . . . . . . . . .
C19orf52 1 . . . . . . . . . . 1 . . . . . . . . . . . .
SMARCA4 . . . . . . . . . . . . . . . . . . . . . . . .
LDLR . . . . 1 . . . . . . . . . . . . . . . . . . .
RAB3D . . . . . . . . . . . . . . . . . . . . . . . .
TMEM205 1 . . . . . . . . . . . . 1 . . 2 . 2 . . . . .
CCDC159 . 1 . . . . . . . . . . . . . . . . . . . . . .
DKFZP761J1410 . . . 1 . . . . . . . . . . . . . . . . . . . .
SWSAP1 . . . . . . . . . . . . . . . . . . . . . . . .
EPOR . . . . . . . . . . . . . . . . . . . . . . . .
CCDC151 . . . . . . . . . . . . . . . . . . . . . . . .
PRKCSH . . 1 . . . . . . . . . . . . . . . . 1 . . . .
ZNF653 . . . . . . . . . . . . . . . . . . . . . . . .
ECSIT . . . . . . . . 1 . 1 . . 2 . . . . . . . . . .
ELOF1 . . . . . . . . . . . . . . . . . . 1 . . . 1 .
ZNF627 . . . . . . . . . . . . . . . . . . . . . . . .
ACP5 . . . . . . . . . . 1 . . . . . . . 1 . . . . .
ZNF823 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF441 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF440 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF439 . . . . . . . . . . . . . . . . . . . . . . 1 .
ZNF69 . . . . . . . . . . . . . . . . . . . . . 1 . .
ZNF700 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF763 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF433 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF844 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF625 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF136 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2666L21.1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF44 . . . . . . . . . . . . . . 1 . . . . . . . . .
ZNF563 . . . . . . . . . . . . . . . . . . . 1 . . . .
ZNF442 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF799 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF443 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF709 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF564 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF490 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF791 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2192J16.22 . . . . . . . . . . . . . . . . . . . . . . . .
MAN2B1 . . . . . . . . . . . . . . . . . . . . . . . .
WDR83 . . . . . . . . . . . . . . . . . . . . . . 1 .
WDR83OS . . 1 . . . 1 . . . . 1 . . . . . . 1 . . . 1 .
DHPS . . . . . . . . . . . . . 1 . . 1 . . . . . . .
FBXW9 . . . . . . . . . . . . . . . . . . . . . . . .
TNPO2 . . . . . . . . . . . . . . . . . . . . . . . .
C19orf43 3 1 1 2 1 1 1 . . . 3 1 2 2 2 . 2 . 3 . 2 . 1 .
ASNA1 . . 1 . . . . . . . . . 1 . . . . . 4 . . . . .
HOOK2 . . . . . . . . . . . . . . . . . . . . 1 . 1 .
CTD-2659N19.9 . . . . . . . . . . . . . . . . . . . . . . . .
JUNB 10 2 33 7 1 2 2 3 3 5 4 14 20 3 1 . 3 7 11 8 28 1 8 1
PRDX2 . . 1 1 . . . 1 . . . . . . 1 . . . . 1 . . . .
RNASEH2A . . . . . . . . . . 1 . . . . . . . . . . . . .
DNASE2 . 1 . . . . . . . . . . . . . . . . . . . . . .
GCDH . . . . . . . . . . 1 . 1 . . . . . . . . . . .
FARSA . . . . . . . . . . . . . . . . . . . . . . . .
CALR . . 1 1 . . 2 . 1 . 1 . 2 . 1 . . . . 2 . . . 1
CTC-425F1.4 . . . . . . . . . . . . . . . . . . . . . . . .
RAD23A . 1 . . . . . . . . . . . . . . 2 . . . . 1 . .
GADD45GIP1 . 1 . . . 1 . . 1 . . . . . . . . . 4 . 1 . 2 .
NFIX . . . . . . . . . . . . . . . . . . . . . . . .
LYL1 . . . 1 . . . . . . . . . . . . . . . . . . . .
TRMT1 . . . . . . . . . . . . . . . . . . . . . . . .
NACC1 . . . . . . . . . . . . . . . . . . . . . . . .
STX10 . . . . . . . . . . . . . . . . . . . . . . . 1
IER2 19 . 7 . 16 1 . 2 1 1 3 . 1 1 1 . 1 . 5 4 3 . 5 3
CTC-250I14.6 . . . . . . 1 . . 1 . . . . . . . . . . . . . .
CACNA1A . 1 . . . . . . . . . . . . . . . . . . . . . .
CCDC130 . 1 1 . . . . . . . . . . . . . . . . 1 . . . .
MRI1 . . . . . . . . . . . . . . . . . . . . . . . .
C19orf53 4 . . 2 . . . 1 . . . 1 . . . . . . . 2 2 . 1 1
ZSWIM4 . . . . . . . . . . . . . . . . . . . . . . . .
MIR24-2 . . 1 1 . . . . . 1 . . . 3 . . . . . . . . . .
CC2D1A . . 1 . . . . . . . . . . . . . . . . . . . . .
DCAF15 . . . . . . . . . . . . . . . . . . . . . . . .
RFX1 1 . . . . . 1 . . . . . . . . . . . . . . . . .
CTB-55O6.4 . . . . . . . . . . . . . . . . . . . . . . . .
IL27RA . . . . . . . . . . . . . . . . . . . . . . . .
hsa-mir-1199 . . . . . . . . . . . . . . . . . . . . . . . .
SAMD1 . . . . . . . . . . . . . . . . . . . . . . . .
PRKACA . . . . . . . . . . . . . . . . . . . . . . . .
ASF1B . . . . . . . . . . . . . . . . . . . . . . . .
CTB-55O6.12 . . . . . . . . . . . . . . . . . . . . . . . .
LPHN1 . . . . . . . . . . . . . . . . . . . . . . . .
CD97 . 1 . . . 1 . 1 . 2 . . . . . . . . . 1 1 . . .
DDX39A . . 5 . . 1 . . . . . . 1 . . . . . 1 . 1 . 1 .
PKN1 . . 1 . 1 . . . . . . . . . . . . . . . . . . .
GIPC1 . . . . . . . . . . . . . . . . . . . . . . . .
DNAJB1 . . 4 . . . . . . . . . 2 1 . . . 1 . 4 1 . . .
TECR . 1 1 . . . . . . . . . . . 1 . . . . . . . 1 .
NDUFB7 . . . 1 1 . 1 . . 1 1 . . 1 . . . . 2 . . . . 1
CLEC17A . . . . . . . . . . . . . . . . . . . . . . 2 .
EMR3 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF333 . . 1 . . . . . . . 1 . . . . . . . . . . . . .
EMR2 . . . . . . . . . . . . . . . . . . . . . . . .
ILVBL . . . . . . . . . . . . . . 1 . . . . . . . . .
EPHX3 . . . . . . . . . . . . . . . . . . . . . . . .
AC004257.3 . . . . . . . . . . . . . . . . . . . . . . . .
BRD4 . . . . . . . . . . . . . . . . . . . . . . . .
AKAP8 . . . . . . . . . . . . . . . . . . . . . . . .
AKAP8L . . . . . . . . . . 1 . . . . . . . . 1 . . . .
WIZ . . . . . . . . . . . . . . . . . . 1 . . . . .
RASAL3 . . . . . . 1 . . . 1 . . . . . . 1 1 . . . . 1
PGLYRP2 . . . 1 . . . . . . . . . . . . . . . . . . . .
CYP4F22 . . . . . . . . . . . . . . . . . . . . . . . .
TPM4 . . . 9 . . . . 1 . 1 . . . . . . . . . 1 . . .
RAB8A . 1 1 1 . . . . . . . . . . . . . . . . . . . 1
CTD-2231E14.8 . . . . . . . . . . . . . . . . . . . . . . . .
HSH2D . . . 1 2 . . . . . 1 . . . . . . . . . . . . .
FAM32A . . 1 . . . . . . . . . 1 1 . . . . . . . . . .
AP1M1 . . . . 1 . . . . . . . . 1 . . . . . . . . . .
CTD-2562J15.6 . . . . . . . . . . . . . . . . . . . . . . . .
KLF2 5 1 9 4 . . . . . 1 . . 1 . . . . . 3 2 4 1 4 1
EPS15L1 1 . . . . . . . . . . . . . . . . . . . . . . .
C19orf44 . . . . . . . . . . . . . . . . . . . . . . . .
CHERP . . . . . . . . . . . . . . . . . . . . 1 . . .
SLC35E1 . . . . . . . . . . . . . . . . . . . . . . . .
MED26 . . . . . . . . . . . . . . . . . . . . . . . .
SMIM7 . . . . . . . . . . 1 . . . . . . . 1 . 1 1 . .
TMEM38A . . . . . . . . . . . . . . . . . . . . . . . .
SIN3B . . . . . . . . . . . . . . . . . . . . . . . .
CPAMD8 . . . . . . . . . . . . . . . . . . . . . . . .
HAUS8 . . . . . . . . . . . . . . . . . . . . . . . .
MYO9B . . . 1 . . . 1 . . . . . . . . . . . . . . . .
USE1 . . . 1 1 . 1 . . . . . . . . . . . 1 . . . . .
OCEL1 . . . . . . . . . . 1 . . . . . . . . . . . . .
NR2F6 . . . . . . . . . . . . . . . . . . . . . . . .
BABAM1 . . . 1 . . . . . . . . . . . . . . . . . . . 2
ANKLE1 . . . . . . . . . . . . . . . . . . . . . . . .
ABHD8 . . . . . . 1 . . . . . . . . . . . . . . . . .
MRPL34 . . . . . . . . 1 . . . . . 1 . . . 1 . 1 . . .
DDA1 . . 1 1 . . . . . . . . 1 . . . 1 . . . . . . .
GTPBP3 . . . . 1 . . . . . . . . . . . . . . . . . 1 .
PLVAP . . . . . . . . . . . . . . . . . . . . . . . .
BST2 . 1 . 1 1 . . . . . 1 . 1 3 1 . . . 3 . . . . .
CTD-2521M24.9 . . . . . . . . . . . . . . . . . . . . . . . .
MVB12A . . 1 . . . . . . . . . . . . . . . . . . . . .
SLC27A1 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-3131K8.2 . . . . . . . . . . . . . . . . . . . . . . . .
PGLS . . . . . . . . 1 . 2 1 . 2 . . . . 3 . . 1 . .
FAM129C . . . . . . . . . . . . . . . . . . . . . . . .
COLGALT1 . . . . . . . . . . . . . . . . . . 1 . . . . .
MAP1S . . . . . 1 . . . . . . . . . . . . . . . . . .
FCHO1 . . . . . . . . . . . . . . . . . . . . 1 . . .
INSL3 . . . . . . . . . . . . . . . . . . . . . . . .
JAK3 . . . . . . . . . . . . . . 1 . . . . . . . . 1
RPL18A 18 40 8 16 4 5 11 14 5 3 45 15 15 21 9 7 6 10 23 23 45 12 28 15
CCDC124 . . . . 1 . . . . . . . . . . . . 1 . . . . . .
KCNN1 . . . . . . . . . . . . . . . . . . . . . . . .
ARRDC2 . . . 1 . . . . . . . . . . . . . . . . . . 1 1
IL12RB1 . . . . . . . . . . . . . . . . . . . . . . . .
MAST3 . . . . . . . . . . . . . . . . . . . . . . . .
PIK3R2 . . . . . . . . . . . . . . . . . . . . . . . .
IFI30 . . . . . . . . . . . . . . . . . . 4 . 1 . . .
MPV17L2 . . . . . . . . . . . . . . . . . . . . . 1 . .
RAB3A . . . . . . . . . . . . . . . . . . . . . . . .
PDE4C . . . 1 . . . . . . . . . . . . . . . . . . . .
KIAA1683 . . . . . . . . . . . . . . . . . . . . . . . .
JUND . . . . . . . . 1 . . . . . . 1 . . 2 . . . 2 .
LSM4 . 1 . . . . . . . . 2 . . . . . . . . . 1 . . .
PGPEP1 . . . . . . . . . . . 1 . . . . . . 1 . . . . .
LRRC25 . . . 1 . . . . . . . . . . . . . . 2 . . . . .
SSBP4 1 1 1 . . . . . . . . . 1 . . . . . . . 1 . . .
ISYNA1 . . . . . . . . . . . . . . . . . . 1 . . . . .
ELL . . . . . . . . . . 1 . . . . . . . . . . . . .
FKBP8 . . 2 1 1 . . . 1 . . . . . . . 1 . . . 1 . . .
KXD1 . . . . . . 1 . . . . . . 1 . . . . . . 1 . . .
AC005253.2 . . . . . . . . . . . . . . . . . . . . . . . .
AC005253.4 . . . . . . . . . . . . . . . . . . . . . . . .
UBA52 5 18 10 8 1 5 2 7 2 2 14 8 9 16 15 3 4 2 12 4 13 4 9 2
C19orf60 . . 1 1 . . . 1 . 1 . 1 . . . . . . 1 . . . . .
KLHL26 . . . . . . . . . . . . . . . . . . . . . . . .
CRTC1 . . . . . . . . . . . . . . . . . . . . . . . .
UPF1 . . . . . . . . . . . . . . . . . . . . . . . .
COPE 1 2 . 1 2 2 1 . . . 2 . . 1 1 . 2 . 3 1 2 . . 1
DDX49 . . 1 1 . . . . . 1 . . . . . . . 1 . . . . . .
HOMER3 . . . . . . . . . . . . . . . . . . . . . . . .
AC005932.1 . . . . . . . . . . . . . 1 . . . . . . . . . .
SUGP2 . 1 . 1 . . . . . . . . . . . . . . . . . . . .
AC004447.2 . . . . . . . . . . . . . . . . . . . . . . . .
ARMC6 . . . . . . . . . . . . . . . . . . . . . . . .
SLC25A42 . . . . . . . . . . . . . . . . . . . . . . . .
TMEM161A . . . . . . . . . . . . . . . . . . . . . . . .
MEF2BNB-MEF2B . . . . . . . . . . . . . . . . . . . . . . . .
MEF2BNB . . . . . . . . . . . . . . . . . . 1 . . . . .
RFXANK . 1 . . . . . . . . . 1 . . . . . . . . 1 . . .
NR2C2AP 1 . . . . . . . . . . . . . . . . . . . . . . .
SUGP1 . . . . 1 . . . . . . . . . . . . 1 . . . . . .
MAU2 . . . . . . . . . . . . . . . . . . . . . . . .
GATAD2A . . . . . . . . . . . . . . . . . . . . . . . .
TSSK6 . . . . . . . . . . . . . . . . . . . . . . . .
NDUFA13 1 . . 2 . . . . . 2 1 . 1 1 2 . . . 5 . 2 . 2 2
PBX4 . . . . . . . . . . . . . . . . . . . . . . . .
LPAR2 . . . . . . . . . . . . . . . . . . . . . . . .
GMIP . . . . . . . . . . . . . . . . . . 1 . . . . 1
ATP13A1 . . . . . . . . . . . . . . . . . . . 1 . . . .
ZNF101 . . . . . . . . . . . . . . . . . . . 1 . . . .
ZNF14 . . . . . . . . . . . . . . . . . . . . . . . .
LINC00663 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF506 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF253 . . . 1 . . . . . . . . . . . . . . . . . . . .
ZNF93 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF682 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF90 . . . . . . . . . . 1 . . . . . . . 1 . . . . .
CTC-260E6.6 . . . . . . . . . . . . . . . . . . . . . . 1 .
ZNF486 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF737 . . . . . 1 . . . . . . . . . . . . . . . . . .
ZNF626 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF85 . . . . . . . . . . . 1 . . . . . . . . . . . .
ZNF430 . . . . . . . . . . . . . . 1 . . . . . . . . .
ZNF714 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF431 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF708 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF738 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF493 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF429 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF100 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF43 . . . . . . . 1 . . . . . . . . . . . . . . . .
ZNF208 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF257 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF492 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF724P . . . . . . . . . . . . . . . . . . . . . . . .
RP11-15H20.7 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF91 . . . . . . . . . . . . . . . . . . . . 1 . . .
ZNF675 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF681 . . . . . . . . . . . . . . . . . . . . . . . .
RPSAP58 5 4 . 3 1 . . 2 2 . 2 3 2 . . . . 1 . . 6 1 3 1
ZNF726 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2017D11.1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF254 . . . . . . . . . . . . . . . . . . . . 1 . . .
LINC00662 . . . . . . . 1 . . . . . . . . . . . . . . . .
CTC-459F4.3 . . . . . . . . . . . . . . . . . . . . . . . .
UQCRFS1 . . . 1 . . . . . . . . 1 . . . . . 2 . . . . .
CTB-32O4.2 . . . . . . . . . . . . . . . . . . . . . . . .
POP4 . . . . . . . . . . . . . 1 . . . . . . . . . .
PLEKHF1 . . . . . . . . . . . . . . . . . . . . . . . .
C19orf12 . . . . . . . . . . . . . . . . . . . . . . . .
CCNE1 . . . . . . . . . . . . . . . . . . . . . . 1 .
URI1 . . . . . . . . . . . . . . 1 . . . . . . . . .
ZNF507 . . . . . . . . . . . . . . . . . . . . . . . .
DPY19L3 . . . . . . . . . . . . . . . . . . . . . . . .
PDCD5 . 1 1 . . 1 . . . . . . . . . . . 2 . . . . . .
ANKRD27 1 . . . . . . . . . . . . . . . . . . . . . . .
CTD-2538C1.2 . . . . . . . . . . . . . 1 . . . . . . . . . .
NUDT19 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2085J24.4 . . . . . . . . . . . . . . . . . . . . . . . .
CEP89 . . . . . . . . . . . . . . . . . . . . . 1 . .
C19orf40 . . . . . . . . . . . . . . . . . . . . . . . .
GPATCH1 . . . . . . . . . . . . . . . . . . . . . . . .
LRP3 . . . . . . . . . . . . . . . . . . . . . . . .
CEBPA . . . . . . . . . . . . . . . . . . . . . . . .
CEBPA-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
CEBPG . . . . . . . . . . . . . . . . . . . . . . . .
PEPD . . . . . . . . . . 1 . . . . . 1 . 1 . 1 . . .
KCTD15 . . . . . . . . . . . . . . . . . . . . . . . .
LSM14A 1 . . . . . . . . . . . . . 1 . . 1 . . 1 . 1 .
KIAA0355 . . . . . . . . . . . . . . . . . . . . . . . .
GPI . 1 . . . . . . . . . . . . . 1 . . . 1 . . . .
PDCD2L . . . . . . . . . . . . . . . . . . . . . . . .
UBA2 . . . . 1 . . . . . . . . . . . . . . . . . . .
SCGB1B2P . . . . . . . . . . . . . . . . . . . . . . . .
ZNF302 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF181 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF599 . . . . . . . . . . . . . . . . . . . . . . . .
CTC-523E23.1 . . . . . . . . . . . . . . . . . . . . . . . .
CTC-523E23.11 . . . . . . . . . . . . . . . . . . . 1 . . . .
ZNF30 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF792 . . . . . . . . . . . . . . . . . . . . . . . .
GRAMD1A . . . . . . 1 . . . . . 1 . . . . . . . . . . .
SCN1B . . . . . . . . . . . . . . . . . . . . . . . .
FXYD1 . . . . . . . . . . . . . . . . . . . 1 . . . .
FXYD7 . . . . . . . . . . . . . . . . . . . . 2 . . .
FXYD5 2 2 2 . 1 3 . 2 1 . 5 . 3 6 . . . 1 3 1 4 1 1 3
LSR . . . . . 1 . . . . . . . . . . . . . 1 . . . .
USF2 . . 1 . 1 . . . . . . . . . . . . . . . . 1 1 .
CD22 . . . . . . . . . . 1 . . 1 . . . . . . . . . .
FFAR3 . . . . . . . . . . . . . . . . . . . . . . . .
GPR42 . . . . . . . . . . . . . . . . . . . . . . . .
FFAR2 . . . . . . . . . . . . . . . . . . . . . . . .
DMKN . . . . . . . . . . . . . . . . . . . . . . . .
AD000090.2 . . . . . . . . . . . . . . . . . . . . . . . .
TMEM147 . 1 . 1 . . . . . . . . . . . . . . 2 . 1 . . .
HAUS5 . . . . . . . . . . . . . . . . . . . . . 1 . .
RBM42 1 . 1 . . . . . . . . . . . . . . . 1 . . . . .
ETV2 . . . . . . . . . . . . . . . . . . . . . . . .
COX6B1 1 2 1 1 1 . 1 . 1 1 1 . . 2 1 2 . 2 3 3 3 . 2 1
ZBTB32 . . . . . . . . . . . . . . . . . . . . . . . .
KMT2B . . . . . . . . . . . . 1 . . . . . . 1 . . . .
IGFLR1 . 1 1 . . . . . . . . . . . . . . . 1 . . . . .
AD000671.6 . . . . . . . . . . . . . . . . . . . . . . . .
U2AF1L4 . . 1 . . . . . . 1 . . . . . . . . . . . . . .
PSENEN . . . . . . 1 . . . . 1 . 1 . . . . . 1 . . . .
LIN37 . . . . . . . . . . . . . . . . . . . . . . . .
C19orf55 . . . . . . . . . . . . . . . . . . . . . 1 . .
ARHGAP33 . . . . . . . . . . . . . . . . . . . . . . . 1
NFKBID . 1 1 . . . . . . . . . . . . . . . . . . . 1 .
HCST . 1 4 . 2 1 1 1 1 . 1 1 1 1 2 . 2 1 1 . 2 . . .
TYROBP . . . 12 1 . . . . 1 . . . 8 . . 5 . 22 1 . . . .
LRFN3 . . . . . . . . . . . . . . . . . . . . . . . .
SDHAF1 . 1 . 1 . . . . . . . . . . . . . . . . . . . .
ALKBH6 . . . . . . . . . . . . . . . . . . . . . . . .
AC002116.7 . . . . . . . . . . . . . . . . . . . . . . . .
THAP8 . . . . . . . . . . . . . . . . . . . . . . . .
WDR62 . . . . . . . . . . . . . . . . . . . . . . . .
POLR2I . . . 7 . . . . . . . 1 . . . . . . 1 . . . . .
TBCB . 2 . 2 1 . . . 1 . . . . . 1 . . . . . . . . .
CAPNS1 . 1 1 1 . . . . . . . . . 1 . . . . . 1 1 . . .
ZNF565 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF146 . . . . . . . . . . . . . . . . . . . . . . . .
LINC00665 . 1 . . 2 . . . . . . . . . . . . . . . . . . 2
ZFP14 . . . . . 1 . . . . . . . . . . . . . . . . . .
ZFP82 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF566 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2630F21.1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF260 . . . . . . . . . . 1 . . . . . . . . . . . . .
ZNF529 . . . . . . . . . . . . . . . . . . . . . . . .
AC092295.7 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF382 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF461 . . . . . . . . . . . . . . . . . . . . . . . .
AC074138.3 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF567 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2162K18.4 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2162K18.5 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF790 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF345 . . . . 1 . . . . . . . . . . . . . . . . . . .
ZNF829 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF568 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF420 . . . . . . . . . . 1 . . . . . . . . . . . . .
CTC-454I21.3 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF585A . . . . . . . . . . . . . . . . . . . . . . . .
ZNF585B . . . . . . . . . . . . . . . . . . . . . . . .
ZNF383 . . . . . . . . . . . . . . . . . . . . . . . .
HKR1 . . . . . 1 . . . . . . . . . . . . . . . . . .
ZNF527 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF569 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF570 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2086O20.3 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-3064H18.1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF793 . . . . . . . 1 . . . . . . . . . . . . . . . .
ZNF571-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF540 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF571 . . . . . . . . . . . . . . . . . . . . . . . .
ZFP30 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF781 . . . . . . . . . . . . . . . . . . . 1 . . . .
ZNF573 . . . . . . . . . . . . . . . . . . . . . . . .
SIPA1L3 . . . . . . . . . . . . . . . . . . . . . . . .
SPINT2 . 1 . . . . . . 1 . . . . . . . . . 1 . 1 . . .
PPP1R14A . 7 . . . . . . . . . . . . . . . . . . . . . .
CTB-102L5.4 . . . . . . . . . . . . . . . . . . . . . . . .
C19orf33 . . . . . . . . . . . . . . . . . . . . . . . .
YIF1B . . . . . . . . . . . . . . . . . . . . . . 1 .
KCNK6 . . . . . . . . . . . . . . . . . . 1 . . . . .
AC026806.2 . . . . . . . . . . . . . . . . . . . . . . . .
CATSPERG . . . . . . . . . . . . . . . . . . . . . . . .
PSMD8 . 2 . 1 1 . 1 . . . . . 1 . . . . . . . . 1 1 2
FAM98C . . . . . . . . . . . . . . . . . . . . . . . .
RASGRP4 . . . . . . . . . . . . . . . . . . . . . . . .
RYR1 . . . . . . . . . . . . . . . . . . . . . . . .
MAP4K1 . 2 . . . . . . . . . . . . . . . . . . . . . .
EIF3K 2 4 1 5 1 . 1 . . . 3 2 . 3 . 2 3 . 2 1 3 . 1 .
ACTN4 . . . 1 . . 5 . . . . . . . . . . . . . . . . .
CAPN12 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2540F13.2 . . . . . . . . . . . . . . . . . . . . . . . .
LGALS4 . . . . . . . . . . . . . . . . . . . . . . . .
ECH1 . 1 2 . . . . . . . 1 . . . . . 1 . . . 1 . 1 .
HNRNPL . . . . 1 . . . . . . . . 1 . . . . 1 . . . 1 .
RINL . . . . . . . . . . . . . . . . . . . . . . . .
SIRT2 . . . . . . . . . . . . . . . . . . . . . . . .
NFKBIB . . . . . . . . . . . . 1 . . . . . . . . 1 . .
SARS2 . . . . . . . . . . . . . . . . . . 1 . . . . .
MRPS12 . . . . . . . . . . . . 1 . . . . . 1 1 . . 1 1
PAPL . . . . . . . . . . . . . . . . . . . . . . . .
PAK4 . . . . . . . . . . . . . . . . . . . . . . . .
IFNL1 . . . . . . . . . . . . . . . . . . . . . . . .
LRFN1 . . . . . . . . . . . . . . . . . . . . . . . .
GMFG . 2 4 1 . . . . . 2 2 3 1 13 . . 2 . 2 7 . . 4 .
CTC-246B18.10 . . . . . . . . . . . . . . . . . . . . . . . .
SAMD4B . . . . . . . . . . . . . . . . . . . . . . . .
PAF1 . . . . . . . . . . . . . . . . . . . . . . . .
MED29 . . . . . . 1 . . . . . . . . . . . . . . . . .
ZFP36 . 2 1 6 2 1 . . . . 1 1 8 8 . 1 . 1 7 4 1 . 3 .
PLEKHG2 . . . . . . . . . . . . . . . . . . . . . . . .
RPS16 5 32 13 4 2 7 4 6 1 1 22 5 12 10 4 4 4 4 18 11 13 8 17 3
SUPT5H . . . . . . . . . . . . . . . . . . . . . . . .
TIMM50 . . . . . . . . . . . . . . . . . . . 1 . . . .
DLL3 . . . . . . . . . . . . . . . . . . . . . . . .
EID2B . . . . . . . . . . . . . . . . . . . . . . . .
EID2 . . . . . . . . . . . . . . . . . . . . . . . .
DYRK1B . . . . 1 . . . . . . . . . . . . . 1 . . . . .
FBL 1 . . 1 . . 2 . 1 . 1 1 . 1 . . . . 1 . 2 . . .
FCGBP . . . . . . . . . . . . . . . . . . . . . . . .
PSMC4 . . 2 . . . . 1 . . . . 1 . . . . . . . . . . .
ZNF546 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF780B . . . . . . . . . . . . . . . . . . . . . . . .
ZNF780A . . . . . . . . . . . . . . . . . . . . . . . .
MAP3K10 . . . . . . . . . . . . . . . . . . . . . . . .
AKT2 . 1 . 1 . . . . . . . . . . . . . . . . . . . .
C19orf47 . . . . . . . . . . . . . . . . . . . . . . . .
PLD3 . . . . . . . . . . . . . . . . . . . . . . . .
SERTAD1 . . . . . . . . . . 2 . 1 . . . . . . . 2 . . .
SERTAD3 . . . . . . . . . . . . . . . . . . 1 . . . 1 .
BLVRB . . . 2 . . . . . . . . . . . . 2 1 3 . 1 . . .
SHKBP1 . 1 . . 1 1 . . . . . . . 2 . . . . . 1 . . . .
LTBP4 . . . . . . 1 . . . . . . . . . . . . . . . . .
NUMBL . . . . . . . . . . . . . . . . . . . . . . . .
ADCK4 . . . . . . . . . . . . . . . . . . . . . . . .
ITPKC . . . . . . . . . . . . . . . . . . . . . . . .
C19orf54 . . . . . . . . . . . . . . . . . . . . . . . .
SNRPA . . . . . . . 1 . . 1 . . . . . . . . . . . . .
RAB4B . . . 1 . . . . . . . . . . . . . . . . . . . .
EGLN2 . . 2 . . . . 1 . . . . . . . . . . . . . . . .
CYP2S1 . . . . . . . . . . . . . . . . . . . . . . . .
AXL . . . . . . . . . . . . . . . . . . . . . . . .
HNRNPUL1 . 1 . . 2 . . . . . . . . . . . . . . . . 1 . .
TGFB1 . 1 . . 1 . . . . 1 . . 1 . . . . . 1 1 1 . . .
CCDC97 . . . . . . . . . . . . . . . . . . . . 1 1 . .
TMEM91 . . . . . . . . . . . . . . . . . . . . . . . .
B9D2 . . . . . . . . . . . . . . . . . . . . . . . .
BCKDHA . . . . . . . . . . . . . 1 . . . . . . . . . .
EXOSC5 . . 1 . . . . . . . . . . . 1 . . . . . 1 . 1 .
B3GNT8 . . . . . . . . . . . . . . . . . . . . . . . .
ATP5SL . . . . . 1 . . . . . . . . . . . . 1 . . . . .
AC006129.1 . . . . . . . . . . . . . . . . . . . . . . . .
AC006129.4 . . . . . . . . . . . . . . . . . . . . . . . .
AC006129.2 . . . . . . . . . . . . . . . . . . . . . . . .
CEACAM21 . . . 1 . . . . . . 1 . . . . . . . . . . . . .
CEACAM4 . . . . . . . . . . . . . . . . . . 1 . . . . .
CEACAM3 . . . . . . . . . . . . . . . . . . . . . . . .
RPS19 22 45 18 13 1 13 13 30 9 10 26 19 19 15 5 4 6 5 49 19 24 9 36 4
CD79A . 3 . . . . . . . . 6 . . . . 1 . . . . 2 2 8 .
ARHGEF1 1 . . . . . . . . . 1 . 1 . . . . . . . . . . .
RABAC1 . 2 . . . 1 1 1 . . . . 1 . . . . . 1 . 1 . . .
ATP1A3 . . . . . . 1 . . . . . . . . . . . . . . . . .
ZNF574 . . . . . . . . . . . . . . . . . . . . . . . .
POU2F2 . 1 . 1 . . . . . . 1 . . . . . . . . 1 . . 2 .
CTC-378H22.1 . . . . . . . . . . . . . . . . . . . . . . 1 .
CTC-378H22.2 . . . . . . . 1 . . . . . . . . . . . . . . . .
DEDD2 . 1 . . . . . . . . . . 2 . . . . . 1 1 1 . . .
ZNF526 . . . . . . . . . . . . . . . . . . . . . . . .
GSK3A . . . 1 . . . 6 . . . . . . . . . . 1 . . . . .
ERF . . . . . . . . . . . . . . . . . . . . . . . .
CIC . . . . . . . . . . . . . . . . . . . . . . . .
PAFAH1B3 . 1 1 . . 2 . . . . . 1 . . . . . . . . . . . .
PRR19 . . . . . . . . . . . . . . . . . . . . . . . .
MEGF8 . . . . . . . . . . . . . . . . . . 1 . . . . .
CNFN . 1 . . . . . . . . . . . . . . . . . . . . . .
LIPE-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
LIPE . . . . . . . . . . . . . . . . . . . . . . . .
CEACAM1 . . . . . . . . . . . . . . . . . . . . . . . .
LYPD3 . . . . . . . . . . . . . . . . . . . . . . . .
PHLDB3 . . . . . . . . . . . . . . . . . . . . . . . .
ETHE1 . . 1 . . . 1 . . . . . . . . . . . . . . . . .
ZNF575 . . . . . . . . . . . . . . . . . . . . . . . .
XRCC1 . . . . . . . . . . . . . . . . . . . . . . . .
PINLYP . . . . . . . . . . . . . . . . . . . . . . . .
IRGQ . . . . . . . . . . . . . . . . 2 . . . . . . .
ZNF576 . . . . . . . . . . . . . . . . . . . . . . . .
SRRM5 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF428 . . . 1 . . . 1 1 . . . . 1 . . . . . . . . . .
PLAUR . . . . . . . . . . . . . . . . . . 1 . . . . .
SMG9 . . . . . . . . . . . . . . . . . . . . . . 1 .
KCNN4 . 1 . . . . . . . . . . . . . . . . . . 1 . . .
ZNF283 . . . . . . . . . . . . . 1 . . . . . . . . . .
ZNF404 . . . . . . . . 1 . . . . . . . . . . . . . . .
RP11-15A1.3 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF45 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF155 . . . . . . . . . . . . . . . . . . . . . . . .
RP11-15A1.7 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF230 . . . . . 1 . . . . . . . . . . . 1 . . . . . .
ZNF222 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF223.1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF284 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF224 . 1 . . . . . . . . . . . . . . . . . . . . . .
ZNF225 . . . . . . . . . . . . . . . . . . . . . . 1 .
ZNF234 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF226 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF227 . . . . . . . . . . . . . . . . . . 2 . . . . .
ZNF235 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF180 . . . . . . . . . . . . . . . . . . . . . . . .
PVR . . . . . . . . . . . . . . . . . . . . . . . .
BCL3 . 1 . . . . . . . . . . . . . . . . . . . . . .
PVRL2 . . . . . . . . . . . . . . . . . . . . . . . .
CTB-129P6.4 . . . . . . . . . . . . . . . . . . . . . . . .
TOMM40 . . 1 . . 11 . . 1 . . . . . . . . . . . . . . .
CLPTM1 . . . . . . . . . . . 1 . . . . . . . . . . . .
RELB . . . . . . . . . . 1 . . . . . . . . . . . . .
CLASRP . . . . . . . . . . . . . . . . . . . . 1 . . .
ZNF296 . . . . . . . . . . . . . . . . . . . . . . . .
GEMIN7 . . . . . . . . . . . . 1 . . . . . . . . . . .
MARK4 . . . . . . . . . . . . . . . . . . . . . . . .
PPP1R37 . . . . . . . . . . . . . . . . . . . . . . . .
TRAPPC6A . . 1 . . . . . . . . 1 . . . . 1 . 2 . . . . .
BLOC1S3 . . . . . . . . . . . . . . . . . . . . . . . .
CKM . . . . . . . . . . . . . . . . . . . . . . . .
ERCC2 . . . . . . . . . . . . . . . . . . . . . . . .
PPP1R13L . . . . . . . . . . . . . . . . . . . . . . . .
CD3EAP . . . . . . . . . . . . . . . . . . . . . . . .
ERCC1 . 1 . . . . . . . . . . . . . . . . . . 1 . . .
FOSB . 1 2 6 . 1 . . . 1 1 1 2 . . . . . 3 2 1 . . 1
PPM1N . . . . . . . . . . . . . . . . . . . . . . . .
VASP 1 . 2 . . 1 . . . . . . . . . . . . 1 1 2 . . .
OPA3 . . . . . . . . . . . . . . . . . . . . 1 . . .
EML2 1 . . . . . . . . . . . . . . . . . . 1 . . . .
C19orf83 . . . . . . . . . . . . . . . . . . . . . . . .
GIPR . . . . . . . . . . . . . . . . . . . . . . . .
SNRPD2 . 3 . 1 1 . 1 . . . . 1 2 2 1 . . 1 1 . 14 . . 1
FBXO46 . . . . . . . . . . . . . . . . . . . . . . . .
DMPK . . . . . . . . . . . . . . . . . . . . . . . .
DMWD . . . . . . . . . . . . . . . . . . . . . . . .
SYMPK . . . . . . . . . . . . . . . . . . . . . . . .
IRF2BP1 . . . . . . . . . . . . . . . . . . . . . . . .
MYPOP . . . . . . . . . . . . . . . . . . . . . . . .
CCDC61 . . . . . . . . . . . . . . . . . . . . . . . .
PPP5C . . . . . . . . . . . . . 1 . . . . . . . . . .
PPP5D1 . . . . . . . . . . . . . . . . . . . . . . . .
CALM3 . . 1 1 . . 1 1 . . 1 . . . . . . 2 2 . 3 . . .
CTB-12A17.3 . . . . . . . . . . . . . . . . . . . . . . . .
PTGIR . . . 1 . . . . . . . . . . . . . . . . . . . .
PRKD2 . 1 . . 1 . . . . . . . 1 . . . . . . . . . . .
STRN4 . . . . . 1 . . . . . . . . . . . . . . . . . .
FKRP . . . . . . . . . . . . . . . . . . . . . . . .
SLC1A5 . . 1 . . 1 . . . . . . . . . . . . . . . . . .
AP2S1 1 2 . 2 . 1 1 . . 4 . . . 1 . . 3 . 5 . 2 . . .
ARHGAP35 . . . . . . . . . . . . . . . . . . . . . . . .
TMEM160 . 1 . 1 . . . . . 1 . . . . 1 . . . . 27 . . 1 .
ZC3H4 . 1 . . . . . . . . . . . . . . . . . . . . . .
SAE1 . . . . . . . . . . . . . . . . . . . 1 1 . . .
BBC3 . . . . . . . . . . . . . . . . . . . . . . . .
CCDC9 . . . . . . . . . . . . . . . . . . . . . . . .
PRR24 . . . . . . . . . . . . . . . . . . 1 . . . . .
C5AR1 . . . 2 . . . . . 1 . . . . . . 1 . . . . . . .
C5AR2 . . . . . . . . . . . . . . . . . . . . . . . .
DHX34 . . . . . . . . . . . . . . . . . . . . . . . .
KPTN . 1 . . . . . . . . . . . . . . . . . . . . . .
NAPA-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
NAPA . . . . . . 1 . . 1 . . . . . . . . . . . . . .
ZNF541 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2571L23.8 . . . . . . . . . . . . . . . . . . . . . . . .
GLTSCR1 . . . . . . . . . . . . . . . . . . . . . . . .
GLTSCR2 2 3 5 3 3 1 2 2 3 . 4 3 5 2 . . 1 1 2 4 3 1 2 1
SEPW1 . 2 . . 1 1 . . . . . 1 1 1 . . . . . 1 . . . .
PLA2G4C . . . . . . . . . . . . . . . . . . . . . . . .
LIG1 . . . . . . . . . . . . . . . . . . . . . . . .
C19orf68 . . . . . . . . . . . . . . . . . . . . . . . .
CARD8 . . . . . . . . . 1 . . . . . . . . . . 4 . . .
CTC-241F20.3 . . . . . . . . . . . . . . . . . . . . 1 . . .
EMP3 1 4 3 5 . 1 1 1 . 5 1 . 1 1 . . 1 1 11 3 14 . 5 .
TMEM143 . . . . . . . . . . . . . . . . . . . . . . . .
KDELR1 . . . . . . . . . . . . . . . . . . . . . . . .
GRWD1 . . . . . . 2 . . . . . . . . . . . . . 1 . . .
KCNJ14 . . . . . . . . . . . . . . . . . . . . . . . .
CYTH2 . . 1 . 1 . . 1 . . . . . . . . . . . . . . 1 .
CTC-273B12.10 . . . . . . . . . . . . . . . . . . . . . . . .
RPL18 4 22 7 4 1 5 7 10 5 1 14 10 10 11 3 2 2 3 13 14 29 8 24 3
SPHK2 . . . . . . . . . . . . . . . . . . . . . . . .
DBP . . . . . . . . . . . . 1 . . . . . . . . . . .
CA11 . . . . . . . 1 . . . . . . . . . . . . . . . .
MAMSTR . . . . . . . . . . . . . . . . . . . . . . . .
RASIP1 . . . . . . . . . . . . . . . . . . . . . . . .
BCAT2 . . . . . . . . . . . . . . . . . . . . . . . .
HSD17B14 . . . . . . . . . . . . . . . . . . . . . . . .
PLEKHA4 . . . . . . . . . . . . . . . . . . . . . . . .
PPP1R15A 1 . 2 . . . . . . . 1 2 1 . . . . . 3 . . . 3 .
TULP2 . . . . . . . . . . . . . . . . . . . . . . . .
NUCB1 . . . 1 . 1 . . . . . . . . . . . . 1 1 . . . .
DHDH . . . . . . . . . . . . . . . . . . . . . . . .
BAX . 1 . . 1 . . . 2 1 . . . 1 . . 1 . 1 . 3 . . .
FTL 12 10 8 78 18 11 5 5 . 44 4 4 9 52 3 2 55 3 142 9 17 7 7 5
GYS1 . . . . . . . . . . . . . . . . . . . . . . . .
RUVBL2 . . . . . . . . . . . 1 1 . . . . . . 1 . . . .
SNRNP70 . . . 1 . . 1 1 . . . . . 1 . . . . . 1 . . 1 .
LIN7B . . . . . . . . . . . . . . . . . . . . . . . .
C19orf73 . . . . . . . . . . . . . . . . . . . . . . . .
TRPM4 . . . . . . . . . . . . . . . . . . . . . . . .
SLC6A16 . . . . . . . . . . . . . . . . . . . . . . . .
CTC-301O7.4 . . . . . . . . . . . . . . . . . . . . . . . .
CD37 2 7 2 3 . . 1 2 2 . 4 43 . 4 . 1 1 . 11 2 8 3 8 .
PIH1D1 . 1 . 1 . 1 . 1 . . 1 . . . . 1 . . . . . . 1 .
ALDH16A1 . . . . . . . . . . . . . . . . . . . . . . . .
FLT3LG 1 . . . . . . . . . . 1 . . . . . . . . 1 . . .
RPL13A 37 81 40 16 1 29 33 13 17 6 98 56 29 18 11 15 13 10 42 44 84 19 47 14
RPS11 1 16 3 6 . 3 8 . 1 . 16 3 6 10 2 . 2 . 17 10 11 3 14 4
FCGRT . 1 . . . . . . . 1 . . . 2 . . 1 . 1 . . . . .
RCN3 . . . . . . . . . . . . . . . . . . . . . . . .
NOSIP . 1 . . 1 . 3 . . . . 2 2 . . . 1 . . 1 . 1 2 .
PRRG2 . . . . . . . . . . . . . . . . . . . . . . . .
PRR12 . . . . . . . . . . . . . . . . . . . . . . . .
RRAS . . . . . . . . . . . . . . . . . . . . . . . .
SCAF1 . . . . . . . . . . 1 . . . . . . . . . . . . .
IRF3 . . . . . . . . . . . 1 . . . . . 1 . . . . . .
BCL2L12 . . . . . . . . . . . . . . . . . . . . . . . .
PRMT1 1 . 1 . . . . . . . 2 1 1 . . . . . . . . . 2 .
TSKS . . . . . . . . . . . . . . . . . . . . . . . .
AP2A1 . . . . . . . . . . . . . . . . . . . . . . . .
FUZ . . . . . . . . . . . . . . . . . . . . . . . .
MED25 . . . . . . . . . . . . . . . . . . . . . . 1 .
PTOV1-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
PTOV1 . . . . . 1 . . . . . . . 1 . . . . . . . . . .
AC018766.4 . . . . . . . . . . . . . . . . . . . . . . . .
PNKP . . . . . . . . . . 1 . 1 . . . . . . . . . . .
AKT1S1 . . . 1 . . . . . . . . . . . . . . . . . . . .
TBC1D17 . . . . . . . . . . . . . . . . . 1 . . . . . .
IL4I1 . . . . . . . . . . . . . . . . . . . . . . . .
NUP62 . . . . . . . . . . . . . . . . . . . . . . . .
ATF5 . . . 1 . . . . . . . . . . . . . . . . . . . .
VRK3 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF473 . . . . . . . . . . . . . . . . . . . . . . . .
KCNC3 . . . . . . . . . . . . . . . . . . . . . . . .
NR1H2 . . . . . . . . . . . . 1 . . . . . . . . 1 . .
NAPSA . . . . . . . . . . . . . . . . . . . . . . . .
POLD1 . . . . . . . . . . . . . . . . . . . 1 . . . .
SPIB . 1 . . . . . . . . . . . . . . . . . . . . 3 .
EMC10 . . 1 . . . . . . . . . . . . . . . . . . . . .
CTD-2545M3.2 . . . . . . . . . . . . . . . . . . . . . . . .
JOSD2 . 1 . . . . . . . . . . . . . . . . . . . . . .
CLEC11A . . . . . . . . . . . . . . . . . . 1 . . . . .
C19orf48 . . . . . . . . . . . . 1 . . . . . . . . . . .
KLK1 . . . . . . . . . . . . . . . . . . . . . . . .
CTU1 . . . . . . . . . . . . . . . . . . . . . . . .
SIGLEC9 . . . . . . . . . . . . . . . . . . . . . . . .
SIGLEC7 . . . . . . . . . . . . . . . . . . . . . . . .
CD33 . . . . . . . . . . . . . 2 . . . . 1 . . . . .
VSIG10L . . . . . . . . . . . . . . . . . . . . . . . .
ETFB . . . . 1 . 1 . . . 1 1 . 1 . . . . 1 . 1 . . .
CLDND2 . . . . . . . . . . . . . . . . . . . . . . . .
NKG7 . 1 . 1 11 . 5 2 1 . . . . . 3 . . 2 2 . 2 . . .
LIM2 . . . . . . . . . . . . . . . . . . . . . . . .
SIGLEC10 . . . . . . . . . . . . . . . . . . 2 . . . . .
CTD-2616J11.2 . . . . . . . . . . . . . . . . . . . . . . . .
SIGLEC12 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF175 . . . . . . . . . . . . . . . . . . . 1 . . . .
AC018755.1 . . . . . . . . . . . . . . . . . . . . . . . .
SIGLEC5 . . . . . . . . . . . . . . . . . . . . . . . .
SIGLEC14 . . . . . . . . . . . . . . . . . . . . . . . .
FPR1 . . . 1 . . . . . . . . . . . . . . 1 . . . . .
FPR2 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF577 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF649 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF613 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF350 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF615 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF614 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF432 . . . . . . 1 . . . . . . . . . . . . . . . . .
ZNF841 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF616 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF836 . . . . . . . . . . . . . . . . . . . . . . . .
PPP2R1A . 2 1 . . . . . . . 1 . . . . 1 . . . . . . 1 .
ZNF766 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF480 . . . . . . . . . . 2 . . . . . . . . . . . . .
ZNF880 . . . . . . . . . . . . . . . . . 1 . . . . . .
ZNF528 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF578 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF808 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF701 1 . . . . . . . . . . . . . . . . . . . . . . .
ZNF83 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF611 . . . . . . . . . . . . . . . . . . . . . . 1 .
ZNF600 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF28 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF468 . 1 . . . . . . . . . . . . . . . . . . . . . .
ZNF320 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF816 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF160 . . . . . . . . . . . . . . . . . . 1 . . . . .
ZNF415 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF347 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF665 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF677 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF845 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF525 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF765 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF813 . . . . . . . . . . . . . . . . . . . . 1 . . .
ZNF331 . . . . . . 1 . . . . . . . . . . . . . . . . .
NLRP12 . . . . . . . . . . . . . . . . . . . . . . . .
MYADM 1 . . . . . 1 . . . . . . . . . . . . . 1 . . .
AC008440.5 . . . . . . . . . . . . . . . . . . . . . . . .
VSTM1 . . . 1 . . . . . . . . . . . . . . 1 . . . . .
OSCAR . . 1 1 . . . . . . . . . 1 . . . . . . 1 . . .
NDUFA3 1 . . 1 . . . . . . . 1 . 1 . . . . . . . . . .
TFPT . . . . . . . . . . . . . . . . . . . 1 . . . .
PRPF31 . . . . . . . 1 . . . . . . . . . . . . . . 1 .
CNOT3 . . . . . . . . . . . . . . . . . . . . 1 . . .
LENG1 . 1 . . . . . . . . . . . 1 . . . . . . 1 . . .
TMC4 . . . . . . . . . . . . . . . . . . . . . . . .
MBOAT7 . . . . . . . . . 1 . . . . . . . . . . . . . .
TSEN34 . . . . . . . . . 1 . . . . . . . . . 1 . . . .
CTD-3093M3.1 . . . . . . . . . . . . . . . . . . . . . . . .
RPS9 7 14 9 9 3 2 7 4 3 5 13 8 17 11 3 1 5 3 26 6 23 10 18 7
LILRB3 . . . 2 . . . . . . . . . . . . . . . . . . . .
LILRA6 . . . . . . . . . . . . . . . . . . . . . . . .
LILRB2 . . . . . . . . . . . . . . . . 1 . . . . . . .
LILRA3 . . . . . . . . . . . . . . . . . . 1 . . . . .
LILRA5 . . . . . . . . . 1 . . . . . . . . 3 . . . . .
LILRA4 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2587H19.1 . . . . . . . . . . . . . . . . . . . . . . . .
LAIR1 . . . . . . . . . . . . . . . . . 1 1 . . . . .
LENG8-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
LENG8 . . . . . . . . . . . . . . . . . . . . . . . .
LENG9 . . . . . . . . . . . . . . . . . . . . . . . .
LAIR2 . . . . 1 . . . . . . . . . . . . . . . . . . .
LILRA2 . . . . . . . . . . . . . . . . . . . . . . . .
LILRB1 . . . . . . . . . . . . . . . . . . . . . . . .
LILRA1 . . . 1 . . . . . . . . . . . . . . 1 . . . . .
LILRB4 . . . . . . . . . . . . . . . . . . . . . . . .
KIR3DL1 . . . . . . . . . . . . . . . . . . . . . . . .
KIR2DL3 . . . . . . . . . . . . . . . . . . . . . . . .
KIR3DL2 . . . . . . 1 . . . . . . . . . . . . . . . . .
FCAR . . . . . . . . . . . . . . . . . . . . . . . .
CTB-61M7.2 . . . . . . . . . . . . . . . . . . . . . . . .
NCR1 . . . . . . . . . . . . . . . . . . . . . . . .
NLRP2 . . . . . . . . . . . . . . . . . . . . . . . .
CTC-550B14.7 . . . . . . . . . . . . . . . . . . . . . . . .
GP6 . . . . . . . . . . . . . . . . . . . . . . . .
RDH13 . . . . . . . . . . . . . . . . . . . . . . . .
EPS8L1 . . . . . . . . . . . . . . . . . . . . . . . .
PPP1R12C . 1 . . . . . . . . . . . . . . . . . . . . . .
TNNT1 . . . . . . . . . . . . . 1 . . . . . . . . . .
SYT5 . . . . . . . . . . . . . . . . . . . . 1 . . .
TMEM86B . . . . . . . . . . . . . . . . . . . . . . . .
PPP6R1 . . . . . . . . . . . . . . . . . . . . . . . .
HSPBP1 . . . . . . . . . . . . . . . . . . 1 . . . 1 .
BRSK1 . . . . . . . . . . . . . . . . . . . . . . . .
TMEM150B . . . . . . . . . . . . . 1 . . . . . . . . . .
SUV420H2 . . . . . . . . . . . . . . . . . . . . . . . .
COX6B2 . . . . . . . . . . . . . . . . . . . . . . . .
TMEM238 . . . . . . . . . . . . . . . . . . . . . . . .
RPL28 28 37 12 15 1 11 16 15 47 4 31 6 12 15 6 3 11 8 16 23 24 8 20 6
UBE2S . . . . . . . . . . 1 . . . . . . . . . . . . .
ISOC2 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF628 . . . . . . . . . . . . . . . . . . . . . . . .
NAT14 . . . . . . . . . . . . . . . . . . . . . . . .
SSC5D . . . . . . . . . . . . . . . . . . . . . . . .
ZNF579 . . . . . . . . . . . . . . . . . . . . . . . .
FIZ1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF524 . . . 1 . . . . . . . . . . . 1 . . . . . . . .
ZNF865 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF784 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF580 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF581 . 1 . . . . . . . . . 1 . . . . . . . . . . . .
CCDC106 . . . . . . . . . . . . . . . . . . . . . . . .
U2AF2 . . . . . . . . . . . . 1 1 . . . . . . . . . .
CTD-2537I9.12 . . . . . . . . . . . . . . . . . . . . . . . .
EPN1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF787 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF444 . . . . . . . . . . . . . . . . . . . . . . . .
ZSCAN5A . . . . . . . . . . . . . . . . . . . . . . . .
AC006116.20 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF582 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF582-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF583 . . . . . . . . . . . . 1 . . . . . . . . . . .
ZNF667 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF667-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
ZFP28 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF470 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF71 . 1 . . . . . . . . . . . . . . . . . . . . . .
AC007228.5 . . . . . . . . . . . . . . . . . . . . . . . .
AC007228.9 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF264 . . . . . . . . . . . . . . . . . . . . . . . .
AURKC . . . . . . . . . . . . . . . . . . . . . . . .
ZNF805 . . . . . . . . . . . . . . . . . . . . . . . .
CTC-444N24.6 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF460 . . . . . . . . . . . . . . . . . . . . . . . .
CTC-444N24.11 . . . . . 1 . . . . . . . . . . . . . . . . . .
ZNF543 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF304 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF547 . . . . . . . . . . . . . . . . . . . . . . . .
TRAPPC2P1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF548 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF17 . . . . . . . . . . . . 1 . . . . . . . . . . .
ZNF749 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF772 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF419 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF773 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF549 . . . . . . . . . . . . . . . . . . . . 1 . . .
ZNF550 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF416 . . . . . . . . . . . . . . . . . . . . . . . .
ZIK1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF530 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF134 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF211 . . . . . . . . . . . . . . . . . . . . 1 . . .
ZNF551 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF154 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF671 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF776 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF586 . . . . . . . . . . . . . . . . . . . . . 1 . .
ZNF552 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF587B . . . . . . . . . . . . . . . . . . . . . . . .
ZNF814 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF587 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF417 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF418 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF256 . . . . . . . . . . . . . . . . . . . . . . . .
C19orf18 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF606 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2368P22.1 . . . . . . . . . . . . . . . . . . 1 . . . . .
ZNF135 . . . . . . . . . . . . . . . . . . . . . . . .
ZSCAN18 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF329 . . . . . . . 1 . . . . . . . . . . . . . . . .
ZNF274 . . . . . . . . . . . . . 1 . . . . . . . . . .
ZNF544 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-3138B18.5 . . . . . . . . . . . . . . . . . . . . . . . .
AC010642.1 . . 1 . 1 . . . . . . . . . . . . . . . 1 . . .
ZSCAN22 . . . . . . . . . . . . . . . . . . . . . . . .
A1BG . . 1 . . . . . . . . . . . . 1 . . . . . . . .
A1BG-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF837 . . . . . . . . . . . . . . . . . . . . . . . .
RPS5 10 27 9 8 . 7 19 6 4 2 23 22 7 12 16 3 3 4 16 6 21 5 27 3
ZNF584 . . . . . . . . . . . . . . . . . . . . . . . .
CTD-2619J13.14 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF324B . . . . . . . . . . . . . . . . . . . . . . . .
ZNF324 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF446 . . . . . . . . . . . . . . . . . . . . . . . .
SLC27A5 . . . . . . . . . . . . . 1 . . . . . . . . 1 .
ZBTB45 . . . . . . . . . . . . . . . . . . . . . . . .
TRIM28 . . . . . . . . . . . . . . . . . . 1 . . . . .
CHMP2A . . 1 1 . . . . . . 1 . . . . . . . 4 2 1 1 . .
UBE2M . . . . . . . . . . . . . . . . . . 1 . . . . .
AC016629.8 . . . . . . . . . . . . . . . . . . . . . . . .
MZF1 . . . . . . . . . . . . . . . . . . . . . . . .
RPS4Y1 5 5 2 3 . 2 4 3 1 2 4 5 5 1 3 1 2 2 6 4 5 2 4 .
ZFY . . . . . . . . . . . . 1 . . . . . . . . . . .
TTTY15 . . . . . . . . . . . . . . . . . . . . . . . .
USP9Y . . . . . . . . . . . . . . . . . . . . . . . .
DDX3Y . . 1 . . . . . . . 1 . . . . . . . . . . . . .
UTY . . . . . . . . . . . . . . . . . . . . . . . .
TMSB4Y . . . . . . . . . . . . . . . . . . . . . . . .
NLGN4Y . . . . . . . . . . . . . . . . . . . . . . . .
TTTY14 . . . . 1 . . . . . . . . . . . . . . . . . . .
KDM5D . . . . . . . . . . 1 . . . . . . . . . . . . 1
EIF1AY . . . . 1 . . . . . . . . . . . . . . 1 . . 1 .
RPS4Y2 . . . . . . . . . . . . . . . . . . . . . . . .
TPTEP1 . . . . . . . . . . . . . . . . . . . . . . . .
IL17RA . . . . . . . . . . . . . . . . . . . . . . . .
CECR6 . . . . . . . . . . . . . . . . . . . . . . . .
CECR5 . . . . . . . . . . . . . . . . . . . . . . . .
CECR5-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
CECR1 . . . . . . . . . . . . . . . . . . . . . . . .
ATP6V1E1 . . . 1 . . . . . . 1 . . . . . . . . . . . 1 .
BCL2L13 . . . . . . . . . . . . . . . . . . . . . . . .
BID . . . . . . . . . 1 . . . 1 . . . . 1 . 1 . . .
LINC00528 . . . . . . . . . . . . . . . . . . 1 . . . . .
MICAL3 . . . . . . . . . . . . . . . . . . . . . . . .
XXbac-B476C20.9 . . . . . . . . . . . . . . . . . . . . . . . .
PEX26 . . . . . . . . . . . . . . . . . . . . . . . .
TUBA8 . . . . . . . . . . . . . . . . . . . . . . . .
USP18 . . . . . . . . . . . . . . . . . . . . . . . .
DGCR6 . . . . . . 1 . . . . . . . . . . . . . . . . .
DGCR2 . . . . . . . . . . . . . . . . . . . . . . . .
DGCR14 . . . . . . . . . . . . . . . . . . . . . . . .
AC004463.6 . . . . . . . . . . . . . . . . . . . . . . . .
SLC25A1 . . . . . . . . . . . . . . . . . . . . . . . .
CLTCL1 . . . . . . . . . . . . . . . . . . . . . . . .
HIRA . . . . . . 1 . . . . . . 1 . . . . . . . . . .
C22orf39 . . . . . . 1 . . . . . . . . . . . . . . . . .
MRPL40 . 1 . . . . . . . 1 . . . . . . . . . 1 1 . . .
UFD1L 1 1 . 2 . . . . . . . . 1 . 1 . . . . . 1 . . .
CDC45 . . . . . . . . . . . . . . . . . . . . . . . .
CLDN5 . . . . . . . . . . . . . . . . . . . . . . . .
SEPT5 . . . . . . . . . . . . . . . . . . . . . . . .
GNB1L 2 . . . . . . . 1 . . . . . . . . . . . . . . .
C22orf29 . . . . . . . . . . . . . . . . . . . . . . . .
TXNRD2 . . . . . . . . . . . . . . . . . . . . . . . .
COMT . . . . . . . . . . . . . . . . . . 1 . . . . .
ARVCF . . . . . . . . . . . . . . . . . . . . . . . .
TANGO2 . . . . . . . . . . . . . . . . . . . . . . . .
AC006547.15 . . . . . . . . . . . . . . . . . . . . . . . .
DGCR8 . . . . . . . . . . . . . . . . . . . . . . . .
TRMT2A . . . . . . . . . 1 . . . . . . . . . . . . . .
RANBP1 . . 1 . . . . . . . . 1 1 1 . . . 1 . 1 1 . . .
ZDHHC8 . . . . . . . . . . . . . . . . . . . . . . . .
RTN4R . . . . . . . . . . . . . . . . . . . . . . . .
DGCR6L . . 1 . . . . . . . . . . . . . . . . 1 . . . .
AC023490.1 . 1 . . . . . . . . . . . . . . . . . . . . . .
ZNF74 . . . . . . . . . . . . . . . . . . . . . . . .
SCARF2 . . . . . . . . . . . . . . . . . . . . . . . .
KLHL22 . . . . . . . . . . . . . . . . . . . . . . . .
MED15 . . . . . . 3 . . . . . . . . . . . . . 1 . . .
PI4KA . . . . . . . . . . . . . . . . . . . . . . . .
SNAP29 . . . . . . . . . . . . . 1 . . . . . . . . . .
CRKL . . . 1 . . . . . . . . . . . . . . . . . . . .
XXbac-B135H6.15 . . . . . . . . . . . . . . . . . . . . . . . .
AIFM3 . . . . . . . . . . . . . . . . . . . . . . . .
LZTR1 . . . . . . . . . . . . . . . . . . . . . . . .
XXbac-B135H6.18 . . . . . . . . . . . . . . . . . . . . . . . .
THAP7 . . . . . . . . . . . . . . . . . . . . 1 . . .
THAP7-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
HIC2 . . . . . . . . . . . . . . . . . . . . . . . .
TMEM191C . . . . . . . . . . . . . . . . . . . . . . . .
UBE2L3 . . 1 1 1 1 1 . . . . . 1 . . . . . 1 . 3 . . .
YDJC . . 1 . . . . . . . . . . . . . . . . . 1 . . .
SDF2L1 1 . . . 1 . . . . . . . . . . . . . 1 . . . . .
PPIL2 . . . . . . . . . . . . . 1 . . . . . . 1 . . .
YPEL1 . . . . . . . . . . . . . . . . . . . . . . . .
MAPK1 . . . . . . . . . . . . . 1 . . . . . . . . . .
PPM1F . 1 . . . . . . . . . . . . . . . . . . 1 . . .
LL22NC03-86G7.1 . . . . . . . . . . . . . . . . . . . . . . . .
TOP3B . . . . . . . . . . . . . . . . . . . . 1 . . .
VPREB1 . . . . . . . . . . . . . . . . . . . . . . . .
LL22NC03-2H8.5 . . . . . . . . . . . . . . . . . . . . . . . .
ZNF280B . . . . . . . . . . . . . . . . . . . . . . . .
IGLL5 . . . . . . . . . . . . . . . . . . . . . . . .
GNAZ . . . . . . . . . . . . . . . . . . . . . . . .
BCR . . . . . . . . . . . . . . . . . . . . . . . .
C22orf43 . . . . . . . . . . . . . . . . . . . . . . . .
RGL4 . 1 1 . . . 1 . . . . . 2 . 1 . . . . 1 . . . .
ZNF70 . . . . . . . . . . . . . . . . . . . . . . . .
VPREB3 . . . . . . . . . . 2 . . . . . . . . . . . 1 .
C22orf15 . . . . . . . . . . . . . . . . . . . . . . . .
CHCHD10 . 1 . 1 . . . . . . 2 . . . . . . . 1 . . . 1 .
MMP11 . . . . . . . . . . . . . . . . . . . . . . . .
SMARCB1 . 1 . . . . . 1 . . 2 . . . . . . . . 1 . . 2 .
DERL3 . . . . . . . . . . . . . . . . . . . . . . . .
SLC2A11 . . 1 . . . . . . . . . . . . . . . . . . . . .
MIF . 1 2 . . 1 1 1 . . 2 . . . . . . . . 1 . . 2 .
AP000350.4 . . . . . . . . . . . . . . . . . . . . . . . .
DDTL . . . . . . . . . . . . . . . . . . . . . . . .
DDT . . 1 1 1 . . . . . . . . 1 . . . . . . 1 . . .
GSTT1 . . . . . . . . . . . . . . . . . . . . . . . .
CABIN1 . 1 . . . . 2 . . . . . . . . . . . . . . . . .
SPECC1L . . . . . . . . . . . . . . . . . . . . . . . .
ADORA2A . . . . . . . . . . . . . . . . . . . . . . . .
ADORA2A-AS1 . . . . . . . . . 1 . . . . . . . . . . . . . .
GUCD1 . . . . . . . . . . . . . . . . . . . . 1 1 . .
SNRPD3 . . . . 1 . 1 . . 1 . . . . . . . . . . . . . .
GGT1 . . . . . . . . . . . . . . . . . . . . . . . .
SGSM1 . . . . . . . . . . . . . . . . . . . . . . . .
KIAA1671 . . . . . . . . . . . . . . . . . . . . . . . .
LRP5L . . . . . . . . . . . . . . . . . . . . . . . .
CTA-407F11.8 . . . . . . . . . . . . . . . . . . . . . . . .
ADRBK2 . . . . . . . . . . . . . . . . . . . . . . 1 .
SEZ6L . . . . . . . . . . . . . . . . . . . . . . . .
ASPHD2 . . . . . . . . . . . . . . . . . . . . . . . .
HPS4 . . . 2 . . . . . . . . . . . . 1 . . . . . . .
SRRD . . . . . . . . . . . . . . . . . . . . . . . .
TFIP11 . . . . . . . . . . . . . . . . . . . . . . . .
CTA-445C9.14 . . . . . . . . . . . . . . . . . . . . . . . .
CTA-445C9.15 . . . . . . . . . . . . . . . . . . . . . . . .
TPST2 1 . . . 1 1 . . . . 1 . . . . . 1 . 1 . . . . .
MIAT . . . . . 1 . . 1 . . . . . . . . . . . . . . .
CTA-373H7.7 . . . . . . . . . . . . . . . . . . . . . . . .
CTA-211A9.5 . . . . . . . . . . . . . . . . . . . . 1 . . .
PITPNB . . . . . . . . . . . . . . . . . . . . . . . .
TTC28 . . . . . . . . . . . . . . . . . . . . . . . .
CHEK2 . . . . . . . . . . . . . . . . . . . . . . . .
HSCB 1 . . . . . . . . . 1 . . . . . . . . . . . . .
CCDC117 . . . . . . . . . . . . . . . . . . . . . . . .
XBP1 . . . . . . 1 . . . . . 2 1 . . . . 1 . 3 . . .
CTA-292E10.6 . . . . . . . . . . . . . . . . . . . . . . . .
RHBDD3 . . . . . . . . . . . . . . . . . . . . . . . .
EWSR1 . . . . . . . . . . 1 . . . . . . 1 . . 2 . . .
GAS2L1 . . . . . . . . . . . . . . . . . . . . . . . .
AP1B1 . . . . . . . . . . . . . . . . . . . . . . . .
RFPL1S . . . . . . . . . . . . . . . . . . . . . . . .
NEFH . . . . . . . . . . . . . . . . . . . . . . . .
THOC5 . . . . . . . . . . . . . . . . . . . . . . . .
NIPSNAP1 . 2 . . . . 1 . . . . . . . . . . . . . . . . .
NF2 . . . . . . . . . . . . . . . . . . . . . . . .
CABP7 . . . . . . . . . . . . . . . . . . . . . . . .
ZMAT5 . . 1 . . . 1 . . . . . . . . . . . . . . . . .
UQCR10 . . . . . . 2 . . . 1 . 1 2 . . . . 3 1 1 . . .
ASCC2 . . . . . . . . . . . . . . . . . . . . . . . .
MTMR3 . . . . . . . . . . . . . . . . . . . . . . . .
OSM . . . . . . . . . . . . . . . . . . . . . . . .
GATSL3 . 1 . . . . . . . . . . . . . . . . . . . . . .
TBC1D10A . . . . . . . . . . . . . . . . . . . . . . . .
SF3A1 . . . . . . 1 . . . . . . . . . . . . . . . 1 .
CCDC157 . . . . . . . . . . . . . . . . . . . . . . . .
SEC14L2 . . . . . . . . . . . . . . . . . . . . . . . .
MTFP1 . . . . . . . 1 . . . . . . . . . . . 1 1 . . .
RP4-539M6.22 . . . . . . . . . . . . . . . . . . . . . . . .
PES1 . . . . . . . . . . . . . . . . . . 1 . . . . .
TCN2 . . . . . . . . . . . . . . . . . . . . . . . .
SLC35E4 . . . . . . . . . . . . . . . . . . . . . . . .
DUSP18 . . . 1 . . . . . . . . . . . . . . . . 1 . . .
MORC2-AS1 . . . . . . . . . . . . 1 . . . . . . . . . . .
MORC2 . . . . . . . . . . . . . . . . . . . . . . . .
TUG1 . . . . . . . . . . . . . . . . . . . . . . . .
SMTN . . . . . . . . . . . . . . . . . . . . . . . .
SELM . . . . . . . 1 . . . 1 . . . . . . . . 1 . . 1
RNF185 . . . . . . . . . . . . 1 . . . . . . . . . . .
LIMK2 . . . . . . . . . . . . . . . . . . . . . . . .
PIK3IP1 1 . . . . . . . . . . 1 . . . . . . . . . . . .
RP3-400N23.6 . . . . . . . . . . . . . . . . . . . . . . . .
PATZ1 . . . . . . . . . . . . . . . . . . . . . . . .
DRG1 . . . . 1 . . . . . 1 . . . . . . . . . . . . .
EIF4ENIF1 . . . . . . . . . . . . . . . . . . . . . . . .
SFI1 . . . . . . . . . . . . . . 1 . . . 1 . . . . .
PISD . . . . . . . . . . . . . . . . . . . . . . . .
PRR14L . . . . . . . . . . . . . . . . . . . . . . . .
DEPDC5 . . . . . . . . . . . . . . . . . . . . . . . .
YWHAH . 1 . 1 . . . . . 1 . . . 1 . . . . 1 . . . 1 .
RFPL2 . . . . . . . . . . . . . . . . . . . . . . . .
RTCB 1 . 1 . . . . . . . . . . 1 . . 1 . . . 1 . . .
FBXO7 . . . . . . . . . . . . . . . . . . . . . . 1 .
LARGE . . . . . . . . . . . . . . . . . . . . . . . .
HMGXB4 . . . . . . . . . . . . . . . . . . . . . . . .
RP3-510H16.3 . . . . . . . . . . . . . . . . 1 . . . . . . .
TOM1 . . . 1 . . . . . . . . . . . . . . . . . . . .
HMOX1 . . . . . 1 . . . . . . . 1 . . . 1 1 . . . . .
MCM5 . . . 1 . . . . . . . . . 1 . . . . . . 1 . . .
APOL6 . . . . . . . . . . . . . . . . . . . . . . . .
RBFOX2 . . . . . . . . . . . . . . . . . . . . . . . .
APOL3 . 1 . . . . 1 1 . . . . . 1 . . . . . . . . . .
APOL2 . . . . . . 1 . . . . . . . . . . . . . . . . .
APOL1 . . . . . . . . . . . . . . . . . . . . . . . .
MYH9 . . . 1 1 . . . 1 . 1 . . 1 . . . . . 1 2 . 3 .
TXN2 1 2 . 1 . . . . . . 1 . 1 . . . . . 1 . 1 . . .
FOXRED2 . . . . . . . . . . . . . . . . . . . . . . . .
EIF3D 1 . . . . 1 . . . . . . . 1 . . 1 . . . 3 . 2 .
IFT27 . . . . . . . . . . . . . . . . . . . . . . 1 .
PVALB . . . . . . . . . . . . . . . . . . . . . . . .
NCF4 . . . . . . . . . . . . . . . . 1 . . . . . 1 .
CSF2RB . . . . . . . . . . . . . . . . . . 1 . . . . .
TST . . . . . . . . . . . . . . . . . . . . . . . .
MPST . . . . . . . 1 . . . . . . . . . . . . . . 1 .
KCTD17 . . . . . . . . . . . . . . . . . . . . . . . .
IL2RB . . 1 . 2 . . . . . . . . . . . . 1 . . . . . .
C1QTNF6 . . . . . . . . . . . . . . . . . . . . . . . .
SSTR3 . . . . . . . . . . . . . . . . . . . . . . . .
RAC2 . 2 4 1 . 1 . . . 1 2 . . . . . . 1 1 . 6 1 2 1
CYTH4 . 1 . . . . . . . . 1 . . . . . . . . . . . . .
MFNG 1 . 2 . . . 1 . . . . 1 1 . 1 . . . . . 1 . 1 .
CDC42EP1 . . . . . . . . . . . . . . . . . . . . . . . .
LGALS2 . . . 2 . . . . . . . . . 6 . . 1 . 9 . . . . .
GGA1 . . . . . . . 1 1 . . . . . . . . . 1 . . . . .
SH3BP1 . . . 1 . . . . . 1 . 1 . 1 . . . . 1 1 . . . .
LGALS1 1 . . 4 2 1 . . 1 3 . . . 15 1 . 3 . 36 . 1 1 . .
NOL12 1 1 1 . . . . . . . . . . . . 1 . . . . . . . .
TRIOBP . . . . . . . . . . . . . . . . . . 7 . . . . .
H1F0 . . . . . . . . . . . . . . . . . . . . . . . .
GCAT . . . . . . . . . . . . . . . . . . . . . . . .
ANKRD54 . . . . . . . . . . . . . . . . . . . . . . . .
EIF3L 2 1 1 . 1 . . 1 1 1 2 2 1 . 1 . 3 . 2 1 1 1 2 1
MICALL1 . . . . . . . . . . . . . . . . . . . . . . . .
POLR2F . 1 . 1 . . . . . . 1 . 1 . . . . 1 1 . . . . .
PICK1 . . . . . . . . . . . . . . . . . . . . . . . .
PLA2G6 . . . . . . . . . . . . . . . . . . . . . . . .
MAFF . . . . . . . . . . . . . . . . . . . . . . . .
TMEM184B . . . . . . . . . . . . . . . . . . . . . . . .
CSNK1E . . . . . . . . . . . . . . . . . . . . . . . .
DDX17 2 . . . . . . 1 . . . . . . . . . . . . 1 . . .
DMC1 . . . . . . . . . . . . . . . . . . . . . . . .
CBY1 . . . . . . . . . . . . . . . . . . . . . . . .
TOMM22 1 . 1 . . . . 1 . 1 . 1 . 3 . 1 . . 1 . . . . 1
JOSD1 . . . 1 . . . . . . . . . . . . . . . . . . . .
GTPBP1 . . 1 . . . . . . . . . . . . . . . . . . . . .
SUN2 . . 2 . . . . . . . . . 1 . . . . . . 1 . . . .
RP3-508I15.19 . . . . . . . . . . . . . . . . . . . . . . . .
RP3-508I15.21 . . . . . . . . . . . . . . . . . . . . . . . .
RP3-508I15.22 . . . . . . . . . . . . . . . . . . . . . . . .
DNAL4 . . . . . . . . . . . . . . . . . . . . . . . .
NPTXR . . . . . . . . . . . . . . . . . . . . . . . .
CBX6 . . . . 1 . . . . . . . 1 . . . . . 1 . . . . .
APOBEC3A . . . . . . . . . . . . . 2 . . . . 1 . . . . .
APOBEC3B . . . . . . . . . . . . . . . . . . 5 . . . . .
APOBEC3C . . . . . . . . . . . . . . . . . . . . . . . .
APOBEC3D . . . . . . . . 1 . . . . . . . . . . . . . . .
APOBEC3F . . . . . . . . . . . . . . . . . . . . . . . .
APOBEC3G 1 . . . . . 1 1 1 . . . . . . . . . 1 1 . . . .
APOBEC3H . . . . . . . . 1 . . . . . . . . . . . . . . .
CBX7 . . . 1 . . . . . . 1 . . . . . . . . . . . . .
PDGFB . . . . . . . . . . . . . . . . . . . . . . . .
RPL3 25 24 13 11 3 13 74 12 10 4 43 33 25 33 4 5 6 7 17 26 36 14 23 11
SYNGR1 . . . . . . . . . . . . . . . . . . 1 . . . . .
TAB1 . 1 . . . . . . . . . . . . . . . . . . . . . .
MIEF1 . . . . . . . . . . . . . . . . . . . . 1 . . .
ATF4 . 1 1 . . 1 . . . . . . . . . . . 1 1 1 1 . . .
RPS19BP1 . 1 1 . . . 1 . . . . . . . . . . . 1 . 1 . 1 .
CACNA1I . . . . . . . . . . . . . . . . . . . . . 1 . .
GRAP2 . . . . . . . . . . . . . . . . . . . . . . . .
TNRC6B . . . . . . . . . . . . . . . . . . . . . . . .
ADSL . . . . . . . . . . . . . . . . . . . . . . 39 .
SGSM3 . . 1 . . . . . . . . . . . . . . . . . . . . .
RP5-1042K10.10 . . . . . . . . . . . . . . . . . . . . . . . .
MKL1 . . . . . . . . . . . . . . . . . . . . . . . .
SLC25A17 . . . . . . . . . . . . . . . . . . . . . . . .
ST13 1 4 1 . . 1 1 1 . 1 2 1 1 1 . . 1 . 1 . 1 . 1 .
XPNPEP3 . . . . . . . . . . . . . . . . . . . . . . . .
RBX1 . . . 1 . . . . . 1 1 . 1 . . . 1 . 3 . 1 . . .
EP300 . . . . . . . . 1 . . . . . . . . . . . . . . .
L3MBTL2 . . . . . . 1 1 . . 1 . . . . . . . . . . . . .
RANGAP1 . . . . . . . . . . . . . . . . . . . . . . . .
ZC3H7B . . . . . . . . . . . . . . . . . . . . . . . .
TEF . . . . . . . . . . . . . . . . . . . . . . . .
TOB2 . . . . . . . . . . . . . . . . . . . . . . . .
PHF5A . 1 1 . . . 1 . . . . . . . . . . . . 1 2 . . .
ACO2 . . . . . . . . . . . . . . . . . . . . . . . .
POLR3H . . . . . . . . . . . . . . . . . . . . . . . .
PMM1 . . . . . . 1 . . 1 . . . . . . . . . . . . . .
DESI1 . . . 1 . . . . . . 1 . . . 1 . 1 . . . 1 . . .
XRCC6 . . . . . . . . . . . . . 1 . 1 . . 1 . . . 1 .
NHP2L1 1 1 . . . 1 . . . 2 3 . . 1 . . . 1 . 1 3 2 1 1
C22orf46 . . . . . . . . . . . . . . . . . 1 . . . . . .
MEI1 . 1 . . . . . . . . 1 . . . . . . . 1 . . . . .
CCDC134 . . . . . . . . . . . . . . . . . . 1 1 . . . .
RP5-821D11.7 . . . . . . . . . . . . . . . . . . . . . . . .
SREBF2 . . . . . . . . . . . . . . . . . . . . . . . .
CTA-250D10.23 . . . . . . . . . . . . . . . . . . . . . . . .
TNFRSF13C . . . . . . . . . . . . . . . . . . . . . . . .
CENPM . . . . . . . . . . . . . . . . . . . . . . . .
WBP2NL . . . . . . . . . . . . . . . . . . . . . . . .
NAGA . . . . . . . . . . . . . . . . . . 1 . . . . .
FAM109B . . . . . . . . . . . . . . . . . . . . . . . .
SMDT1 . . 2 2 . 3 . 1 1 . 1 1 . . . . . . 2 . 1 . . .
NDUFA6 . . . 1 . . . . . . . . . . . . . . . . . . 2 .
CYP2D6 . . . . . . . . . . . . . . . . . . . . . . . .
TCF20 . . . . . . . . . . . . . 1 . . . . . . . . . .
NFAM1 . . . . . . . . . . . . . . . . . . . . . . . .
RRP7A 1 . . . . . . . . . . . . . . . . . . . 1 . . .
SERHL2 . . . . . . . . . . . . . . . . . . . . . . . .
POLDIP3 . . . . . . . . . . . . 1 . . . . . . . . . 1 .
RNU12 . . . . . . . . . . . . . . . . . . . . . . . .
CYB5R3 . . . . . . . . . . . . . . . . . . . . . . 1 .
ATP5L2 . . . . . . . . . . . . . . . . . . . . . . . .
ARFGAP3 . . . . . 1 . . . . . . . . . . . . . . . . . 1
PACSIN2 . . . . . . . . . . . . . 1 . . . . . . 1 . . .
TTLL1 . . . . . . . . . . . . . . . . . . . . . . . .
BIK . . . . . . . . . . . . . . . . . . . . . . . .
MCAT . . . . . . . . . . 1 . . . . . . . . . . . . .
TSPO 1 1 . . . . . 1 . . . . . 1 . . 4 . 3 1 1 1 2 1
TTLL12 . . . . . . . . . . . . . . . . . . . . 1 . . .
SAMM50 . . . . . . . . . . 1 . . . . . . . . . . . 1 .
PARVB . . . . . . . . . . . . . . . . . . . . . . . .
PARVG . . . 1 . . 1 . . . . . . . . . . . . 1 . . . .
LDOC1L . . . . . . . . . . . . . . . . . . . . . . . .
PRR5 1 . 1 . . . . . . . . . . . . . . . . . . . . .
CTA-217C2.1 . . . . . . . . . . . . . . . . . . . . . . . .
NUP50 . . . . . . . . . . . . . . . . . . . 1 . . . .
KIAA0930 . . . . . . . . . . . . . . . . . . . . . . . .
UPK3A . . . . . . . . . . . . . . . . . . . . . . . .
FAM118A . . . . . . . . . . . . . . . . . . . . . . . .
ATXN10 . 1 . . . . 1 . . . . . . . . . . . . 2 . . . .
CITF22-92A6.1 . . . . . . . . . . . . . . . . . 1 . . . . . .
C22orf26 . . . . . . . . . . . . . . . . . . . . . . . .
RP6-109B7.3 . . . . . . . . . . . . . . . . . . . . . . . .
FLJ27365 . . . . . . . . . . . . . . . . . . . . . . . .
PPARA . . . . . . . . . . 1 . . . . . . . . . . . . .
CDPF1 . . . . . . . 1 . . . . . . . . . . . . . . . .
TTC38 . . . . . . . . . . . . . . . . . . . . . . . .
GTSE1 . . . . . . . . . . . . . . . . . . . . . . . .
TRMU . . . . . . . . . . . 1 . . . . . . . . . . . .
CELSR1 . . . . . . . . . . . . . . . . . . . . . . . .
GRAMD4 . . . . . . . . . . . . . . . . . . . . . . . .
CERK . . . . . 1 . . . . . . . . . . . . . . . . . .
CTA-29F11.1 . . . . . . . . . . . . . 1 . . 1 . . . . . . .
TBC1D22A . 1 . 1 . . . . . . 1 . 1 . . . . . . . 1 . . .
LL22NC03-75H12.2 . . . . . . . . . . . . . . . . . . . . . . . .
C22orf34 . . . . . . . . . . . . . . . . . . . . . . . .
RP1-29C18.10 . . . . . . . . . . . . . . . . . . . . . . . .
BRD1 . . . . . . . . . . . . . . . . . . . . . . . .
ZBED4 . . . . . . . . . . . . . . . . . . . . . . . .
ALG12 . . . . . . . . . . . . . . . . . . . . . . . .
CRELD2 . . 1 . . . . . . . . . . . . . . . . . . . . .
PIM3 . . . . . . . . . . . . 1 . . . . . . . . . . .
MLC1 . . . . . . . . . . . . . . . . . . . . . . . .
MOV10L1 . . . . . . . . . . . . . . . . . . . . . . . .
TRABD . . 1 1 . . . . . . . . . . . . . . 1 . 2 . . .
RP3-402G11.25 . . . . . . . . . . . . . . . . . . . . . . . .
RP3-402G11.26 . . . . . . . . . . . . . . . . . . . . . . . .
SELO . . . . . . . . . . . . . . . . . . . . . . . .
TUBGCP6 . . . . . . . . . . . . . . . . . . . . . . . .
HDAC10 . . . 1 . 1 . . . . 1 . . . . . . . . . . . . .
MAPK11 . . . . . . . . . . . . . . . . . . . . . . . .
PLXNB2 . . . 1 . . . . . . . . . . . . . . . . . . . .
DENND6B . . . . . . . . . . . . . . . . . . 2 . . . . .
PPP6R2 . . . . . . . . . . . . . . . . . . . . . . . .
SBF1 1 . . . . . 1 . . . . . . . . . . . . . . . . .
LMF2 . . . . . . . . . . . . . . 1 . . . . . . . . .
NCAPH2 . . . . . . . . . . . . . . . . . . . . . . . .
SCO2 . . . 1 . . . . . . . . 1 1 . . . . 2 . . . . .
CTA-384D8.36 . . . . . . . . . . . . . . . . . . . . . . . .
TYMP . . . 3 . . 1 . . 3 1 1 . 4 . . 2 . 8 . . . . .
ODF3B . . . 1 . . . . . . . . . 1 . . . . . . . . . .
CTA-384D8.35 . . . . . . . . . . . . . . . . . . . . . . . .
CTA-384D8.34 . . . . . . . . . . . . . . . . . . . . . . . .
KLHDC7B . . . . . . . . . . . . . . . . . . . . . . . .
SYCE3 . . . . . . . . . . . . . . . . . . . . . . . .
CPT1B . . . . . . . . . . . . . . . . . . . . . . . .
CHKB . . . . . . . . . . . . . . . . . . . . . . . .
CHKB-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
ARSA . . . . . . . . . . . . . . . . . . . . . . . .
RABL2B . . . . . . . . . . . . . . . . . . . . . . . .
RBM11 . . . . . . . . . . . . . . . . . . . . . . . .
HSPA13 . . . . . . . . . . . . . . . . . . . . . . . .
SAMSN1 . . . . . 1 . . . . . . . . . . . . 1 . 1 . . .
AF127936.7 . . . . . . . . . . . . . . . . . . . . . . . .
NRIP1 . . . . . . . . . . . . . . . . . . . . . . . .
USP25 . . . . . . . . . . . . 1 . . . . . . . . . . .
BTG3 . . . . . . . . . . . . . . . . . 1 . 1 . . . .
C21orf91 . . . . . . . . . . . . . . . . . . . . . . . .
AP000476.1 . . . . . . . . . . . . . . . . . . . . . . . .
MIR155HG . . . . . . . . . . . . . . . . . . . . . . . .
MRPL39 . . . . . . . . . . . . . 1 . . . . . . . . . .
ATP5J . . 1 . . . . 1 . . 1 1 1 . . . . 1 2 . 3 . . .
GABPA 1 . . . . . . . . . . . . . . . . . . . . . . .
APP . . . . . . . . . . . . . . . . . . . . . . . .
AF131217.1 . . . . . . . . . . . . . . . . . . . . . . . .
N6AMT1 . . . . . . . . . . 1 . . . . . . . . . . . . .
LTN1 1 . . . . 1 . . . . . . . . . . . . . . . . 1 .
RWDD2B . . . . . . . . . . . . . . . . . . . . . . . .
RP1-100J12.1 . . . . . . . . . . . . . . . . . . . . . . . .
USP16 . . . . . . 1 1 . . . . . . . . . . . . . . . .
CCT8 . 1 2 . . 1 . . . . 2 . 1 . . . . . . . . . . .
MAP3K7CL . . . . . . . . . . . . . . . . . 1 . . . . . .
BACH1 . . . . . 1 . . . . . . . . . . . . . . . . . .
TIAM1 . . . . . . . . . . . . . . . . . . . . . . . .
SOD1 1 1 1 1 . . . 2 . . . 4 2 1 . . . . . . 1 . 1 1
AP000254.8 . . . . . . . . . . . . . . . . . . . . . . . .
SCAF4 . . . . . . . . . . . . . . . . . . . . . . . .
MIS18A . . . . . . . . . . . . . . . . . . . . . . 1 .
URB1 . . . . . . . . . . . . . . . . . . . . . . . .
C21orf119 . . . . . . . . . . . . . . . . . . . . . . . .
EVA1C . . . . . . 1 . . . . . . . . . . . . . . . . .
TCP10L . . 1 . . . . . . . . . . . . . . . . . . . . .
C21orf59 . . . 1 . . 1 . . . . . . . 1 . . . . . . . . .
SYNJ1 . . . . . . . . . . . . . . . . . . . . . . . .
PAXBP1-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
PAXBP1 . . . . . . . . . . . . . . . . . . . . . . . .
C21orf49 . . . . . . . . . . . . . . . . . . . . . . . .
OLIG1 . . . . . . . . . 1 . . . . . . . . . . . . . .
IFNAR2 . . . . . . . 2 . . . . . . . . . . . . . . . .
IL10RB-AS1 . . . . . . . . . . . . . . . . . . . . . . . .
IL10RB . . . 1 . . . 1 . . . . . . . . . . . . . . . .
IFNAR1 . . . . 1 . . . . . . . . . . . . . . . . . . .
IFNGR2 . . . 2 . . 1 . . . . . . . . . . . . . . . . .
TMEM50B . . . . . . . . . . . . . . . . . . . . 1 . . .
DNAJC28 . . . . . . . . . . . . . . . . . . . . . . . .
GART . . . . . . . . . . . . . . . . . . . . . . . .
SON . . . 2 . . . . . . 1 . . 1 . . . . 1 1 2 . . .
DONSON . . . . . . . . . . . . . . . . . . . . . . . .
CRYZL1 . . . . . . . . . . . . . . . . . . . . . 1 . .
ITSN1 . . 1 . . . . . . . . . . . . . . . . . . . . .
ATP5O . 1 . 1 . 35 . . . . 3 2 . . . . . . 1 1 . . . .
LINC00649 . 1 . . . . . . . . . . . . . . . 1 . . . . . .
MRPS6 . . 1 . . . . . . . 1 . . . . . . . . . 1 . . .
SMIM11 . . . . . . . . . . . 2 . . . . . . . . . . . .
KCNE1 . . . . . . . . . . . . . . . . . . . . . . . .
RCAN1 . . . . . . . . . . . . . . . . . . . . . . . .
RUNX1 . . . 1 . . . . . . . . . . . . . . . . . . . .
SETD4 . . . . . . . . . . . . . . . . . . . . . . . .
CBR1 . . . . . . . . . . . . 1 . . . . . 1 . . . . .
CBR3 . . . . . . . . . . . . . . . . . . . . . . . .
DOPEY2 . 1 . . . . . . . . 1 . . . . . . . . . . . . .
AP000692.10 . . . . . . . . . . . . . . . . . . . . . . . .
MORC3 . . . . . . . . . . . . . . . . . . . . . . . .
CHAF1B . . . . . . . . . . . . . . . . . . . . . . . .
HLCS . . . . . . . . . . . . . . . . . . . . . . . .
PIGP . . . . . . . . . . . . . . 1 . . . . . . . . .
TTC3 1 . . . . . . . . . . . . 1 . . . . . . . . 1 .
DSCR9 . . . . . . . . . . . . . . . . . . . . . . . .
DSCR3 . . . . . . . . . . . 1 . . . . 1 . . . 1 . . .
AP001412.1 . . . . . . . . . . . . . . . . . . . . . . . .
DYRK1A . . . 1 . . . . . . . . . . . . . . . . . . . .
ETS2 . . . . . . . . . . . . . . . . . . . . . . . .
PSMG1 . . . . . . . . . . 1 . . . . . . . . 1 . . . .
BRWD1 . 1 . . . . . . . . . . . . . . . . . . . . . .
HMGN1 1 . 1 1 . . . . 1 . 1 3 2 . . . . . . 1 2 2 2 1
WRB . 1 . . . . . . . . . . . . . . . . . 1 . . . .
BACE2 . . . . . . . . . . . . . . . . . . . . . . . .
FAM3B . . . . . . . . . . . . . . . . . . . . . . . .
MX2 . . . . . . . . . . 1 . . . . . . . . . . . . .
MX1 . . . 1 . . . . . . 1 . 1 . . . . . . . 1 . 1 .
AP001610.5 . . . . . . . . . . . . . . . . . . . . . . . .
PRDM15 . . . . . . . . . . . . . . . . . . . . . . . .
C2CD2 . . . . . . . . . . . . . . . . . 1 . . . . . .
ZBTB21 . . . . . . . . . . . . . . . . . . . . . . . .
ABCG1 . . . . . . . . . . . . . . . . . . . . . . . .
TMPRSS3 . . . . . . . . . . . . . . . . . . . . . . . .
UBASH3A . . 2 . . . . . . . . . . . . . . 1 . 1 . . . .
SLC37A1 . . . . . . . . . . . . . . . . . . . . . . . .
PDE9A . . . . . . . . . . . . . . . . . . . . . . . .
WDR4 . . . . . . . . . . . . . . . . . . . . . . . .
NDUFV3 . 1 . . . . . . . . . . . . . . . . . . . . . .
PKNOX1 . . 1 . . . . . . . . . . . . . . . . . . . . .
U2AF1 . . . 1 . 1 . . . . . . . 1 . . . . . . . . 2 .
AP001046.5 . . . . . . . . . . . . . . . . . . . . . . . .
AP001046.6 . . . . . . . . . . . . . . . . . . . . . . . .
SIK1 . . . . . . . . . . . . . 1 . . . . . . 1 . . .
HSF2BP . . . . . . . . . . . . . . . . . . . . . . . .
RRP1B . . . . . . . 1 . . 1 . . . . . . . . . . . . .
PDXK . . . . . . . . . . . . . . . . . . . . . . . .
CSTB . 1 . 1 1 2 . . . 1 . . . . . . . . 7 1 . . . .
RRP1 . . . . . . . . . . . . . . . . . . . . . . 1 .
AP001053.11 . . . . . . . . . . . . . . . . . . . . . . . .
AGPAT3 . . . . . . . . . . . . . . . . . . . . . . . .
TRAPPC10 . . . . . . . . . . . . . . . . . . 1 . . . . .
PWP2 . . . . . . . . . . . . . . . . . . . . . . . .
C21orf33 . 1 . 1 . . . . . . 1 . 1 . . . . . . . . . . .
AP001055.6 . . . . . . . . . . . . . . . . . . . . . . . .
AP001058.3 . . . . . . . . . . . . . . . . . . . . . . . .
ICOSLG . . . . . . . . . . 1 . . . . . . . . . . . . .
AIRE . . . . . . . . . . . . . . . . . . . . . . . .
PFKL . . . . 1 . . . . . . . . . . . . . . . 2 . . .
C21orf2 . . . . . . . . . . . . . . . . . . 1 . . . . .
AP001062.7 . . . . . . . . . . . . . . . . . . . . . . . .
TRPM2 . . . . . . . . 1 . . . . . . . . . . . . . . .
LRRC3 . . . . . . . . . . . . . . . . . . . . . . . .
UBE2G2 . . . . . . . . . . 1 . . . . . . . . . . . . .
SUMO3 . . . 1 . . 2 . . . . . . . . . . . . 1 1 . 1 .
PTTG1IP . . . . . . . . . . . . . . . . . . . 1 . . . .
ITGB2 . 2 . 1 2 . . . . 1 1 1 . 1 1 . 1 . 1 . 10 . . .
ITGB2-AS1 . 1 . 1 . . 1 . 1 1 . . . . . . . . 1 . . . . .
LL21NC02-1C16.2 . . . . . . . . . . . . . . . . . . . . . . . .
FAM207A . . . . . . . . . . . . . . . . . . . . 15 . . .
ADARB1 . . . . . . . . . . . . . . . . . . . . . . . .
POFUT2 . . . . . . . . . . . . . . . . . . 1 . . . . .
COL18A1 . . . . . . . . . . 1 . . . . . . . . . . . . .
SLC19A1 . . . . . . . . . . . . . . . . . . . . . . . .
PCBP3 . . 1 . . . . . . . . . . . . . . . . . . . . .
COL6A1 . . . . . . . . . . . . . . . . . . . . . . . .
COL6A2 . . . . . . . . . . . . . . . . . . . . . . . .
SPATC1L . . . . . . . . . . 1 . . . . . . . . . . . . .
LSS . . . . . . . . . . . . . . . . . . . . . . . .
MCM3AP-AS1 . . . . . . . . . . 1 . . . . . . . . . . . . .
MCM3AP . . . . . . . . . . . . . . . . . . . . . . . .
AP001469.9 . . . . . . . . . . . . . . . . . . . . . . . .
YBEY . 1 . . . . . . . . . . . . . . . . . . . . . .
C21orf58 . . . . . . . . . . . . . . . . . . . . . . . .
PCNT . . . . . . . . . . . . . . . . . . . . . . . .
DIP2A . . . . . . . . . . . . . . . . . . . . . . . .
S100B . . . . . . . . . . . . . . . . . . . . . . . .
PRMT2 . . 1 . . . . 1 . . . . . . . . . . . 1 . . . .
MT-ND1 8 8 4 5 1 4 8 7 . . 12 6 1 6 6 1 3 1 9 5 10 5 8 .
MT-ND2 3 10 2 5 . 3 18 16 . 3 7 1 1 2 4 3 5 10 4 3 7 2 9 2
MT-CO1 25 44 7 12 7 19 10 6 4 6 27 10 7 15 5 3 17 15 28 20 17 10 25 8
MT-CO2 10 53 2 7 . 2 15 5 3 10 4 1 2 3 5 3 8 11 6 9 8 11 10 .
MT-ATP8 . . . . . . . . . . . . . . . . . . . . . . . .
MT-ATP6 6 12 . 5 . 1 3 5 . 5 1 . 1 3 2 4 3 8 . 1 3 2 2 1
MT-CO3 5 15 4 4 1 . 10 10 2 4 17 4 5 7 1 1 30 3 5 5 8 4 11 2
MT-ND3 1 2 . . . . 1 . 3 . . . 1 . . . 1 . . . 1 . 1 1
MT-ND4L . . . 1 . . . . . . 1 1 1 . . . 2 . . . 1 . . .
MT-ND4 10 33 3 3 . 4 8 13 2 3 15 1 3 5 3 2 8 10 6 2 11 7 8 2
MT-ND5 1 1 2 2 2 . 2 1 . 1 3 . 1 1 . 1 . . . . 3 . 2 .
MT-ND6 . . . . . . . . . . . 1 . . . . . . . . . . . .
MT-CYB 4 8 4 2 1 3 8 7 1 . 16 1 3 1 1 1 3 1 6 5 7 3 9 1
AC145212.1 . . . . . . . . . . . . . . . . . . . . . . . .
AL592183.1 . 1 . . . . . . . . . . . . . . . . . . . . . .
AL354822.1 . . . . . . . . . . . . . . . . . . . . . . . .
PNRC2.1 . . . . . . . . . . . . . . . . . . . . . . . .
SRSF10.1 . . . . . . . . . . . . . . . . . . . . . . . .
HMHA1 . . 1 . . 1 . . ......
POLR2E . 1 . 1 1 . . . ......
GPX4 2 . . . 14 1 1 3 ......
SBNO2 . . . . . . . . ......
STK11 . . . . . . . . ......
ATP5D 5 1 1 2 4 . . 2 ......
MIDN . . . 1 1 . . 1 ......
CIRBP 2 1 1 1 1 5 2 5 ......
C19orf24 1 . . . . . . 9 ......
MUM1 . . . . . . . . ......
NDUFS7 1 1 1 . . 1 1 1 ......
GAMT . . . . . 1 . . ......
DAZAP1 . . . 1 . . . . ......
RPS15 7 9 8 10 27 20 7 6 ......
AC027307.3 . . . . . . . . ......
C19orf25 . . . . . . . . ......
PCSK4 . . . . . . . . ......
REEP6 . . . . . . . . ......
ADAMTSL5 . . . . . . . . ......
MBD3 . . . . . . . . ......
UQCR11.1 2 2 . . 2 2 . 4 ......
TCF3 . . . . . . . . ......
ATP8B3 . . . . . . . . ......
REXO1 . . . . . . . . ......
KLF16 . . . . . . . . ......
CTB-31O20.2 . . . 1 . . . . ......
ABHD17A . . . . . . . . ......
SCAMP4 . . . . . . . . ......
ADAT3 . . . . . . . . ......
CSNK1G2 . . . . . . . . ......
BTBD2 . . . . . . . . ......
AC005306.3 . . . . . . . . ......
MKNK2 . . . . . . . . ......
MOB3A . . . . 1 . . . ......
IZUMO4 . . . . . . . . ......
AP3D1 . . . . . . . . ......
DOT1L . . . . . . . . ......
PLEKHJ1 . . . . . . . . ......
SF3A2 . . . . . . . 1 ......
JSRP1 . . . . . . . . ......
OAZ1 10 1 . 2 25 5 6 24 ......
C19orf35 . . . . . . . . ......
LINGO3 . . . . . . . . ......
LSM7 . 1 . . . . . . ......
TIMM13 . . 1 . . 1 . . ......
LMNB2 . . . . . . . . ......
GADD45B . . . 1 . . . . ......
GNG7 . 1 . . . . . . ......
DIRAS1 . . . . . . . . ......
SLC39A3 . . . . . . . . ......
SGTA . . . 1 . . . . ......
THOP1 . . . . . . . . ......
ZNF554 . . . . . . . . ......
ZNF555 . . . . . . . . ......
ZNF57 . . . . . . . . ......
ZNF77 . . . . . . . . ......
TLE2 . . . . . . . . ......
AC005944.2 . . . . . . . . ......
AES 2 . 1 1 1 . 2 1 ......
GNA11 . . . . . . . . ......
GNA15 . . . . . . . . ......
S1PR4 1 . . . 1 . . . ......
NCLN . . . . . . . . ......
NFIC . . . . . . . . ......
C19orf77 . . . . . . . . ......
DOHH . . . . . . . . ......
FZR1 . 1 . . . . . . ......
MFSD12 . . . . . . . . ......
HMG20B . . . . . . . . ......
TBXA2R . . . . . . . . ......
CACTIN . . . . . . . . ......
PIP5K1C . . . . . . . . ......
TJP3 . . . . . . . . ......
APBA3 . . . . 1 . . . ......
MRPL54 1 . . . . . . . ......
MATK . . . . . . . . ......
ZFR2 . . . . . . . . ......
DAPK3 . . . . . . . . ......
EEF2 3 5 . 4 4 4 . 3 ......
PIAS4 . . . . . . . . ......
AC016586.1 . . . . . . . . ......
ZBTB7A . . . . . . . . ......
MAP2K2 . . . . . . . . ......
SIRT6 . . . . . . . . ......
EBI3 . . . . . . . . ......
CCDC94 . . . . . . . . ......
TMIGD2 . . . . . . . . ......
FSD1 . . . . . . . . ......
STAP2 . . . . . . . . ......
MPND . . . . . . . . ......
SH3GL1 . . . . . . . . ......
CHAF1A . . . . . . . . ......
UBXN6 . . . . . . . . ......
HDGFRP2 . . . . . . . 1 ......
PLIN5 . . . . . . . . ......
LRG1 . . . . . . . . ......
SEMA6B . . . . . . . . ......
TNFAIP8L1 . . . . . 1 . . ......
C19orf10 . . . . 3 . . . ......
DPP9 . . . 1 . . . . ......
FEM1A . . . . . . . . ......
AC005523.2 . . . . . . . . ......
TICAM1 . . . . . . . . ......
CTC-518P12.6 . . . . . . . . ......
PLIN3 . . . . . 1 . . ......
ARRDC5 . . . . . . . . ......
KDM4B . . . . . . . . ......
SAFB2 . . . . . 1 1 . ......
SAFB . . . . . . . 1 ......
RPL36 4 7 14 12 19 12 7 3 ......
C19orf70 . 1 . . . . . . ......
HSD11B1L . . . . . . . 1 ......
LONP1 . 1 . . . . . . ......
PRR22 . . . . . . . . ......
DUS3L . . . . . . . . ......
AC024592.12 . . . . . . . . ......
NDUFA11 2 . . 1 . . . 1 ......
VMAC . . . . . . . . ......
AC104532.4 . . . . 1 . . . ......
CAPS . . . . . . . . ......
RANBP3 . . . . . . . . ......
RFX2 . . . . . . . . ......
CTC-503J8.6 . . . . . . . . ......
CLPP . . 1 . . 1 . . ......
ALKBH7 . . . 1 1 2 1 1 ......
PSPN . . . . . . . . ......
GTF2F1 2 . . . 1 . . . ......
KHSRP . . . . . . . . ......
SLC25A23 . . . . . . . . ......
CRB3 . . . . . . . . ......
DENND1C . . . . . . . . ......
TUBB4A . . . . . . . . ......
CD70 . . . . . . . . ......
TNFSF14 . . . . . . . . ......
C3 . . . . . . . . ......
GPR108 . . . . 1 . . . ......
TRIP10 . . . . . . . . ......
SH2D3A . . . . . . . . ......
VAV1 . . . . . . . 3 ......
EMR1 . . . . . . . 1 ......
ZNF557 . . . . . . . . ......
INSR . . . . . . . . ......
CTB-133G6.1 . . . . . . . . ......
CTB-133G6.2 . . . . . . . . ......
ARHGEF18 . . . . . . . . ......
PEX11G . . . . . . . . ......
ZNF358 . . . . . . . . ......
MCOLN1 . . . . . 1 . . ......
PNPLA6 . . . . . . . . ......
XAB2 . . . . . . 1 . ......
PET100 . 1 . . . . . 1 ......
CTD-3214H19.4 . . . . . . . . ......
PCP2 . . . . . . . . ......
STXBP2 . 1 . . 1 . . 1 ......
RETN 2 . . . . . . . ......
C19orf59 2 . . . . . . . ......
TRAPPC5 1 . . . 1 1 1 1 ......
FCER2 . 1 . . . . . . ......
CLEC4G . . . . . . . . ......
EVI5L . . . . . . . . ......
MAP2K7 . . . . . . . . ......
TGFBR3L . . . . . . . . ......
SNAPC2 1 . . . . . . . ......
TIMM44 . . . . . . . . ......
ELAVL1 . . . . . . . . ......
CTD-2325M2.1 . . . . . . . . ......
CERS4 . . . . . . . . ......
CD320 . . . . . . . . ......
NDUFA7.1 1 . . . 1 . 1 . ......
RPS28 5 10 6 14 20 10 5 7 ......
KANK3 . . . . . . . . ......
RAB11B-AS1 . . . . . . . . ......
RAB11B . . . 1 . 1 . . ......
MARCH2 . . . . . . . 1 ......
HNRNPM . 1 . . . . . . ......
PRAM1 2 . . . . . . 1 ......
ZNF414 . . . . . . . . ......
MYO1F . . . 1 6 . . . ......
ADAMTS10 . . . . . . . . ......
ZNF558 . . . . . . . . ......
ZNF317 . . . . . . . . ......
ZNF699 . . . . . . . . ......
ZNF559 . . . . . . . . ......
ZNF266 . . . . . . . . ......
ZNF426 . . . . . . . . ......
ZNF121 . . . . . . . . ......
ZNF561 . . . . . . . . ......
C19orf82 . . . . . . . . ......
ZNF562 . . . . . . . . ......
ZNF846 . . . . . . . . ......
FBXL12 . . . . . . . . ......
UBL5 1 1 . . 1 1 . 1 ......
CTD-2623N2.11 . . . . . . . . ......
PIN1 . . . . 1 . . . ......
OLFM2 . . . . . . . . ......
C19orf66 . . . 1 1 1 4 . ......
ANGPTL6 . . . . . . . . ......
PPAN . . . . . . . . ......
P2RY11 . . . . . . . . ......
EIF3G 2 . . 2 2 . 1 . ......
DNMT1 . . . . 1 . . . ......
CTD-2369P2.2 . . . . . . . . ......
MRPL4 . . . 1 . . . . ......
ICAM1 . . . . . . . . ......
ICAM4 . . . . . . . 1 ......
ZGLP1 1 . . . . . . . ......
FDX1L . . . . . . . . ......
RAVER1 1 . . . . . . . ......
ICAM3 1 . . 2 2 1 1 3 ......
TYK2 . . . . . . . . ......
CDC37 1 . . . . . . . ......
PDE4A . . . . . . . . ......
KEAP1 . . . . . . 1 . ......
S1PR5 . . . . . . . . ......
ATG4D . . . . . . . . ......
KRI1 . . 1 . . . . . ......
CDKN2D . . . . . 2 . . ......
SLC44A2 . 1 . . 3 . . . ......
ILF3-AS1 . . . . 1 2 . . ......
ILF3 . . . . 1 . . . ......
QTRT1 . . . . 1 . . . ......
DNM2 . . . . . . . . ......
TMED1 . . . . . . . . ......
C19orf38 . . . . . . . . ......
CARM1 . . . . . . . . ......
YIPF2 . . . . . . . . ......
C19orf52 . . . . . . . . ......
SMARCA4 . . . . . . . . ......
LDLR . . . . . . . . ......
RAB3D . . . . . . . . ......
TMEM205 . . . . . . . . ......
CCDC159 . . . . . . . . ......
DKFZP761J1410 . . . . . . . . ......
SWSAP1 . . . . . . . . ......
EPOR . . . . . . . . ......
CCDC151 . . . . . . . . ......
PRKCSH . . 1 1 . . . . ......
ZNF653 . . . . . . . . ......
ECSIT . . . . . . . 1 ......
ELOF1 . . . . . . . . ......
ZNF627 . . . . . . . . ......
ACP5 . . . . . . . . ......
ZNF823 . . . . . . . . ......
ZNF441 . . . . . . . . ......
ZNF440 . . . . . . . . ......
ZNF439 . . . . . . . . ......
ZNF69 . . . . . . . . ......
ZNF700 . . . . . . . . ......
ZNF763 . . . . . . . . ......
ZNF433 . . . . . . . . ......
ZNF844 . . . . . . . . ......
ZNF625 . . . . . . . . ......
ZNF136 . . . . . . . . ......
CTD-2666L21.1 . . . . . . . . ......
ZNF44 . . . . . . . . ......
ZNF563 . . . . . . . . ......
ZNF442 . . . . . . . . ......
ZNF799 . . . . . . . . ......
ZNF443 . . . . . . . . ......
ZNF709 . . . . . . . . ......
ZNF564 . . . . . . . . ......
ZNF490 . . . . . . . . ......
ZNF791 . . . . . . . . ......
CTD-2192J16.22 . . . . . . . . ......
MAN2B1 . . . . . . . . ......
WDR83 . . . . . . . . ......
WDR83OS . 1 . . 5 . . 10 ......
DHPS . . . . . . . . ......
FBXW9 . . . . . . . 1 ......
TNPO2 . . . . . . . . ......
C19orf43 . 2 . 1 5 . . 2 ......
ASNA1 . 1 . . . . . . ......
HOOK2 . . . . . . . . ......
CTD-2659N19.9 . . . . . . . . ......
JUNB 15 3 5 13 25 12 4 10 ......
PRDX2 . . 1 2 . 1 . . ......
RNASEH2A . 1 . . . . . . ......
DNASE2 . . . . 1 . . . ......
GCDH . . . . . . 1 . ......
FARSA . 1 . . . . . . ......
CALR . . . . . . 1 1 ......
CTC-425F1.4 . . . . . . . . ......
RAD23A . . . . . 8 . . ......
GADD45GIP1 . . . 2 1 . 1 . ......
NFIX . . . . . . . . ......
LYL1 . 1 . . 2 . . 1 ......
TRMT1 . . . . . . . . ......
NACC1 . . . . . . . . ......
STX10 . . . . . . . 1 ......
IER2 1 . . 2 . . . 2 ......
CTC-250I14.6 . . . . . . . . ......
CACNA1A . . . . . . . . ......
CCDC130 . . . . . . . . ......
MRI1 . . . . . . . . ......
C19orf53 . . . 3 1 . . . ......
ZSWIM4 . . . . . . . . ......
MIR24-2 . . . 1 . 1 . 1 ......
CC2D1A . . . . . . . 1 ......
DCAF15 . . . . . . . . ......
RFX1 . . . . . . . . ......
CTB-55O6.4 . . . . . . . . ......
IL27RA . . . . . 2 . . ......
hsa-mir-1199 . . . . . . . . ......
SAMD1 . . . . . . . . ......
PRKACA . . . . . . . . ......
ASF1B . . . . . . . . ......
CTB-55O6.12 1 . . . . . . . ......
LPHN1 . . . . . . . . ......
CD97 . . . . 2 . . 1 ......
DDX39A . . . . 1 . . . ......
PKN1 1 . . . 2 . . 2 ......
GIPC1 . . . . . . . . ......
DNAJB1 . . 1 . 1 . . 1 ......
TECR . . 1 . . . . . ......
NDUFB7 . . . . 2 . . 2 ......
CLEC17A . . . . . . . . ......
EMR3 . . . . . . . . ......
ZNF333 . . . . . . . . ......
EMR2 . . . . . . . 2 ......
ILVBL . . . . . . . . ......
EPHX3 . . . . . . . 1 ......
AC004257.3 . . . . . . . . ......
BRD4 . . . . . . 1 1 ......
AKAP8 . . . . . . . . ......
AKAP8L . . . . 1 . . . ......
WIZ . . . . . . . . ......
RASAL3 . . . . . . . . ......
PGLYRP2 . . . . . . . . ......
CYP4F22 . . . . . . . . ......
TPM4 . . . . . . . . ......
RAB8A . . . 1 2 . . . ......
CTD-2231E14.8 . . . . . . . . ......
HSH2D 1 . . . 1 . . 1 ......
FAM32A . . . 1 . . . 1 ......
AP1M1 1 . . . . . . . ......
CTD-2562J15.6 . . . . . . 1 . ......
KLF2 . . . . 5 1 1 2 ......
EPS15L1 1 . . . . . . . ......
C19orf44 . . . . . . . . ......
CHERP . . . . . . . . ......
SLC35E1 . . . . . . . 1 ......
MED26 . . . . . . . . ......
SMIM7 . . . . . . . 1 ......
TMEM38A . . . . . . . . ......
SIN3B . . . . . . . . ......
CPAMD8 . . . . . . . . ......
HAUS8 . . . . . . . . ......
MYO9B 1 . . . 1 . . . ......
USE1 1 1 . . . . . . ......
OCEL1 . . 1 . . 1 . . ......
NR2F6 . . . . . . . . ......
BABAM1 . . . . 1 . 1 . ......
ANKLE1 . . . . . . . . ......
ABHD8 . . . . 1 . . . ......
MRPL34 . . 1 . 2 . . . ......
DDA1 . . . . . . . . ......
GTPBP3 . . . . 1 . . . ......
PLVAP . . . . . . . . ......
BST2 . 2 1 . 1 . 3 1 ......
CTD-2521M24.9 . . . 1 . . . . ......
MVB12A . . . . . . . . ......
SLC27A1 . . . . . . . . ......
CTD-3131K8.2 . . . . . . . . ......
PGLS 1 . . . 3 . 2 2 ......
FAM129C . . . . . . . . ......
COLGALT1 . . . . . . . . ......
MAP1S . . . . . . . . ......
FCHO1 . . . . . . . . ......
INSL3 . . . . . . . . ......
JAK3 . . . . . . . . ......
RPL18A 8 18 10 22 35 23 10 15 ......
CCDC124 . . . . . . . . ......
KCNN1 . . . . . . . . ......
ARRDC2 . 1 . 1 . . . 1 ......
IL12RB1 . . . . . . . . ......
MAST3 . . . . . . . . ......
PIK3R2 . . . . . . . . ......
IFI30 1 . . . . . . . ......
MPV17L2 . . . 1 . . . . ......
RAB3A . . . . . . . . ......
PDE4C . . . . . . . . ......
KIAA1683 . . . . . . . . ......
JUND . . . . 1 1 . . ......
LSM4 . . . . . . 1 1 ......
PGPEP1 . . . . 1 . . . ......
LRRC25 . . . . 1 . . 2 ......
SSBP4 . . . 1 1 . 1 . ......
ISYNA1 . . . . . . . . ......
ELL 1 . . 1 . . . . ......
FKBP8 1 . . . 4 . . . ......
KXD1 . 1 . . . . . . ......
AC005253.2 . . . . . . . . ......
AC005253.4 . . . . . . . . ......
UBA52 12 2 12 3 14 4 5 6 ......
C19orf60 1 . . . 1 . . 1 ......
KLHL26 . . . . . . . . ......
CRTC1 . . . . . . . . ......
UPF1 . . . . 1 . . . ......
COPE 2 . . . 1 . 1 1 ......
DDX49 . . . . . . . . ......
HOMER3 . . . . . . . . ......
AC005932.1 . . . . . . . . ......
SUGP2 . . . . . . . . ......
AC004447.2 . . . . . . . . ......
ARMC6 . . . . . . . . ......
SLC25A42 . . . . . . . . ......
TMEM161A . . . . . . . . ......
MEF2BNB-MEF2B . . . . . . . . ......
MEF2BNB . . . . . . . . ......
RFXANK . . . . 1 . . . ......
NR2C2AP . . . 1 . . . . ......
SUGP1 . . . . . . . . ......
MAU2 . . . . . . . . ......
GATAD2A . . . . . . . . ......
TSSK6 . . . . . . . . ......
NDUFA13 2 1 . . 5 1 . 1 ......
PBX4 . . . . . . . . ......
LPAR2 . . . . . . . . ......
GMIP . . . . . . . . ......
ATP13A1 . . . . . . . . ......
ZNF101 . . . 1 . . . . ......
ZNF14 . . . . . . . . ......
LINC00663 . . . . . . . . ......
ZNF506 . . . . . . . . ......
ZNF253 . . . . . . . . ......
ZNF93 . . . . . . . . ......
ZNF682 . . . . . . . . ......
ZNF90 . . . . . . 1 . ......
CTC-260E6.6 . . . . . . . . ......
ZNF486 . . . . . . . . ......
ZNF737 . . . . . . . . ......
ZNF626 . . . . . . . . ......
ZNF85 . . . . . . . . ......
ZNF430 . . . . . . 1 . ......
ZNF714 . . . . . . . . ......
ZNF431 . . . . . . . . ......
ZNF708 . . . . . . . . ......
ZNF738 . . . . . . . . ......
ZNF493 . . . . . . . . ......
ZNF429 . . . . . . 2 . ......
ZNF100 . 1 . . . . . . ......
ZNF43 . . . . . . . . ......
ZNF208 . . . . . . . . ......
ZNF257 . 1 . . . . . . ......
ZNF492 . . . . . . . . ......
ZNF724P . . . . . . . . ......
RP11-15H20.7 . . . . . . . . ......
ZNF91 . . . 1 . . . . ......
ZNF675 . . . . . . . . ......
ZNF681 . . . . . . . . ......
RPSAP58 . 5 1 2 1 5 1 1 ......
ZNF726 . . . . . . . . ......
CTD-2017D11.1 . . . . . . . . ......
ZNF254 . 2 . . . . . . ......
LINC00662 1 . . . . . . . ......
CTC-459F4.3 . . . . . . . . ......
UQCRFS1 1 . . . 1 1 1 1 ......
CTB-32O4.2 . . . . . . . . ......
POP4 . . . . . . . . ......
PLEKHF1 . . . . . . . . ......
C19orf12 . . . . 1 . . . ......
CCNE1 . . . . . . . . ......
URI1 . . . . . . . . ......
ZNF507 . . . . . . . . ......
DPY19L3 . . . . . . . . ......
PDCD5 . . . . . . 1 1 ......
ANKRD27 . . . . . . . . ......
CTD-2538C1.2 . . . . . . . . ......
NUDT19 . . . . . . . . ......
CTD-2085J24.4 . . . . . . . . ......
CEP89 . . . . . . . . ......
C19orf40 . . . . . 1 . . ......
GPATCH1 . . . 1 . . . . ......
LRP3 . . . . . . . . ......
CEBPA . . . . . . . . ......
CEBPA-AS1 . . . . . . . . ......
CEBPG . . . . . . . . ......
PEPD . . . . . . . . ......
KCTD15 . . . . . . . . ......
LSM14A . . . . 1 . 1 1 ......
KIAA0355 . . . . . . 1 . ......
GPI . . . 1 . . . 1 ......
PDCD2L . 1 . . . . . . ......
UBA2 . . . . . 1 1 . ......
SCGB1B2P . . . . . . . . ......
ZNF302 1 . . . . . . . ......
ZNF181 . . . . . . 1 . ......
ZNF599 . . . . . . . . ......
CTC-523E23.1 . . . . . . . . ......
CTC-523E23.11 . . . . . . . . ......
ZNF30 . . . . . . . . ......
ZNF792 . . . . . . . . ......
GRAMD1A . . . . 1 . . . ......
SCN1B . . . . . . . . ......
FXYD1 . . . . . . . . ......
FXYD7 . . . . . . . . ......
FXYD5 2 2 2 3 4 1 2 2 ......
LSR . . . . 1 . . . ......
USF2 . . . . . . . . ......
CD22 . . . . . . . . ......
FFAR3 . . . . . . . . ......
GPR42 . . . . . . . . ......
FFAR2 1 . . . . . . . ......
DMKN . . . . . . . . ......
AD000090.2 . . . . . . . . ......
TMEM147 . 2 . . . . . . ......
HAUS5 . . . . . . . . ......
RBM42 . . . . . . . 1 ......
ETV2 . . . . . . . . ......
COX6B1 . 2 . 2 4 1 1 4 ......
ZBTB32 . . . . . . . . ......
KMT2B . . . . . . . . ......
IGFLR1 . . . . . 1 . . ......
AD000671.6 . . . . . . . . ......
U2AF1L4 . . . . . . . . ......
PSENEN 1 . . . . . . 1 ......
LIN37 . . . . . . . . ......
C19orf55 . . . . . . . . ......
ARHGAP33 . . 1 . . . . . ......
NFKBID . . . . . . . . ......
HCST . . 1 3 . . . 1 ......
TYROBP 10 . 1 . 17 . . 5 ......
LRFN3 . . . . . . . . ......
SDHAF1 1 . 1 . 1 . . . ......
ALKBH6 . . . . . . . . ......
AC002116.7 . . . . . . . . ......
THAP8 . . . . . . . . ......
WDR62 . . . . . . . . ......
POLR2I . . . . 2 . . . ......
TBCB . . . . 1 . . . ......
CAPNS1 1 . . . 2 . 1 1 ......
ZNF565 . . . . . . . . ......
ZNF146 . . . . 1 . . . ......
LINC00665 . . . . . . . . ......
ZFP14 . . . . . 1 . . ......
ZFP82 . . . . . . . . ......
ZNF566 . . . . . . . . ......
CTD-2630F21.1 . . . . . . . . ......
ZNF260 . . . . . 1 . . ......
ZNF529 . . . . . . . . ......
AC092295.7 . . . . . . . . ......
ZNF382 . . . . . . . . ......
ZNF461 . . . . . . . . ......
AC074138.3 . . . . . . . . ......
ZNF567 . . . . . . . . ......
CTD-2162K18.4 . . . . . . . . ......
CTD-2162K18.5 . . . . . . . . ......
ZNF790 . . . . . . . . ......
ZNF345 . . . . . . . . ......
ZNF829 . . . . . . . . ......
ZNF568 . . . . . . . . ......
ZNF420 . . . . . . 1 . ......
CTC-454I21.3 . . . . . . . . ......
ZNF585A . . . . . . . . ......
ZNF585B . . . . . . . . ......
ZNF383 . . . . . . . . ......
HKR1 . . . . . . . . ......
ZNF527 . . . . . . . . ......
ZNF569 . . . . . . . . ......
ZNF570 . . . . . . . . ......
CTD-2086O20.3 . . . . . . . . ......
CTD-3064H18.1 . . . . . . . . ......
ZNF793 . . . . . . . . ......
ZNF571-AS1 . . . . . . . . ......
ZNF540 . . . . . . . . ......
ZNF571 . . . . . . . . ......
ZFP30 . . . . . . . . ......
ZNF781 . . . . . . . . ......
ZNF573 . . . . . . . . ......
SIPA1L3 . . . . . . . . ......
SPINT2 . . . . . . . . ......
PPP1R14A . . . . . . . . ......
CTB-102L5.4 . . . . . . . . ......
C19orf33 . . . . . . . . ......
YIF1B . . . . . . . . ......
KCNK6 . . . . . . . . ......
AC026806.2 . . . . . . . . ......
CATSPERG . . . . . . . . ......
PSMD8 . . . . . 2 2 1 ......
FAM98C . . . . . . . . ......
RASGRP4 . . . . . . . 1 ......
RYR1 . . . . . . . . ......
MAP4K1 . . . . . . . . ......
EIF3K 2 . . 3 4 2 1 1 ......
ACTN4 . . . . . . . . ......
CAPN12 . . . . . . . . ......
CTD-2540F13.2 . . . . . . . . ......
LGALS4 . . . . . . . . ......
ECH1 . . . 1 . . . . ......
HNRNPL . . . . . . . . ......
RINL . . . 1 . . . . ......
SIRT2 1 . . . . . . 1 ......
NFKBIB . . . . . . 1 . ......
SARS2 . . . . . . . . ......
MRPS12 . 1 . . . . . . ......
PAPL . . . . . . . . ......
PAK4 . . . . . . . . ......
IFNL1 . . . . . . . . ......
LRFN1 . . . . . . . . ......
GMFG 1 1 . 2 4 2 2 2 ......
CTC-246B18.10 . . . . . . . . ......
SAMD4B . . . . . . . . ......
PAF1 . . . . . . . . ......
MED29 . . . . . . . 1 ......
ZFP36 1 . . . 4 . . 5 ......
PLEKHG2 . . . . . . . . ......
RPS16 6 14 5 18 21 23 22 9 ......
SUPT5H . . . . . . . . ......
TIMM50 . . . . 1 . . . ......
DLL3 . . . . . . . . ......
EID2B . . . . . . . . ......
EID2 . . . . . . . . ......
DYRK1B . . . . . . . . ......
FBL . 1 . . 1 1 1 3 ......
FCGBP . . . . . . . . ......
PSMC4 . . . . . . . 1 ......
ZNF546 . . . . . . . . ......
ZNF780B . . . . . . . . ......
ZNF780A . . . . . . . . ......
MAP3K10 . . . . . . . . ......
AKT2 . . . . 1 . . . ......
C19orf47 . . . . . . . . ......
PLD3 . . . . . . . . ......
SERTAD1 . . . . . . . 1 ......
SERTAD3 . . . . . . . . ......
BLVRB 1 . . . 1 . . . ......
SHKBP1 1 . . 1 . . . . ......
LTBP4 . . . . . . . . ......
NUMBL . . . . . . . . ......
ADCK4 . . . . . . . . ......
ITPKC . . . . . . . . ......
C19orf54 . . . . . . . . ......
SNRPA . 1 . . . . . . ......
RAB4B . . . . . . . . ......
EGLN2 . . . . . . . . ......
CYP2S1 . . . . . . . . ......
AXL . . . . . . . . ......
HNRNPUL1 . . . . 1 . . 1 ......
TGFB1 1 . . . 1 . 1 . ......
CCDC97 . . . . . . . . ......
TMEM91 . . . . . . . . ......
B9D2 1 . . . . . . . ......
BCKDHA . . . . . . . . ......
EXOSC5 . . . . . . . . ......
B3GNT8 . . . . . . . . ......
ATP5SL . . . . . . . . ......
AC006129.1 . . . . . . . . ......
AC006129.4 . . . . . . . . ......
AC006129.2 . . . . . . . . ......
CEACAM21 . . . . . . . . ......
CEACAM4 1 . . . . . . . ......
CEACAM3 . . . . . . . . ......
RPS19 5 14 8 38 100 22 26 27 ......
CD79A . 2 . . . . . . ......
ARHGEF1 . . . . . 1 1 . ......
RABAC1 . . . . 1 . . 1 ......
ATP1A3 . . . . . . . . ......
ZNF574 . . . . . . . . ......
POU2F2 . 1 . . 1 . . 1 ......
CTC-378H22.1 . . . . . . . . ......
CTC-378H22.2 . . . . . . . . ......
DEDD2 . . . . . . . . ......
ZNF526 . . . . . . . . ......
GSK3A . . . . . . . . ......
ERF . . . . . . . . ......
CIC . . . . . . . . ......
PAFAH1B3 . . . . . . . . ......
PRR19 . . . . . . . . ......
MEGF8 . . . . . . . . ......
CNFN . . . . . . . . ......
LIPE-AS1 . . . . . . . . ......
LIPE . . . . . . . . ......
CEACAM1 . . . . . . . . ......
LYPD3 . . . . . . . . ......
PHLDB3 . . . . 1 . . . ......
ETHE1 . . . . 2 . 1 . ......
ZNF575 . . . . . . . . ......
XRCC1 . . . . . . . . ......
PINLYP . . . . . . . . ......
IRGQ . . . . . . . . ......
ZNF576 . . . . . . . . ......
SRRM5 . . . . . . . . ......
ZNF428 . . . . . . . . ......
PLAUR . . . . 1 . . . ......
SMG9 . . . . . . . . ......
KCNN4 . . . . . . . . ......
ZNF283 . . . . . . . . ......
ZNF404 . . . . . . . . ......
RP11-15A1.3 . . . . . . . . ......
ZNF45 . . . . . . . . ......
ZNF155 . . . . . . . . ......
RP11-15A1.7 . . . . . . . . ......
ZNF230 . . . . . . . . ......
ZNF222 . . . . . . . . ......
ZNF223.1 . . . . . . . . ......
ZNF284 . . . . . . . . ......
ZNF224 . . . . . . . . ......
ZNF225 . . . . . . . . ......
ZNF234 . . . . . . . . ......
ZNF226 1 . . . . . . 1 ......
ZNF227 . . . . . . . . ......
ZNF235 . . . . . . . . ......
ZNF180 . . . . . . . . ......
PVR . . . . . . . . ......
BCL3 . . . . . . . . ......
PVRL2 . . . . . . . . ......
CTB-129P6.4 . . . . . . . . ......
TOMM40 . . . . . . . . ......
CLPTM1 . . . . . . 1 1 ......
RELB . . . . . . . . ......
CLASRP . . . . . . . . ......
ZNF296 . . . . . . . . ......
GEMIN7 . . . . . . . . ......
MARK4 . . . . . . . . ......
PPP1R37 . . . . . . . . ......
TRAPPC6A . . 2 1 1 . . 1 ......
BLOC1S3 . . . . . . . . ......
CKM . . . . . . . . ......
ERCC2 . . . . . . . . ......
PPP1R13L . . . . . . . . ......
CD3EAP . . . . . . . . ......
ERCC1 . . . . . . . . ......
FOSB 5 . . . 2 . 1 2 ......
PPM1N . . . . 1 . . . ......
VASP 1 . . 1 2 . . 3 ......
OPA3 . . . . . . . . ......
EML2 . . . . . . . . ......
C19orf83 . . . . . . . . ......
GIPR . . . . . . . . ......
SNRPD2 1 1 . 2 2 2 1 1 ......
FBXO46 . . . . . . . . ......
DMPK . . . . . . . . ......
DMWD . . . . . . . . ......
SYMPK . . . . . . . . ......
IRF2BP1 . . . . . . . . ......
MYPOP . . . . . . . . ......
CCDC61 . . . . . . . . ......
PPP5C . . . . . . . . ......
PPP5D1 . . . . . . . . ......
CALM3 . . . 1 1 . . . ......
CTB-12A17.3 . . . . . . . . ......
PTGIR . . . . . . . . ......
PRKD2 . . . . . . . . ......
STRN4 . . . . . . . . ......
FKRP . . . . . . . . ......
SLC1A5 . . . . . . . . ......
AP2S1 . . 1 . 5 . . 1 ......
ARHGAP35 . 1 . . . . . . ......
TMEM160 . 1 1 1 . . . . ......
ZC3H4 . 1 . . . . . . ......
SAE1 . . . . . . . . ......
BBC3 . . . . . . . . ......
CCDC9 . . . . . . . . ......
PRR24 . . . . . . . . ......
C5AR1 1 . . 1 1 . . 2 ......
C5AR2 . . . . . . . . ......
DHX34 . . . . . . . . ......
KPTN . . . . . . . . ......
NAPA-AS1 . . . . . . . . ......
NAPA . . . 1 1 . . . ......
ZNF541 . . . . . . . . ......
CTD-2571L23.8 . . . . . . . . ......
GLTSCR1 . . . . . . . . ......
GLTSCR2 . . 1 2 4 5 3 . ......
SEPW1 1 . . . 1 . . 1 ......
PLA2G4C . . . . . . . . ......
LIG1 . . . . . . . . ......
C19orf68 . . . . . . . . ......
CARD8 . . . . . . . 1 ......
CTC-241F20.3 . . . . . . . . ......
EMP3 2 2 1 . 2 1 2 8 ......
TMEM143 . . . . . . . . ......
KDELR1 . . . . . . . . ......
GRWD1 . . . . . . . . ......
KCNJ14 . . . . . . . . ......
CYTH2 . . . . 1 . . . ......
CTC-273B12.10 . . . . . . . . ......
RPL18 7 7 7 16 47 19 10 7 ......
SPHK2 . . . . . . . . ......
DBP . 1 . . . . . . ......
CA11 . . . . . 1 . . ......
MAMSTR . . . . . . . . ......
RASIP1 . . . . . . . . ......
BCAT2 . . . . . . . . ......
HSD17B14 . . . . . . . . ......
PLEKHA4 . . . . . . . . ......
PPP1R15A . 1 . 1 3 . . . ......
TULP2 . . . . . . . . ......
NUCB1 1 . . 1 . . . . ......
DHDH . . . . . . . . ......
BAX 2 . . 1 2 . 1 1 ......
FTL 113 11 3 7 163 39 4 89 ......
GYS1 . . . . . . . . ......
RUVBL2 . . . . . . . . ......
SNRNP70 1 . . 1 . 1 1 . ......
LIN7B . . . . . . . . ......
C19orf73 . . . . . . . . ......
TRPM4 . . . . . . . . ......
SLC6A16 . . . . . . . . ......
CTC-301O7.4 . . . . . . . . ......
CD37 3 2 1 . 1 2 . 3 ......
PIH1D1 . . . . . . . . ......
ALDH16A1 . . . . . . . . ......
FLT3LG . . . . . 2 1 1 ......
RPL13A 18 41 23 49 58 52 40 24 ......
RPS11 2 6 1 4 15 6 . 6 ......
FCGRT 1 . . . 3 . . 2 ......
RCN3 . . . . . . . . ......
NOSIP . . . . . 2 1 1 ......
PRRG2 . . . . . . . . ......
PRR12 . . . . . . . . ......
RRAS . . . . . . . . ......
SCAF1 . . . . . . . . ......
IRF3 . . . . 2 . . . ......
BCL2L12 . . . . . . . . ......
PRMT1 . . . 1 . . 1 . ......
TSKS . . . . . . . . ......
AP2A1 . . . . . . . . ......
FUZ 1 . . . . . . . ......
MED25 . . . . . . . . ......
PTOV1-AS1 . . . . . . . . ......
PTOV1 . . 1 . . . 1 . ......
AC018766.4 . . . . . . . . ......
PNKP . 1 . . . . . . ......
AKT1S1 1 . . . . . . . ......
TBC1D17 . . . . . . . . ......
IL4I1 . . . . . . . . ......
NUP62 . . . . 3 . . . ......
ATF5 . . . . . . . . ......
VRK3 . 1 . . . . 1 . ......
ZNF473 . . . . . . . . ......
KCNC3 . . . . . . . . ......
NR1H2 . . . . 1 . . . ......
NAPSA . . . . . . . . ......
POLD1 . . . . . . . . ......
SPIB . . . . . . . . ......
EMC10 . . . . . . . 1 ......
CTD-2545M3.2 . . . . . . . . ......
JOSD2 1 . . . 2 . . . ......
CLEC11A . . . . 1 . . . ......
C19orf48 . 1 . . 1 . . . ......
KLK1 . . . . . . . . ......
CTU1 . . . . . . . . ......
SIGLEC9 . . . . . . . . ......
SIGLEC7 . . . . . . . . ......
CD33 . . . . 1 . . 1 ......
VSIG10L . . . . . . . . ......
ETFB . . . . . . . 1 ......
CLDND2 . . . . . . . . ......
NKG7 . . . . 2 . . . ......
LIM2 . . . . . . . . ......
SIGLEC10 . . . . . . . 2 ......
CTD-2616J11.2 . . . . . . . . ......
SIGLEC12 . . . . . . . . ......
ZNF175 . . . . . . . . ......
AC018755.1 . . . . . . . . ......
SIGLEC5 . . . . . . . 1 ......
SIGLEC14 . . . . . . . . ......
FPR1 2 . . . . . . . ......
FPR2 . . . . . . . . ......
ZNF577 . . . . . . . . ......
ZNF649 . . . . . . . . ......
ZNF613 . . . . . . . . ......
ZNF350 . . . . . . . . ......
ZNF615 . . . . . 1 . . ......
ZNF614 . . . . . . . . ......
ZNF432 . . . . . . . . ......
ZNF841 . . . . . . . . ......
ZNF616 . . . . . . . . ......
ZNF836 . . . . . . . . ......
PPP2R1A 1 . . . 1 . . 3 ......
ZNF766 . . . . . . . . ......
ZNF480 . . . 1 . . . . ......
ZNF880 . . . . . . . . ......
ZNF528 . . . . . . . . ......
ZNF578 . . . . . . . . ......
ZNF808 . . . . . . . . ......
ZNF701 . . . . . . . . ......
ZNF83 . . . . . . . . ......
ZNF611 . . . . . . . . ......
ZNF600 . . . . . . . . ......
ZNF28 . . . . . . . . ......
ZNF468 . . . . . . . . ......
ZNF320 . . . . . . . . ......
ZNF816 . . . . . . . . ......
ZNF160 . . . . . . 1 . ......
ZNF415 . . . . . . . . ......
ZNF347 . . . . . . . . ......
ZNF665 . . . . . . . . ......
ZNF677 . . . . . . . . ......
ZNF845 . . . . . . . . ......
ZNF525 . . . . . . . . ......
ZNF765 . . . . . . . . ......
ZNF813 . . . . . . . . ......
ZNF331 . . . . . 1 . . ......
NLRP12 . . . . . . . . ......
MYADM . . 1 . . . . 1 ......
AC008440.5 . . . . . . . . ......
VSTM1 . . . . . . . . ......
OSCAR . . . . 1 . . . ......
NDUFA3 . . 1 . 2 . . 1 ......
TFPT . . . . . . . . ......
PRPF31 . . . . . . . . ......
CNOT3 . . . . 1 . . . ......
LENG1 . 1 . 2 1 . . . ......
TMC4 . . . . . . . . ......
MBOAT7 . . . . . . . . ......
TSEN34 1 . . . . . . . ......
CTD-3093M3.1 . . . . . . . . ......
RPS9 10 7 2 14 72 25 7 12 ......
LILRB3 1 . . . . . . . ......
LILRA6 . . . . 1 . . . ......
LILRB2 . . . 1 3 . . . ......
LILRA3 1 . . . 2 . . 1 ......
LILRA5 . . . . 1 . . 1 ......
LILRA4 . . . . . . . . ......
CTD-2587H19.1 . . . . . . . . ......
LAIR1 . . . . 1 . . . ......
LENG8-AS1 . . . . . . . . ......
LENG8 . . . . 1 1 . . ......
LENG9 . . . . . . . . ......
LAIR2 . . . . . . . . ......
LILRA2 . . . . . . . . ......
LILRB1 . . 1 . . . . . ......
LILRA1 . . . . . . . . ......
LILRB4 . . . . . . . . ......
KIR3DL1 . . . . . . . . ......
KIR2DL3 . . . . . . . . ......
KIR3DL2 . . . . . . . . ......
FCAR . . . . . . . . ......
CTB-61M7.2 . . . . . . . . ......
NCR1 . . . . . . . . ......
NLRP2 . . . . . . . . ......
CTC-550B14.7 . . . . . . . . ......
GP6 . . . . . . . . ......
RDH13 . . . . . . . . ......
EPS8L1 . . . . . . . . ......
PPP1R12C . . . . . . . . ......
TNNT1 . . . . . . . . ......
SYT5 . . . . . . . . ......
TMEM86B . . . . . . . . ......
PPP6R1 . . . . . . . . ......
HSPBP1 . . . . . 1 . . ......
BRSK1 . . . . . . . . ......
TMEM150B . . . . . . . . ......
SUV420H2 . . . . . . . . ......
COX6B2 . . . . . . . . ......
TMEM238 . . . . . . . . ......
RPL28 17 7 6 16 68 19 16 18 ......
UBE2S . . . . . . . 1 ......
ISOC2 . . . . . . . . ......
ZNF628 . . . . . . . . ......
NAT14 . . . . . . . . ......
SSC5D . . . . . . . . ......
ZNF579 . . . . . . . 1 ......
FIZ1 . . . . . . . . ......
ZNF524 . . 1 . 1 . . . ......
ZNF865 . . . . . . . . ......
ZNF784 . . . . . . . . ......
ZNF580 . . . . . . . . ......
ZNF581 . . . . 1 . . . ......
CCDC106 . . . . . . . . ......
U2AF2 . . . 1 . . . . ......
CTD-2537I9.12 . . . . . . . . ......
EPN1 . . . . 1 . . 1 ......
ZNF787 . . . . . . . . ......
ZNF444 . . . 1 . 1 . . ......
ZSCAN5A . . . . . . . . ......
AC006116.20 . . . . . . . . ......
ZNF582 . . . . . . . . ......
ZNF582-AS1 . . . . . . . . ......
ZNF583 . . . . . . . . ......
ZNF667 . . . . . . . . ......
ZNF667-AS1 . . . . . 1 . . ......
ZFP28 . . . . . . . . ......
ZNF470 . . . . . . . . ......
ZNF71 . . . . . . . . ......
AC007228.5 . . . . . . . . ......
AC007228.9 . 1 . . . . . . ......
ZNF264 . . . . . . . . ......
AURKC . . . . . . . . ......
ZNF805 . . . . . . . . ......
CTC-444N24.6 . . . . . . . . ......
ZNF460 . . . . . . . . ......
CTC-444N24.11 . . . . . . . . ......
ZNF543 . . . . . . . . ......
ZNF304 . . . . . . . . ......
ZNF547 . . . . . . . . ......
TRAPPC2P1 1 . . . . . . . ......
ZNF548 . . . . . . . . ......
ZNF17 . . . . . . . . ......
ZNF749 . . . . . . . . ......
ZNF772 . . . . . . . . ......
ZNF419 . . . . . 1 . . ......
ZNF773 . . . . . . . . ......
ZNF549 . . . . . . . . ......
ZNF550 . . . . . . . . ......
ZNF416 . . . . . . . . ......
ZIK1 . . . . . . . . ......
ZNF530 . . . . . . . . ......
ZNF134 . . . . . . . . ......
ZNF211 . . . . . . . . ......
ZNF551 . . . . . . . . ......
ZNF154 . . . . . . . . ......
ZNF671 . . . . . . . . ......
ZNF776 . . . . . . . . ......
ZNF586 . . . . . . . . ......
ZNF552 . . . . . . . . ......
ZNF587B . . . . . . . . ......
ZNF814 . . . . . . . . ......
ZNF587 . . . . . . . . ......
ZNF417 . . . . . . . . ......
ZNF418 . . . . . . . . ......
ZNF256 . . . . . . . . ......
C19orf18 . . . . . . . . ......
ZNF606 . . . . . . . . ......
CTD-2368P22.1 . . . . . . . . ......
ZNF135 . . . . . . . . ......
ZSCAN18 . . . . . . . . ......
ZNF329 . . . . . . . . ......
ZNF274 . . . . . . . . ......
ZNF544 . . . . . . . . ......
CTD-3138B18.5 . . . . . . . . ......
AC010642.1 . . . . 1 . . 1 ......
ZSCAN22 . . . . . . . . ......
A1BG . . . . . . . . ......
A1BG-AS1 . . . . . . . . ......
ZNF837 . . . . . . . . ......
RPS5 6 14 6 35 16 22 8 7 ......
ZNF584 . . . . . . . . ......
CTD-2619J13.14 . . . . . . . . ......
ZNF324B . . . . . . . . ......
ZNF324 . . . . . . . . ......
ZNF446 . . . . . . . . ......
SLC27A5 . . . . . . . . ......
ZBTB45 . . . . . . . . ......
TRIM28 . . . . 1 . . . ......
CHMP2A 1 1 . 1 . . . . ......
UBE2M . . . . . . . . ......
AC016629.8 . . . . 2 . . . ......
MZF1 . . . . . . . . ......
RPS4Y1 1 3 3 14 1 5 7 1 ......
ZFY . . . . . . . . ......
TTTY15 . . . . . . . . ......
USP9Y . . . . . . . . ......
DDX3Y . . . . . . . . ......
UTY . . . . . . . . ......
TMSB4Y . . . . . . . . ......
NLGN4Y . . . . . . . . ......
TTTY14 . . . . . . . . ......
KDM5D . . . . . . . . ......
EIF1AY . . . . . . 2 . ......
RPS4Y2 . . . . . . . . ......
TPTEP1 . . . . . . . 1 ......
IL17RA . . . . . . . . ......
CECR6 . . . . . . . . ......
CECR5 . . . . . . . 1 ......
CECR5-AS1 . . . . . . . . ......
CECR1 . . . . . . . . ......
ATP6V1E1 . . . 1 1 . . . ......
BCL2L13 . . . . . . . . ......
BID . . . . . . . 4 ......
LINC00528 . . . . . . . . ......
MICAL3 . . . . . . . . ......
XXbac-B476C20.9 . . . . . . . . ......
PEX26 . . . . . . . . ......
TUBA8 . . . . . . . . ......
USP18 . . . . . . . . ......
DGCR6 . . . . . . . . ......
DGCR2 . . . . . . . . ......
DGCR14 . . . . . . . . ......
AC004463.6 . . . . . . . . ......
SLC25A1 . . . . . . . . ......
CLTCL1 . . . . . . . . ......
HIRA . . . . . . . 1 ......
C22orf39 . . . . . . . . ......
MRPL40 . . . . . . . . ......
UFD1L . . . 1 . 2 . . ......
CDC45 . . . . . . . . ......
CLDN5 . . . . . . . . ......
SEPT5 . . . . . . . . ......
GNB1L . . . . . . . . ......
C22orf29 . . . . . . . . ......
TXNRD2 . . . . . . . . ......
COMT 1 . . . . . . . ......
ARVCF 1 . . . . . . . ......
TANGO2 . . . . . . . . ......
AC006547.15 . . . . . . . . ......
DGCR8 . . . . . . . . ......
TRMT2A . . . . . 1 . . ......
RANBP1 1 . . . . . 2 . ......
ZDHHC8 . . . . . . . . ......
RTN4R . . . . . . . . ......
DGCR6L . . . 1 . . . . ......
AC023490.1 . . . . . . . . ......
ZNF74 . . . . . . . . ......
SCARF2 . . . . . . . . ......
KLHL22 . . . . . . . . ......
MED15 . . . . . . . . ......
PI4KA . . . . . . . . ......
SNAP29 . . . . 2 . . . ......
CRKL . . . . . . . . ......
XXbac-B135H6.15 . . . . . . . . ......
AIFM3 . . . . . . . . ......
LZTR1 . . . . . . . . ......
XXbac-B135H6.18 . . . . . . . . ......
THAP7 . . . 2 1 . . . ......
THAP7-AS1 . . . . . . . . ......
HIC2 . . . . . . . . ......
TMEM191C . . . . . . . . ......
UBE2L3 . 1 . 1 1 . 1 . ......
YDJC . . . 1 . . . . ......
SDF2L1 . . . . . . . . ......
PPIL2 . . . . . . . . ......
YPEL1 . . . . . . . . ......
MAPK1 . . . . . . . . ......
PPM1F . . . . . . . . ......
LL22NC03-86G7.1 . . . . . . . . ......
TOP3B . . . . . . . . ......
VPREB1 . . . . . . . . ......
LL22NC03-2H8.5 . . . . . . . . ......
ZNF280B . . . . . . . . ......
IGLL5 . . . . . . . . ......
GNAZ . . . . . . . . ......
BCR . . . . . . . . ......
C22orf43 . . . . . . . . ......
RGL4 . . . . . . . . ......
ZNF70 . . . . . . . . ......
VPREB3 . 1 . . . . . . ......
C22orf15 . . . . . . . . ......
CHCHD10 . . . 1 3 . . 1 ......
MMP11 . . . . . . . . ......
SMARCB1 . . . . . 1 . 3 ......
DERL3 . . . . . . . . ......
SLC2A11 . . . . . . . . ......
MIF . 1 1 1 1 . 1 . ......
AP000350.4 . . . . . . . . ......
DDTL . . . . . . . . ......
DDT 1 . . . . . . . ......
GSTT1 . . . . . . . . ......
CABIN1 . . . . . . . . ......
SPECC1L . . . . . . . . ......
ADORA2A . . . . . . . . ......
ADORA2A-AS1 . . . . . . . . ......
GUCD1 . . . . . . . . ......
SNRPD3 . . . . 1 . . . ......
GGT1 . . . . . . . . ......
SGSM1 . . . . . . . . ......
KIAA1671 . . . . . . . . ......
LRP5L . . . . . . . . ......
CTA-407F11.8 . . . . . . . . ......
ADRBK2 . . . . . . . . ......
SEZ6L . . . . . . . . ......
ASPHD2 . . . . . . . . ......
HPS4 . . . . . . . . ......
SRRD . . . . . . . . ......
TFIP11 . . . . . . . . ......
CTA-445C9.14 . . . . . . . . ......
CTA-445C9.15 . . . . . . . . ......
TPST2 . . . . . . . . ......
MIAT . . . . . . . . ......
CTA-373H7.7 . . . . . . . . ......
CTA-211A9.5 . . . . . . . . ......
PITPNB . . . . . . . . ......
TTC28 . . . . . . . . ......
CHEK2 . . . . . . . . ......
HSCB . . . . . . . . ......
CCDC117 . . . . 1 . . . ......
XBP1 . 1 1 . . 2 2 1 ......
CTA-292E10.6 . . . . . . . . ......
RHBDD3 . . . . . . . . ......
EWSR1 . 1 . 1 . . 1 . ......
GAS2L1 . . . . . . . . ......
AP1B1 . . . . . . . . ......
RFPL1S . . . . . . . . ......
NEFH . . . . . . . . ......
THOC5 . . . . . . . . ......
NIPSNAP1 . . . . . . . . ......
NF2 . . . . . . . . ......
CABP7 . . . . . . . . ......
ZMAT5 . . . . 1 . . . ......
UQCR10 1 . . 1 1 1 1 . ......
ASCC2 . . . . . . . . ......
MTMR3 . . . . . . . . ......
OSM . . . . . . . . ......
GATSL3 . . . . . . . . ......
TBC1D10A . . . . . . 1 . ......
SF3A1 . . . . 1 . . . ......
CCDC157 . . . . . . . . ......
SEC14L2 . . . . . . . . ......
MTFP1 . . . . 1 . . . ......
RP4-539M6.22 . . . . . . . . ......
PES1 . . . . . . . . ......
TCN2 . . . . . . . . ......
SLC35E4 . . . . . . . . ......
DUSP18 . . . . . . . . ......
MORC2-AS1 . . . . . . . . ......
MORC2 . . . . . . . . ......
TUG1 . . . . . . . . ......
SMTN . . . . . . . . ......
SELM . 1 . . . . . . ......
RNF185 . . . . . . . . ......
LIMK2 . . . . . . . . ......
PIK3IP1 . . 1 . 2 5 . . ......
RP3-400N23.6 . . . . . . . . ......
PATZ1 . 1 . . . . . . ......
DRG1 . . . . . . . . ......
EIF4ENIF1 . . . . . . . . ......
SFI1 . . . . 1 . . . ......
PISD . . . . . . . . ......
PRR14L . . . . . . . . ......
DEPDC5 . . . . . . . . ......
YWHAH . 1 . . . . 1 . ......
RFPL2 . . . . . . . . ......
RTCB . . . . 1 . . 1 ......
FBXO7 . . . . . . . . ......
LARGE . . . . . . . . ......
HMGXB4 . 2 . . . . . . ......
RP3-510H16.3 . . . . . . . . ......
TOM1 . . . . 1 . . . ......
HMOX1 . . . . . . . . ......
MCM5 . . . . . . . . ......
APOL6 . . . . . . . . ......
RBFOX2 . . . . . . . . ......
APOL3 . . 1 . . . . . ......
APOL2 . . . . . . 1 1 ......
APOL1 . . . . . . . . ......
MYH9 . . . . 1 1 . 2 ......
TXN2 . 1 . . . 1 . . ......
FOXRED2 . . . . . . . . ......
EIF3D . 1 . 1 1 1 1 . ......
IFT27 . . . . . . . . ......
PVALB . . . . . . . . ......
NCF4 . . . . . . 1 . ......
CSF2RB . . . . . . . . ......
TST . . . . . . . . ......
MPST 1 . . . . . . . ......
KCTD17 . . . . . . . . ......
IL2RB . . . . . . . . ......
C1QTNF6 . . . . . . . . ......
SSTR3 . . . . . . . . ......
RAC2 . . 1 5 4 . . 2 ......
CYTH4 . . . . . . . 2 ......
MFNG . . . . . . . 1 ......
CDC42EP1 . . . . . . . . ......
LGALS2 . . . . . . . 1 ......
GGA1 1 . 1 . . . . . ......
SH3BP1 . . . 1 1 . . . ......
LGALS1 7 . . . 34 . . 17 ......
NOL12 1 . . . . . . . ......
TRIOBP . . . . . . . . ......
H1F0 . . . . . . . . ......
GCAT . . . . . . . . ......
ANKRD54 . . . . . 1 . . ......
EIF3L 1 . 4 3 2 14 1 2 ......
MICALL1 . . . . . . . . ......
POLR2F 1 . . . . . . 2 ......
PICK1 . . . . . . . . ......
PLA2G6 . . . . . . . . ......
MAFF . . . . . . . . ......
TMEM184B . . . . . . . . ......
CSNK1E . . . . . . . . ......
DDX17 1 . . . . . . . ......
DMC1 . . . . . . . . ......
CBY1 . . . . . . . . ......
TOMM22 1 . . . . . . . ......
JOSD1 . . . . . . . . ......
GTPBP1 . . . . . 1 . . ......
SUN2 . 1 . . . . . 1 ......
RP3-508I15.19 . . . . . . . . ......
RP3-508I15.21 . . . . . . . . ......
RP3-508I15.22 . . . . . . . . ......
DNAL4 . . . . . . . . ......
NPTXR . . . . . . . . ......
CBX6 . . . . . . . . ......
APOBEC3A . . . . . . . . ......
APOBEC3B . . . . . . . . ......
APOBEC3C . . . . . . . . ......
APOBEC3D . . . . . . . . ......
APOBEC3F . . . . . . . . ......
APOBEC3G . . . . 1 . . . ......
APOBEC3H . . . . . . . . ......
CBX7 . . . . . . . . ......
PDGFB . . . . . . . . ......
RPL3 21 23 21 31 28 22 13 16 ......
SYNGR1 . . . . . . . . ......
TAB1 . . . . . . . . ......
MIEF1 . . . . . . . . ......
ATF4 1 . . 1 1 . 2 . ......
RPS19BP1 . . . . . . . 1 ......
CACNA1I . . . . . . . . ......
GRAP2 . . . 1 . . . . ......
TNRC6B . . . . 1 . . . ......
ADSL . . . . . . . . ......
SGSM3 . . . . . . . . ......
RP5-1042K10.10 . . . . . . . . ......
MKL1 1 . . . . . 1 1 ......
SLC25A17 . . . . . . . . ......
ST13 . . 2 1 2 2 . 3 ......
XPNPEP3 . . . . 1 . . . ......
RBX1 1 . . . 3 . . 3 ......
EP300 . . . . . . . . ......
L3MBTL2 . . . . . . . . ......
RANGAP1 . . . . . . 1 . ......
ZC3H7B . . . . . . . . ......
TEF . . . . . . . . ......
TOB2 . 1 . . . . . . ......
PHF5A . . . . . . 1 . ......
ACO2 . . . . . . . . ......
POLR3H . . . . 1 . . . ......
PMM1 . . . . . . . . ......
DESI1 . . . . . . . . ......
XRCC6 . . . . . . 1 . ......
NHP2L1 . 2 . 1 . . . 3 ......
C22orf46 . . . . . . . . ......
MEI1 . . . . . . . . ......
CCDC134 . . . . . . . . ......
RP5-821D11.7 . . . . . . . . ......
SREBF2 . . . . . . . . ......
CTA-250D10.23 . 1 . . . . . . ......
TNFRSF13C . 2 . . . . . . ......
CENPM . . . . . . . . ......
WBP2NL . . . . . . . . ......
NAGA . . . . . . . . ......
FAM109B . . . . . . . . ......
SMDT1 1 1 . . 1 1 1 1 ......
NDUFA6 . . . . . . . . ......
CYP2D6 . . . . . . . . ......
TCF20 . . . . . . . . ......
NFAM1 . . . . 1 . . . ......
RRP7A 1 . . . . . . . ......
SERHL2 . . . . . . . . ......
POLDIP3 1 . . . . . . . ......
RNU12 . 1 . . 1 . . . ......
CYB5R3 . . . . . . . . ......
ATP5L2 . . . . . . . 1 ......
ARFGAP3 . . . . . . . . ......
PACSIN2 . . . . 2 . . . ......
TTLL1 . . . . . . . . ......
BIK . . . . . . . . ......
MCAT . 1 . . . . . . ......
TSPO 5 . . . 1 1 . . ......
TTLL12 . . . . . . . . ......
SAMM50 . . . . . . . . ......
PARVB . . . . . . . . ......
PARVG . . . 1 . . . . ......
LDOC1L . . . . . . . . ......
PRR5 . . . . 1 . . . ......
CTA-217C2.1 . . . . . . . . ......
NUP50 . . . . . . . . ......
KIAA0930 . . . . . . . . ......
UPK3A . . . . . . . . ......
FAM118A . . . . . . . . ......
ATXN10 . . . . . . . . ......
CITF22-92A6.1 . . . . . . . . ......
C22orf26 . . . . . . . . ......
RP6-109B7.3 . . . . . . . . ......
FLJ27365 . . . . . . . . ......
PPARA . . . . . . . . ......
CDPF1 . . . . 1 . . . ......
TTC38 . . . . . . . . ......
GTSE1 . . . . . . . . ......
TRMU . . . . . . . . ......
CELSR1 . . . . . . . . ......
GRAMD4 . . . . . . . . ......
CERK . . . . . . . . ......
CTA-29F11.1 . . . . . . . . ......
TBC1D22A . . . . . . . . ......
LL22NC03-75H12.2 . . . . . . . . ......
C22orf34 . . . . . . . . ......
RP1-29C18.10 . . . . . . . . ......
BRD1 . . . . . . . . ......
ZBED4 . . . . . . . . ......
ALG12 . . . . . . . 1 ......
CRELD2 . 1 . . 1 . . . ......
PIM3 . . . . . . . . ......
MLC1 . . . . . . . . ......
MOV10L1 . . . . . . . . ......
TRABD 1 . 1 . . . . . ......
RP3-402G11.25 . . . . . . . . ......
RP3-402G11.26 . . . . . . . . ......
SELO . . . . . . . . ......
TUBGCP6 . . . . . . . . ......
HDAC10 1 . . . . . . . ......
MAPK11 . . . . . . . . ......
PLXNB2 . . . . . . . . ......
DENND6B . . . . . . . . ......
PPP6R2 . . . . . . 1 . ......
SBF1 . . . . . . 1 . ......
LMF2 . . . . . . 1 . ......
NCAPH2 . . . 1 . . . . ......
SCO2 1 . . . . . . 2 ......
CTA-384D8.36 . . . . . . . . ......
TYMP 5 . . . 2 . 1 10 ......
ODF3B . . . . . . . . ......
CTA-384D8.35 . . . . . . . . ......
CTA-384D8.34 . . . . . . . . ......
KLHDC7B . . . . . . . . ......
SYCE3 . . . . . . . . ......
CPT1B . . . . . . . . ......
CHKB . . . . . . . . ......
CHKB-AS1 . . . . . . . . ......
ARSA 1 . . . . . . . ......
RABL2B . . . . . . . . ......
RBM11 . . . . . . . . ......
HSPA13 . . . . . 1 . . ......
SAMSN1 . . . . . . . . ......
AF127936.7 . . . . . . . . ......
NRIP1 . . . . . . . . ......
USP25 . . . . . . . . ......
BTG3 . . . . . 1 . . ......
C21orf91 . . . . . . . . ......
AP000476.1 . . . . . . . . ......
MIR155HG . . . . . . . . ......
MRPL39 . 1 . . . . . . ......
ATP5J . 1 . . . . . . ......
GABPA . . . . . . . . ......
APP . . . . . . . . ......
AF131217.1 . . . . . . . . ......
N6AMT1 . . . . . . . . ......
LTN1 . 1 . . . . . . ......
RWDD2B . . . . . . . . ......
RP1-100J12.1 . . . . . . . . ......
USP16 . 1 . . . . 1 1 ......
CCT8 . . 1 . 1 1 1 1 ......
MAP3K7CL . . . . . . . . ......
BACH1 . . . . . . . . ......
TIAM1 . . . . . . . . ......
SOD1 . . . 1 1 . 1 2 ......
AP000254.8 . . . . . . . . ......
SCAF4 . . . . . . . . ......
MIS18A . . . . . . . . ......
URB1 . . . . . . . . ......
C21orf119 . . . . . . . . ......
EVA1C . . . . . . . . ......
TCP10L . . . . . . . . ......
C21orf59 . . . . . . . . ......
SYNJ1 . . . . . . . . ......
PAXBP1-AS1 . . . . . . . . ......
PAXBP1 . . . . . . . . ......
C21orf49 . . . . . . . . ......
OLIG1 . . . . . . . . ......
IFNAR2 . . . . 1 . . . ......
IL10RB-AS1 . . . . . . . . ......
IL10RB . . . . . 1 . . ......
IFNAR1 . . . 1 . . . . ......
IFNGR2 . . . . 1 . . 1 ......
TMEM50B . . . . . . . . ......
DNAJC28 . . . . . . . . ......
GART . . . . . . . . ......
SON . 2 1 . 1 2 . 1 ......
DONSON . . . . . . . . ......
CRYZL1 . . 1 . . . . . ......
ITSN1 . . . . . . . . ......
ATP5O 2 . . 1 1 1 . . ......
LINC00649 . . . . . . . . ......
MRPS6 . . 2 . . 1 . . ......
SMIM11 . . . . . . . . ......
KCNE1 . . . . . . . . ......
RCAN1 . . . . . . . . ......
RUNX1 . . . . . . . . ......
SETD4 . . . . . . . . ......
CBR1 2 . . . 1 . 1 1 ......
CBR3 . . . . . . 1 . ......
DOPEY2 . . . . . . . . ......
AP000692.10 . . . . . . . . ......
MORC3 . . . 1 . . . . ......
CHAF1B . . . . . . . . ......
HLCS . . . . . . . . ......
PIGP . . . . . . . 1 ......
TTC3 . . 1 . . 1 . . ......
DSCR9 . . . . . . . . ......
DSCR3 . . . . . . . . ......
AP001412.1 . . . . . . . . ......
DYRK1A . . . . . . . . ......
ETS2 . . . . . . . . ......
PSMG1 . . . . . . . . ......
BRWD1 . . . . . . 1 . ......
HMGN1 . 3 . . . . . . ......
WRB . . . . . . . . ......
BACE2 . . . . . . . . ......
FAM3B . . . . . . . . ......
MX2 . . . . . . 1 . ......
MX1 1 1 . . 1 . 2 . ......
AP001610.5 . . . . . . . . ......
PRDM15 . . . . . . . . ......
C2CD2 . . . . . . . . ......
ZBTB21 . . . . . . . . ......
ABCG1 . . . . . . . . ......
TMPRSS3 . . . . . . . . ......
UBASH3A . . . . . . . . ......
SLC37A1 . . . . . . . . ......
PDE9A . . . . . . . . ......
WDR4 . . . . . . . . ......
NDUFV3 . . . . . . . 1 ......
PKNOX1 . 1 . . . . . . ......
U2AF1 . 1 . 1 1 . . 1 ......
AP001046.5 . . . . . . . . ......
AP001046.6 . . . . . . . . ......
SIK1 . . . . 1 . . . ......
HSF2BP . . . . . . . . ......
RRP1B . . . . 1 . . . ......
PDXK . . . . 1 . . 2 ......
CSTB 1 2 1 . 1 . . 3 ......
RRP1 . . . . . . . . ......
AP001053.11 . . . . . . . . ......
AGPAT3 . . . . . . . . ......
TRAPPC10 . . . . . . . . ......
PWP2 . . . . . . . . ......
C21orf33 . 1 . 1 . . . . ......
AP001055.6 . . . . . . . . ......
AP001058.3 . . . . . . . . ......
ICOSLG . . . . . . . . ......
AIRE . . . . . . . . ......
PFKL . . . . . . . . ......
C21orf2 . . . . . 1 . . ......
AP001062.7 . . . . . . . . ......
TRPM2 . . . . . . . . ......
LRRC3 . . . . . . . . ......
UBE2G2 . . . . . . . . ......
SUMO3 . . . 1 1 . . . ......
PTTG1IP . . . . . . . . ......
ITGB2 2 . . 2 1 . . 5 ......
ITGB2-AS1 . . . . . . . . ......
LL21NC02-1C16.2 . . . . . . . . ......
FAM207A . . . . . . . . ......
ADARB1 . . . . . . . . ......
POFUT2 . . . . . . . . ......
COL18A1 . . . . . . . . ......
SLC19A1 . . . . . . . . ......
PCBP3 . . . . . . . . ......
COL6A1 . . . . . . . . ......
COL6A2 . . . . . . . . ......
SPATC1L . . . . . 1 . . ......
LSS . . . . . 2 . . ......
MCM3AP-AS1 . . . . . . . . ......
MCM3AP . . . . . . . . ......
AP001469.9 . . . . . . . . ......
YBEY . . . 1 . . . 1 ......
C21orf58 . . . . . . . . ......
PCNT . . . . . . . . ......
DIP2A . . . . . . . . ......
S100B . . . 3 2 . . . ......
PRMT2 . . . . . 1 . . ......
MT-ND1 4 1 3 3 17 5 5 12 ......
MT-ND2 4 . 2 1 10 7 2 12 ......
MT-CO1 23 7 7 13 42 9 12 30 ......
MT-CO2 9 2 5 13 19 12 9 106 ......
MT-ATP8 . . . . . . . . ......
MT-ATP6 1 . . . 3 3 3 4 ......
MT-CO3 18 10 3 16 18 9 8 14 ......
MT-ND3 . . . . . . . . ......
MT-ND4L . . . . 1 . . . ......
MT-ND4 4 5 2 11 9 5 6 9 ......
MT-ND5 . . . 2 8 2 2 1 ......
MT-ND6 . . . . . . . . ......
MT-CYB 4 2 6 4 11 9 5 1 ......
AC145212.1 . . . . . . . . ......
AL592183.1 . . . . . . . . ......
AL354822.1 . . . . 1 . . . ......
PNRC2.1 . . . . . . . . ......
SRSF10.1 . . . . 1 . . . ......dim(pbmc3k_read_rds@assays$RAW$counts)[1] 13714 2698dim(pbmc3k_read_rds@assays$RNA$counts)[1] 13714 2698
sessionInfo()R version 4.4.1 (2024-06-14)
Platform: x86_64-pc-linux-gnu
Running under: Ubuntu 22.04.5 LTS
Matrix products: default
BLAS: /usr/lib/x86_64-linux-gnu/openblas-pthread/libblas.so.3
LAPACK: /usr/lib/x86_64-linux-gnu/openblas-pthread/libopenblasp-r0.3.20.so; LAPACK version 3.10.0
locale:
[1] LC_CTYPE=en_US.UTF-8 LC_NUMERIC=C
[3] LC_TIME=en_US.UTF-8 LC_COLLATE=en_US.UTF-8
[5] LC_MONETARY=en_US.UTF-8 LC_MESSAGES=en_US.UTF-8
[7] LC_PAPER=en_US.UTF-8 LC_NAME=C
[9] LC_ADDRESS=C LC_TELEPHONE=C
[11] LC_MEASUREMENT=en_US.UTF-8 LC_IDENTIFICATION=C
time zone: Etc/UTC
tzcode source: system (glibc)
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] Seurat_5.2.1 SeuratObject_5.0.2 sp_2.2-0 BPCells_0.3.0
[5] workflowr_1.7.1
loaded via a namespace (and not attached):
[1] RColorBrewer_1.1-3 rstudioapi_0.17.1 jsonlite_1.8.9
[4] magrittr_2.0.3 ggbeeswarm_0.7.2 spatstat.utils_3.1-2
[7] farver_2.1.2 rmarkdown_2.28 fs_1.6.4
[10] zlibbioc_1.52.0 vctrs_0.6.5 ROCR_1.0-11
[13] spatstat.explore_3.3-4 htmltools_0.5.8.1 sass_0.4.9
[16] sctransform_0.4.1 parallelly_1.38.0 KernSmooth_2.23-24
[19] bslib_0.8.0 htmlwidgets_1.6.4 ica_1.0-3
[22] plyr_1.8.9 plotly_4.10.4 zoo_1.8-13
[25] cachem_1.1.0 whisker_0.4.1 igraph_2.1.4
[28] mime_0.12 lifecycle_1.0.4 pkgconfig_2.0.3
[31] Matrix_1.7-0 R6_2.5.1 fastmap_1.2.0
[34] GenomeInfoDbData_1.2.13 MatrixGenerics_1.18.1 fitdistrplus_1.2-2
[37] future_1.34.0 shiny_1.10.0 digest_0.6.37
[40] colorspace_2.1-1 patchwork_1.3.0 S4Vectors_0.44.0
[43] ps_1.8.1 rprojroot_2.0.4 tensor_1.5
[46] RSpectra_0.16-2 irlba_2.3.5.1 GenomicRanges_1.58.0
[49] labeling_0.4.3 progressr_0.15.0 spatstat.sparse_3.1-0
[52] polyclip_1.10-7 httr_1.4.7 abind_1.4-8
[55] compiler_4.4.1 withr_3.0.2 fastDummies_1.7.5
[58] highr_0.11 MASS_7.3-60.2 tools_4.4.1
[61] vipor_0.4.7 lmtest_0.9-40 beeswarm_0.4.0
[64] httpuv_1.6.15 future.apply_1.11.3 goftest_1.2-3
[67] glue_1.8.0 callr_3.7.6 nlme_3.1-164
[70] promises_1.3.2 grid_4.4.1 Rtsne_0.17
[73] getPass_0.2-4 cluster_2.1.6 reshape2_1.4.4
[76] generics_0.1.3 gtable_0.3.6 spatstat.data_3.1-4
[79] tidyr_1.3.1 data.table_1.16.2 XVector_0.46.0
[82] BiocGenerics_0.52.0 spatstat.geom_3.3-5 RcppAnnoy_0.0.22
[85] ggrepel_0.9.6 RANN_2.6.2 pillar_1.10.1
[88] stringr_1.5.1 spam_2.11-1 RcppHNSW_0.6.0
[91] later_1.3.2 splines_4.4.1 dplyr_1.1.4
[94] lattice_0.22-6 deldir_2.0-4 survival_3.6-4
[97] tidyselect_1.2.1 miniUI_0.1.1.1 pbapply_1.7-2
[100] knitr_1.48 git2r_0.35.0 gridExtra_2.3
[103] IRanges_2.40.1 scattermore_1.2 stats4_4.4.1
[106] xfun_0.48 matrixStats_1.5.0 stringi_1.8.4
[109] UCSC.utils_1.2.0 lazyeval_0.2.2 yaml_2.3.10
[112] evaluate_1.0.1 codetools_0.2-20 tibble_3.2.1
[115] cli_3.6.3 uwot_0.2.3 xtable_1.8-4
[118] reticulate_1.41.0 munsell_0.5.1 processx_3.8.4
[121] jquerylib_0.1.4 Rcpp_1.0.13 GenomeInfoDb_1.42.3
[124] spatstat.random_3.3-2 globals_0.16.3 png_0.1-8
[127] ggrastr_1.0.2 spatstat.univar_3.1-2 parallel_4.4.1
[130] ggplot2_3.5.1 dotCall64_1.2 listenv_0.9.1
[133] viridisLite_0.4.2 scales_1.3.0 ggridges_0.5.6
[136] purrr_1.0.2 rlang_1.1.4 cowplot_1.1.3