Comandos Basicos
Miguel Tripp
Last updated: 2021-07-24
Checks: 7 0
Knit directory: 2021/
This reproducible R Markdown analysis was created with workflowr (version 1.6.2). The Checks tab describes the reproducibility checks that were applied when the results were created. The Past versions tab lists the development history.
Great! Since the R Markdown file has been committed to the Git repository, you know the exact version of the code that produced these results.
Great job! The global environment was empty. Objects defined in the global environment can affect the analysis in your R Markdown file in unknown ways. For reproduciblity it’s best to always run the code in an empty environment.
The command set.seed(20210412) was run prior to running the code in the R Markdown file. Setting a seed ensures that any results that rely on randomness, e.g. subsampling or permutations, are reproducible.
Great job! Recording the operating system, R version, and package versions is critical for reproducibility.
Nice! There were no cached chunks for this analysis, so you can be confident that you successfully produced the results during this run.
Great job! Using relative paths to the files within your workflowr project makes it easier to run your code on other machines.
Great! You are using Git for version control. Tracking code development and connecting the code version to the results is critical for reproducibility.
The results in this page were generated with repository version 4f1cff5. See the Past versions tab to see a history of the changes made to the R Markdown and HTML files.
Note that you need to be careful to ensure that all relevant files for the analysis have been committed to Git prior to generating the results (you can use wflow_publish or wflow_git_commit). workflowr only checks the R Markdown file, but you know if there are other scripts or data files that it depends on. Below is the status of the Git repository when the results were generated:
Ignored files:
Ignored: .Rhistory
Ignored: .Rproj.user/
Untracked files:
Untracked: Curso_Bioestadistica_MTripp_cuatriII.docx
Untracked: Curso_Bioestadistica_MTripp_cuatriII.pdf
Untracked: Diapositivas/
Untracked: Prueba_markdown.Rmd
Untracked: Prueba_markdown.pdf
Untracked: README.html
Untracked: Resources/
Untracked: Tarea_Tstudent.Rmd
Untracked: Tarea_Tstudent.docx
Untracked: Tarea_Tstudent.html
Untracked: Tarea_Tstudent.pdf
Untracked: analysis/Clase13_noParam.Rmd
Untracked: analysis/images/
Untracked: code/tarea_macrograd.R
Untracked: data/CS_subset.csv
Untracked: data/Consumo_oxigeno_wide.csv
Untracked: data/Darwin_esp.csv
Untracked: data/Data_enzimas_Experimento1.txt
Untracked: data/Data_enzimas_Experimento2.txt
Untracked: data/Data_enzimas_Experimento3.txt
Untracked: data/Data_enzimas_Experimento4.txt
Untracked: data/DownloadFestival(No Outlier).dat
Untracked: data/Festival.csv
Untracked: data/Hful_metabolitos_ver2.csv
Untracked: data/Longitud_noParam.csv
Untracked: data/LungCapData.txt
Untracked: data/LungCapDataEsp.csv
Untracked: data/RExam.dat
Untracked: data/Rexamendat.csv
Untracked: data/Tabla1_Muestreo.txt
Untracked: data/Transcriptome_Anotacion.csv
Untracked: data/Transcriptome_DGE.csv
Untracked: data/Vinogradov_2004_Titanic.tab
Untracked: data/Vinogradov_2004_Titanic.tab.csv
Untracked: data/data_tukey.txt
Untracked: data/datasets_Pokemon.csv
Untracked: data/datasets_Pokemon.xls
Untracked: data/exp_macrogard_growth.tab
Untracked: data/exp_macrogard_rna-dna.tab
Untracked: data/fertilizantes_luz.csv
Untracked: data/gatos_sueno.csv
Untracked: data/macrogard_crecimiento.csv
Untracked: data/penguins_size.csv
Untracked: data/pokemon_extended.csv
Untracked: output/Plot_all_penguins.pdf
Untracked: output/Plot_all_penguins.tiff
Untracked: output/graficos/
Note that any generated files, e.g. HTML, png, CSS, etc., are not included in this status report because it is ok for generated content to have uncommitted changes.
These are the previous versions of the repository in which changes were made to the R Markdown (analysis/Clase1_Basicos.Rmd) and HTML (docs/Clase1_Basicos.html) files. If you’ve configured a remote Git repository (see ?wflow_git_remote), click on the hyperlinks in the table below to view the files as they were in that past version.
| File | Version | Author | Date | Message |
|---|---|---|---|---|
| Rmd | 4f1cff5 | Miguel Tripp | 2021-07-24 | workflowr::wflow_publish(c(“analysis/index.Rmd”, “analysis/about.Rmd”, |
| html | 9d09420 | Miguel Tripp | 2021-07-22 | Build site. |
| html | 0f7eb2d | Miguel Tripp | 2021-07-12 | Build site. |
| html | 82e4deb | Miguel Tripp | 2021-07-08 | Build site. |
| html | bc7c1d7 | Miguel Tripp | 2021-07-07 | Build site. |
| html | 01ac301 | Miguel Tripp | 2021-07-04 | Build site. |
| html | 392444f | Miguel Tripp | 2021-07-04 | Build site. |
| html | c188ae8 | Miguel Tripp | 2021-06-29 | Build site. |
| html | 1136768 | Miguel Tripp | 2021-06-28 | Build site. |
| html | a33d4bb | Miguel Tripp | 2021-06-23 | Build site. |
| html | 209299f | Miguel Tripp | 2021-06-21 | Build site. |
| html | d025507 | Miguel Tripp | 2021-06-17 | Build site. |
| html | fb9e91e | Miguel Tripp | 2021-06-16 | Build site. |
| html | c5dfe6a | Miguel Tripp | 2021-06-14 | Build site. |
| html | 2352c47 | Miguel Tripp | 2021-06-12 | Build site. |
| html | 99c3644 | Miguel Tripp | 2021-06-05 | Build site. |
| html | fbd7857 | Miguel Tripp | 2021-06-05 | Build site. |
| html | 87a646f | Miguel Tripp | 2021-06-02 | Build site. |
| html | 47a7147 | Miguel Tripp | 2021-05-31 | Build site. |
| html | 95c661f | Miguel Tripp | 2021-05-31 | Build site. |
| html | 34660de | Miguel Tripp | 2021-05-24 | Build site. |
| html | da04854 | Miguel Tripp | 2021-05-24 | Build site. |
| html | 3321935 | Miguel Tripp | 2021-05-24 | Build site. |
| html | 3d12902 | Miguel Tripp | 2021-05-16 | Build site. |
| html | 20b3aab | Miguel Tripp | 2021-05-16 | Build site. |
| html | f20dcb1 | Miguel Tripp | 2021-05-16 | Build site. |
| html | b1ca7f3 | Miguel Tripp | 2021-05-16 | Build site. |
| html | b3002cc | Miguel Tripp | 2021-05-04 | Build site. |
| html | 2c60db3 | Miguel Tripp | 2021-05-04 | Build site. |
| html | 65fa0ee | Miguel Tripp | 2021-05-04 | Build site. |
| html | b3c3d89 | Miguel Tripp | 2021-04-26 | Build site. |
| html | 195c2e9 | Miguel Tripp | 2021-04-25 | Build site. |
| html | e322dac | Miguel Tripp | 2021-04-25 | Build site. |
| html | b237a5e | Miguel Tripp | 2021-04-22 | Build site. |
| html | 4c3c22d | Miguel Tripp | 2021-04-20 | Build site. |
| Rmd | 43a6b26 | Miguel Tripp | 2021-04-20 | Publish the initial files for myproject |
| html | 945bc0f | Miguel Tripp | 2021-04-18 | Build site. |
| html | 527e486 | Miguel Tripp | 2021-04-18 | Build site. |
| html | cddaa8a | Miguel Tripp | 2021-04-18 | Build site. |
| html | 02e6728 | Miguel Tripp | 2021-04-18 | Build site. |
| Rmd | 8c50c39 | Miguel Tripp | 2021-04-18 | Publish the initial files for myproject |
1 Introducción

Este curso es mi pequeña aportación para un mundo mejor donde no hay que pagar grandes sumas de dinero para hacer una ANOVA y donde los investigadores muestran sus resultados en otra cosa que no sea un gráfico de barras. Así que toma tu toalla favorita y empecemos.
2 Generalidades
En este primer capítulo vamos a revisar algunas funcionalidades básicas de R tales como crear objetos y vectores asi como revisar los diferentes tipos de estructuras de datos.
Antes de empezar, es importante mencionar algunas aspectos:
R es sensible a las mayúsculas y minúsculas, de manera que
Aes diferente queade la misma forma que la funciónanova()es diferente a la funciónAnova()El símbolo
#sirve para insertar un comentario en la línea de comando, de manera que todo lo que siga después de#no se ejecutara. Es importante incluir comentarios como parte de las buenas practicas en el uso de R ya que le facilitará el trabajo a tus colaboradores y revisores saber que estas haciendo en casa paso y te salvará la vida cuando intentes correr tu script cinco años después de haberlo escritoEn la consola usualmente veremos el símbolo
>lo que la indica que la consola esta lista para recibir nuevas instrucciones. Si por el contrario vemos el símbolo+entonces nos esta indicando que no hemos terminado de completar correctamente una línea de código.Si despúes de ejecutar un comando la consola nos arroja un mensaje de advertencia
WARNINGsignifica que el comando se ejecutó pero hay algo inesperad que debe revisarse. Por el contrario si nos arroja un mensaje de errorERRORentonces hay algo mal en la línea de codigo y por lo tanto no se pudo ejecutar.
3 Uso de la línea de comando como calculadora
Podemos utilizar R para realizar operaciones básicas:
1 + 2 #suma[1] 34 - 3 #resta[1] 15 * 5 #multiplicación[1] 254 / 2 #división[1] 24 ^ 2 #Exponente[1] 164 ** 2 #Exponente[1] 16!! ¿por qué hay un [1] en cada resultado?
El resultado es un vector, aunque solo consista en un elemento. Por lo tanto, R indica que este el primer [1] elemento de un vector
Operadores en R:
| Operador | significado |
|---|---|
| * | Multiplicación |
| - | Resta |
| + | Suma |
| ^ | Elevar a potencia |
| / | División |
| < | Menor que |
| > | Mayor que |
| <= | Menor o igual que |
| >= | Mayor o igual que |
| == | Exactamente igual |
| != | Distinto |
4 Uso de objetos
Para crear un nuevo objeto se puede usar “=” o “<-”. Este nuevo objeto, se almacenará en la memoria y se puede usar para otras operaciones.
x <- 2 #asignar a la variable x el valor de 2
x
y = 3 #asignar a la variable y el valor de 3
y
#resultado de la suma x + y
x + y
z <- x + y#asiganr el resultado de x + y a la variable z
z
#uso de c() para incluir mas de un elemento
numeros <- c(1,2,3,4,5)
numeros
variables <- c(x,y,z)
variables
variables_mas <- variables + 1
variables_mas
variables_expo <- variables ^2
2 ^ variables -> expo_variables
variables_numero <- variables * numeros
#¿que pasara aqui?Las variables no solo puede contener números, sino caracteres, listas o incluso graficas
caracter <- "siete"
caracter[1] "siete"mix <- c(1,2,"tres")
mix[1] "1" "2" "tres"¿cual serial el resultado de multiplicar la variable mix x 3?
¿Cuales de estas formas de definir una variables es incorrecta?
mi.variable <-mi_variable <-mi variable <-mi-variable <-
5 Tipos y estructura de datos (Classes)
R tiene los siguientes tipos de datos basicos:
logical:valores lógicos, pueden ser TRUE o FALSEnumeric:números reales (1.0,2,3.1416, 3E-10)character:caracteres (letras y/o números). Delimitados por "" (“uno”, “Miguel”, “FALSO”)interger:valores enteros (1,2,3)NA:dato no disponibleNaN:no es un número (not a number)
Por otro lado, las estructuras de datos pueden ser de tipo vector, data.frame, matriz, arreglo o lista:
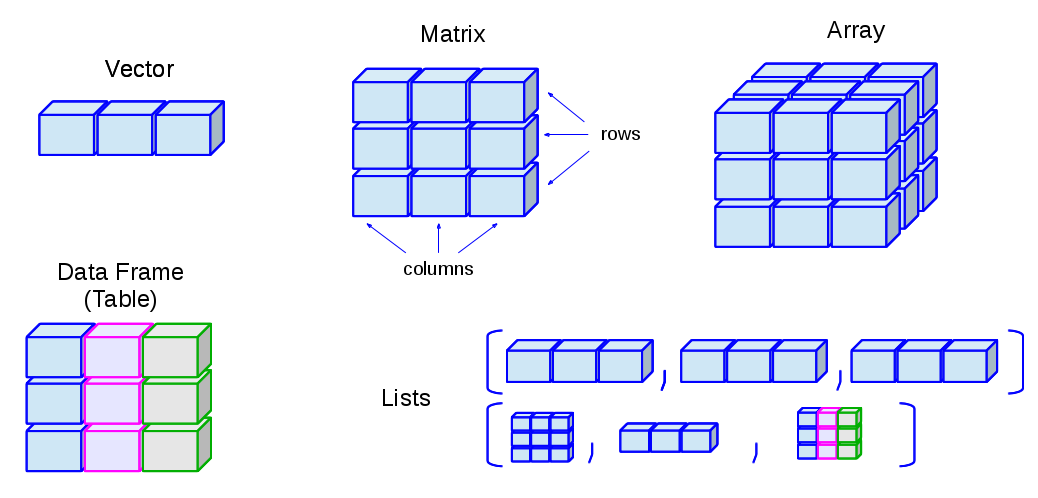
fuente: http://venus.ifca.unican.es/Rintro/_images/dataStructuresNew.png
Es importante resaltar que solamente los data.frame pueden contener distintos tipos de datos (números, caracteres, etc) mientras que el resto solo puede contener un tipo de dato (solo carácteres, solo números, etc).
Si no sabes que tipo de dato contiene una variable, se puede sar la funcions class()
class(numeros) [1] "numeric"class(caracter) [1] "character"class(numeros)[1] "numeric"class(mix)[1] "character"R utiliza funciones para realizar operaciones, por ejemplo class(). Para utilizar una función deben especificarse determiandos argumentos, los cuales se escriben dentro del parentesis.
Por ejemplo, en la función round(), la ayuda nos dice que debemos especificar round(x, digits = 0) donde x es el objecto (número o vector) y digits es el número de decimales.
5.1 Trabajando con vectores
Ejercicio:
Supon que tienes un cultivo de abulones y tienes que revisarlo todos los dias y contar los organismos muertos. Despues de una semana tienes el siguiente resultado:
- lunes: 4
- martes: 6
- miercoles: 0
- jueves: 10
- viernes: 12
- sabado: 8
- domingo: 4
1: crea un vector con los valores diarios 2: nombre cada valor con el dia de la semana usando la funcion names()
mortalidad <- c(4,6,0,10,12,8,4)
mortalidad[1] 4 6 0 10 12 8 4names(mortalidad) <- c("Lunes", "Martes", "Miercoles", "Jueves", "Viernes", "Sabado", "Domingo")
mortalidad Lunes Martes Miercoles Jueves Viernes Sabado Domingo
4 6 0 10 12 8 4 #nombrar la mortalidad con la funcion namesOperaciones basicas con vectores
| Función | significado |
|---|---|
| min() | Minimo |
| max() | Máximo |
| length() | longitud |
| range() | intervalo de valores |
| sort() | Ordena de menor a mayor |
| unique() | Valores unicos |
| mean() | Promedio |
| sum() | suma |
| which() | proporciona los indices TRUE |
Ejercicio: Contesta lo siguiente:
- ¿cual fue el valor minimo y el maximo?
- ¿cuantos abulones murieron esa semana?
6 Indices
Una tarea común al trabajar con vectores de datos es el de aislar uno o varios valores que cumplen con algún criterio.
Mostrar el primer valor del vector
mortalidad[1]Lunes
4 Mostrar todos los valores excepto el segundo
mortalidad[-2] Lunes Miercoles Jueves Viernes Sabado Domingo
4 0 10 12 8 4 Se puede obtener multiples series de elementos al mismo tiempo usando c()
mortalidad[c(1,2:4)] Lunes Martes Miercoles Jueves
4 6 0 10 o si por el contrario queremos eliminar estos elementos se usa c(-)
mortalidad[-c(1,2:4)]Viernes Sabado Domingo
12 8 4 Obtener los dias en donde la mortalidad fue mayor a 5
mortalidad > 5 Lunes Martes Miercoles Jueves Viernes Sabado Domingo
FALSE TRUE FALSE TRUE TRUE TRUE FALSE #Mostrar solo los dias donde la mortalidad fue > 5
mortalidad[mortalidad > 5] Martes Jueves Viernes Sabado
6 10 12 8 #Mediante una variable
mor_5 <- mortalidad > 5
mortalidad[mor_5] Martes Jueves Viernes Sabado
6 10 12 8 Ejercicio
Identificar:
- ¿En que dia se observó la menor mortalidad?
- ¿Que dias tuvieron una mortalidad igual a 12?
- ¿Que dias tuvieron una mortalidad menor al promedio?
7 Enlistar y borrar objectos
Las funciones ls()y objects()hacen lo mismo: enlistan los objetos que hemos definido en la sesión.
ls() [1] "caracter" "expo_variables" "mix" "mor_5"
[5] "mortalidad" "numeros" "variables" "variables_expo"
[9] "variables_mas" "variables_numero" "x" "y"
[13] "z" Y si queremos borrar objectos, se usa la funcion rm() Si queremos borrrar todos los objectos del ambiente, se ejecuta rm(list=ls()).
8 Trabajando con tablas
Ahora supongamos que tienes que revisar tus estanques por otra semana. Sin embargo, el jueves de la tercera semana no pudiste ir, por lo que no tienes valores para ese dia. Estos son los resultados.
- lunes: 16
- martes: 12
- miércoles: 8
- jueves: no hay dato
- viernes: 14
- sábado: 4
- domingo: 10
Ejercicio: Construye un vector para la semana 2 con el nombre semana2.
De que forma podríamos nombrar cada día de la semana en semana2?
9 Crear una tabla con data.frame
dia_sem <- c("Lunes", "Martes", "Miercoles", "Jueves", "Viernes", "Sabado", "Domingo")
# Indicamos row.names= NULL para que no se incluyan los nombres del vector como row.names
tabla <- data.frame(dia = dia_sem, semana1 = mortalidad, semana2 = semana2, row.names = NULL)
tabla dia semana1 semana2
1 Lunes 4 16
2 Martes 6 12
3 Miercoles 0 8
4 Jueves 10 NA
5 Viernes 12 14
6 Sabado 8 4
7 Domingo 4 10# Alternativamente, ejecutamos row.names = dia_sem para indicar los días de la semana como row.names
tabla <- data.frame(row.names = dia_sem, semana1 = mortalidad, semana2 = semana2)
tabla semana1 semana2
Lunes 4 16
Martes 6 12
Miercoles 0 8
Jueves 10 NA
Viernes 12 14
Sabado 8 4
Domingo 4 10¿Que tipo de dato es el objecto tabla?
Ejercicio: Para cada semana, busca el valor mínimo, máximo y promedio. Con estos valores crea un vector para cada semana y nombralos
Semana1_sumySemana2_sum.
Para esto es posible seguir dos aproximaciones:
- usando indices:
tabla[filas , columnas] - con el operado
$:tabla$columna
por ejemplo:
max(tabla$semana1)[1] 12min(tabla[,1])[1] 0mean(tabla$semana2, na.rm = TRUE)[1] 10.6666710 Estructura de una tabla
#muestra las primeras n filas de la tabla
head(tabla) semana1 semana2
Lunes 4 16
Martes 6 12
Miercoles 0 8
Jueves 10 NA
Viernes 12 14
Sabado 8 4#muestra el numero de filas
nrow(tabla)[1] 7#muestra el numero de columnas
ncol(tabla)[1] 2#muestra el nombre de las columnas
colnames(tabla)[1] "semana1" "semana2"#muestra el nombre de las filsa
rownames(tabla)[1] "Lunes" "Martes" "Miercoles" "Jueves" "Viernes" "Sabado"
[7] "Domingo" otras operaciones utiles para data.frames:
colSums ()
rowSums ()
colMeans()
rowMeans()
Ejercicio: En el objetvo tabla, crea una columan con el promedio de organismos muertos por dia
Ejercicio: Te diste cuenta que esta nueva columna no te sirve. ¿como la eliminas?
pista: Puedes usar indices o NULL

sessionInfo()R version 4.0.5 (2021-03-31)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows 10 x64 (build 19043)
Matrix products: default
locale:
[1] LC_COLLATE=English_United States.1252
[2] LC_CTYPE=English_United States.1252
[3] LC_MONETARY=English_United States.1252
[4] LC_NUMERIC=C
[5] LC_TIME=English_United States.1252
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] workflowr_1.6.2
loaded via a namespace (and not attached):
[1] Rcpp_1.0.5 whisker_0.4 knitr_1.30 magrittr_2.0.1
[5] R6_2.5.0 rlang_0.4.11 fansi_0.4.2 stringr_1.4.0
[9] tools_4.0.5 xfun_0.23 utf8_1.2.1 git2r_0.27.1
[13] htmltools_0.5.1.1 ellipsis_0.3.1 rprojroot_2.0.2 yaml_2.2.1
[17] digest_0.6.27 tibble_3.0.4 lifecycle_1.0.0 crayon_1.4.1
[21] later_1.1.0.1 vctrs_0.3.8 promises_1.1.1 fs_1.5.0
[25] glue_1.4.2 evaluate_0.14 rmarkdown_2.6 stringi_1.5.3
[29] compiler_4.0.5 pillar_1.6.0 httpuv_1.5.4 pkgconfig_2.0.3