Dplyr y Tidyverse
Miguel Tripp
Last updated: 2021-07-31
Checks: 7 0
Knit directory: 2021/
This reproducible R Markdown analysis was created with workflowr (version 1.6.2). The Checks tab describes the reproducibility checks that were applied when the results were created. The Past versions tab lists the development history.
Great! Since the R Markdown file has been committed to the Git repository, you know the exact version of the code that produced these results.
Great job! The global environment was empty. Objects defined in the global environment can affect the analysis in your R Markdown file in unknown ways. For reproduciblity it’s best to always run the code in an empty environment.
The command set.seed(20210412) was run prior to running the code in the R Markdown file. Setting a seed ensures that any results that rely on randomness, e.g. subsampling or permutations, are reproducible.
Great job! Recording the operating system, R version, and package versions is critical for reproducibility.
Nice! There were no cached chunks for this analysis, so you can be confident that you successfully produced the results during this run.
Great job! Using relative paths to the files within your workflowr project makes it easier to run your code on other machines.
Great! You are using Git for version control. Tracking code development and connecting the code version to the results is critical for reproducibility.
The results in this page were generated with repository version b4e67dc. See the Past versions tab to see a history of the changes made to the R Markdown and HTML files.
Note that you need to be careful to ensure that all relevant files for the analysis have been committed to Git prior to generating the results (you can use wflow_publish or wflow_git_commit). workflowr only checks the R Markdown file, but you know if there are other scripts or data files that it depends on. Below is the status of the Git repository when the results were generated:
Ignored files:
Ignored: .Rhistory
Ignored: .Rproj.user/
Ignored: analysis/hero-image.html
Ignored: analysis/poke_logo.png
Untracked files:
Untracked: Curso_Bioestadistica_MTripp_cuatriII.docx
Untracked: Curso_Bioestadistica_MTripp_cuatriII.pdf
Untracked: Diapositivas/
Untracked: Prueba_markdown.Rmd
Untracked: Prueba_markdown.pdf
Untracked: README.html
Untracked: Resources/
Untracked: Tarea_Tstudent.Rmd
Untracked: Tarea_Tstudent.docx
Untracked: Tarea_Tstudent.html
Untracked: Tarea_Tstudent.pdf
Untracked: analysis/Clase13_noParam.Rmd
Untracked: analysis/hero_backgroud.jpg
Untracked: analysis/images/
Untracked: analysis/style.css
Untracked: analysis/test.Rmd
Untracked: code/tarea_macrograd.R
Untracked: data/CS_subset.csv
Untracked: data/Consumo_oxigeno_wide.csv
Untracked: data/Darwin_esp.csv
Untracked: data/Data_enzimas_Experimento1.txt
Untracked: data/Data_enzimas_Experimento2.txt
Untracked: data/Data_enzimas_Experimento3.txt
Untracked: data/Data_enzimas_Experimento4.txt
Untracked: data/DownloadFestival(No Outlier).dat
Untracked: data/Festival.csv
Untracked: data/Hful_metabolitos_ver2.csv
Untracked: data/Longitud_noParam.csv
Untracked: data/LungCapData.txt
Untracked: data/LungCapDataEsp.csv
Untracked: data/PalmerPenguins.csv
Untracked: data/Pokemon_tabla.csv
Untracked: data/Pokemon_tabla.xls
Untracked: data/RExam.dat
Untracked: data/Rexamendat.csv
Untracked: data/Tabla1_Muestreo.txt
Untracked: data/Transcriptome_Anotacion.csv
Untracked: data/Transcriptome_DGE.csv
Untracked: data/Vinogradov_2004_Titanic.tab
Untracked: data/Vinogradov_2004_Titanic.tab.csv
Untracked: data/data_tukey.txt
Untracked: data/exp_macrogard_growth.tab
Untracked: data/exp_macrogard_rna-dna.tab
Untracked: data/fertilizantes_luz.csv
Untracked: data/gatos_sueno.csv
Untracked: data/macrogard_crecimiento.csv
Untracked: data/pokemon_extended.csv
Untracked: output/Plot_all_penguins.pdf
Untracked: output/Plot_all_penguins.tiff
Untracked: output/graficos/
Unstaged changes:
Modified: analysis/_site.yml
Note that any generated files, e.g. HTML, png, CSS, etc., are not included in this status report because it is ok for generated content to have uncommitted changes.
These are the previous versions of the repository in which changes were made to the R Markdown (analysis/CLase4_Dplyr.Rmd) and HTML (docs/CLase4_Dplyr.html) files. If you’ve configured a remote Git repository (see ?wflow_git_remote), click on the hyperlinks in the table below to view the files as they were in that past version.
| File | Version | Author | Date | Message |
|---|---|---|---|---|
| html | 03db7ff | Miguel Tripp | 2021-07-25 | Build site. |
| html | fcb9047 | Miguel Tripp | 2021-07-25 | Build site. |
| Rmd | 9a992a0 | Miguel Tripp | 2021-07-25 | workflowr::wflow_publish(c(“analysis/index.Rmd”, “analysis/about.Rmd”, |
| html | 2adc7a9 | Miguel Tripp | 2021-07-24 | Build site. |
| html | 9d09420 | Miguel Tripp | 2021-07-22 | Build site. |
| html | 0f7eb2d | Miguel Tripp | 2021-07-12 | Build site. |
| html | 82e4deb | Miguel Tripp | 2021-07-08 | Build site. |
| html | bc7c1d7 | Miguel Tripp | 2021-07-07 | Build site. |
| html | 01ac301 | Miguel Tripp | 2021-07-04 | Build site. |
| html | 392444f | Miguel Tripp | 2021-07-04 | Build site. |
| html | c188ae8 | Miguel Tripp | 2021-06-29 | Build site. |
| html | 1136768 | Miguel Tripp | 2021-06-28 | Build site. |
| html | a33d4bb | Miguel Tripp | 2021-06-23 | Build site. |
| html | 209299f | Miguel Tripp | 2021-06-21 | Build site. |
| html | d025507 | Miguel Tripp | 2021-06-17 | Build site. |
| html | fb9e91e | Miguel Tripp | 2021-06-16 | Build site. |
| html | c5dfe6a | Miguel Tripp | 2021-06-14 | Build site. |
| html | 2352c47 | Miguel Tripp | 2021-06-12 | Build site. |
| html | 99c3644 | Miguel Tripp | 2021-06-05 | Build site. |
| html | fbd7857 | Miguel Tripp | 2021-06-05 | Build site. |
| html | 87a646f | Miguel Tripp | 2021-06-02 | Build site. |
| html | 47a7147 | Miguel Tripp | 2021-05-31 | Build site. |
| html | 95c661f | Miguel Tripp | 2021-05-31 | Build site. |
| html | 34660de | Miguel Tripp | 2021-05-24 | Build site. |
| html | da04854 | Miguel Tripp | 2021-05-24 | Build site. |
| html | 3321935 | Miguel Tripp | 2021-05-24 | Build site. |
| html | 3d12902 | Miguel Tripp | 2021-05-16 | Build site. |
| html | 20b3aab | Miguel Tripp | 2021-05-16 | Build site. |
| html | f20dcb1 | Miguel Tripp | 2021-05-16 | Build site. |
| html | b1ca7f3 | Miguel Tripp | 2021-05-16 | Build site. |
| Rmd | 5126d03 | Miguel Tripp | 2021-05-16 | Publish the initial files for myproject |
| html | b3002cc | Miguel Tripp | 2021-05-04 | Build site. |
| html | 2c60db3 | Miguel Tripp | 2021-05-04 | Build site. |
| html | 65fa0ee | Miguel Tripp | 2021-05-04 | Build site. |
| Rmd | f504fea | Miguel Tripp | 2021-05-04 | Publish the initial files for myproject |
1 Generalidades
Como hemos visto hasta este punto, las funciones base de R contienen herramientas poderosas para el análisis y visualización de datos. Sin embargo, existen herramientas especializadas para la manipulación y presentación de datos (“data wrangling”) los cuales tienen el objetivo de facilitar estas tareas, haciendo los análisis mas eficientes y reproducibles.
2 Tidyverse

El Tidyverse es una colección de paquetes de R que comparten una filosofía común para el manejo de datos de tipo tabular, a lo que se le conoce como tidy data. La filosofía básica de las funciones de los paquetes del Tidyverse es recibir los datos Tidy y devolver como resultados datos del mismo tipo 1 de esta manera es posible “encadenar” una serie de procesos para la manipulación compleja de los datos así como la presentación y visualización
2.1 Instalación de Tidyverse
Como se mencionó anteriormente, Tidyverse no es en si un paquete sino una colección de paquetes. Sin embargo, es posible acceder a los principales paquetes del Tidyverse instalando la librería Tidyverse.
install.packages("tidyverse")y posteriormente se carga la librería lo que producira una salida como la siguiente:
library(tidyverse)-- Attaching packages --------------------------------------- tidyverse 1.3.1 --v ggplot2 3.3.5 v purrr 0.3.4
v tibble 3.0.4 v dplyr 1.0.5
v tidyr 1.1.3 v stringr 1.4.0
v readr 1.4.0 v forcats 0.5.1-- Conflicts ------------------------------------------ tidyverse_conflicts() --
x dplyr::filter() masks stats::filter()
x dplyr::lag() masks stats::lag()indicando todos los paquetes disponibles.
Los paquetes núcleo de tidyverse son los siguientes:
ggplot2: poderosa herramienta para la visualización de datos con la posibilidad de personalización extrema
tibble: implementación de una estructura de datos mejorada de los data.frames.
tidyr: permite realizar transformaciones de los datos tal como la transformación de formatos largo y ancho de tablas
readr: facilita la lectura de archivos de texto plano, como es el caso de los archivos CSV.
purrr: facilita el trabajo con funciones.
dplyr: facilita la manipulación de datos utilizando haciendo uso de un lenguaje intuitivo de acciones sobre estos
stringr: facilita el trabajo con cadenas de texto y vectores.
forcats: facilita el trabajo con datos categóricos.
Si bien el Tidyverse incluye estos ocho paquetes núcleo, hay un número cada vez mas creciente de paquetes que comparten esta filosofía y lenguaje, haciendo compatible estas herramientas.
3 DPLYR
DPLYR Es una librería orientada a la manipulación de datos. Cada función de esta librería realiza una sola tarea y sus nombres son verbos, lo cual hace mas sencillo recordar y comprender su función.
Dplyr es parte del tidyverse y se integra perfectamente con el resto de las librerías que forman parte de este.
En esta introducción nos vamos a enfocar a las funciones:
filter()select()arrangemutate()summarise()%>%
En esta clase, usaremos dplyr así como readxl para importar y modificar una tabla. Dplyr ya se encuentra disponible al momento de cargar el Tidyverse mientras que readxl necesita ser instalado y cargado de manera independiente, aunque es compatible con el resto de los paquetes del Tidyverse.
install.packages("readxl")Una vez instalados, es necesario cargar la librera a nuestro ambiente de trabajo
library(readxl)4 Los Datos

Para esta sección utilizaremos la base de datos de POKEMON de KAGGLE. Este set de datos incluye 721 pokemons con su nombre, tipo en primer y segunto nivel, HP, Ataque, Defensa, Ataque especial, Defensa especial, velocidad y si es legendario o no.
- ID: Número de identificador de cada pokémon
- Name: Nombre de cada Pokémon
- Type 1: Tipo de Pokémon, esto determina su resistencia o debilidad a los ataques
- Type 2: Algunos Pokémon tienen tipo dual
- Total: Es la suma de todos las estadísticas que siguen. Sirve como referencía de que tan fuerte es un pokémon
- HP: Hit Points, define que tanto daño puede tolerar un Pokémon antes de desvanecerse
- Attack: Ataque
- Defense: Defensa
- SP Atk: Ataque especial
- SP Def: Defensa especial
- Speed: Velocidad. Determina cual Pokémon ataca primero en cada ronda
pokemon <- read_excel("data/Pokemon_tabla.xls", sheet = "pokemon")
pokemon# A tibble: 800 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 Bulbasaur Grass Pois~ 318 45 49 49 65 65 45
2 2 Ivysaur Grass Pois~ 405 60 62 63 80 80 60
3 3 Venusaur Grass Pois~ 525 80 82 83 100 100 80
4 3 VenusaurMeg~ Grass Pois~ 625 80 100 123 122 120 80
5 4 Charmander Fire <NA> 309 39 52 43 60 50 65
6 5 Charmeleon Fire <NA> 405 58 64 58 80 65 80
7 6 Charizard Fire Flyi~ 534 78 84 78 109 85 100
8 6 CharizardMe~ Fire Drag~ 634 78 130 111 130 85 100
9 6 CharizardMe~ Fire Flyi~ 634 78 104 78 159 115 100
10 7 Squirtle Water <NA> 314 44 48 65 50 64 43
# ... with 790 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>usa ?read_excel para ver todas las opciones para importar datos
Nota que el encabezado de la tabla es diferente a los data frames que hemos utilizado hasta ahora. Esto se debe a que los datos tienen una estructura de tibble.
¿Tibble?
Los tibbles son data frames pero modifican algunas características antiguas para hacernos la vida más fácil. Los tibbles tienen un método de impresión en la consola refinado: solo muestran las primeras 10 filas y solo aquellas columnas que entran en el ancho de la pantalla. Esto simplifica y facilita trabajar con bases de datos grandes. Además del nombre, cada columna muestra su tipo. Esto último es una gran característica tomada de str().
Si ya te has familiarizado con data.frame(), es importante que tomes en cuenta que tibble() hace menos cosas: nunca cambia el tipo de los inputs (p. ej., ¡nunca convierte caracteres en factores!), nunca cambia el nombre de las variables y nunca asigna nombres a las filas.
Un tibble puede usar nombres de columnas que no son nombres de variables válidos en R (también conocidos como nombres no sintácticos). Por ejemplo, pueden empezar con un cáracter diferente a una letra o contener caracteres poco comunes, como espacios. Para referirse a estas variables, tienes que rodearlos de acentos graves: ```
¿Entonces es read.csv o read_csv?
Otro aspecto que vas a notar al trabajar con el tidyverse es que funciones que estan separadas con un punto (.) ahora se van a ver separadas con un guion bajo (_). Esto es una caracteristica de muchos de los paquetes del Tidyverse o compatibles con la filosofía Tidyverse y que nos permite distinguirlo de los paquetes del núcleo base de R. De manera que la función read.csv() de R base, se vuelve read_csv() en readr, y la función t.test() de R base, se vuelve t_test() en el paquete rstatxs (modulo 7)
5 Uso de Dplyr
El paquete incluye un conjunto de comandos que coinciden con las acciones más comunes que se realizan sobre un conjunto de datos (filtrar filas filter(), seleccionar columnas select(), ordenar arrange(), transformar o añadir nuevas variables mutate(), resumir mediante alguna medida numérica summarise()). Lo que hace que la sintaxis sea especialmente clara es la correspondencia tan nítida entre el comando y la acción. Para llevar a cabo estas acciones debemos tener en cuenta algunas características comunes:
- El primer argumento siempre es un tibble o data.frame
- El resto de los argumentos indican los parametros de lo que queremos hacer
- El resultado siempre tiene tambien estructura de tibble o data frame
5.1 select()
Permite seleccionar una o un set de varias columnas
Por ejemplo: seleccionar las columnas Name y Attack
select(pokemon, Name, Attack)# A tibble: 800 x 2
Name Attack
<chr> <dbl>
1 Bulbasaur 49
2 Ivysaur 62
3 Venusaur 82
4 VenusaurMega Venusaur 100
5 Charmander 52
6 Charmeleon 64
7 Charizard 84
8 CharizardMega Charizard X 130
9 CharizardMega Charizard Y 104
10 Squirtle 48
# ... with 790 more rowsAl igual que en ejemplo anteriores, podemos seleccionar todo excepto una clumna especifica usando el operador “-” (resta)
select(pokemon, -Attack)# A tibble: 800 x 12
ID Name Type1 Type2 Total HP Defense Sp_Atk Sp_Def Speed Generation
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 Bulbasa~ Grass Pois~ 318 45 49 65 65 45 1
2 2 Ivysaur Grass Pois~ 405 60 63 80 80 60 1
3 3 Venusaur Grass Pois~ 525 80 83 100 100 80 1
4 3 Venusau~ Grass Pois~ 625 80 123 122 120 80 1
5 4 Charman~ Fire <NA> 309 39 43 60 50 65 1
6 5 Charmel~ Fire <NA> 405 58 58 80 65 80 1
7 6 Chariza~ Fire Flyi~ 534 78 78 109 85 100 1
8 6 Chariza~ Fire Drag~ 634 78 111 130 85 100 1
9 6 Chariza~ Fire Flyi~ 634 78 78 159 115 100 1
10 7 Squirtle Water <NA> 314 44 65 50 64 43 1
# ... with 790 more rows, and 1 more variable: Legendary <lgl>Para seleccionar un intervalo de columnas, se puede usar el operador :
select(pokemon, Name, Type1:HP)# A tibble: 800 x 5
Name Type1 Type2 Total HP
<chr> <chr> <chr> <dbl> <dbl>
1 Bulbasaur Grass Poison 318 45
2 Ivysaur Grass Poison 405 60
3 Venusaur Grass Poison 525 80
4 VenusaurMega Venusaur Grass Poison 625 80
5 Charmander Fire <NA> 309 39
6 Charmeleon Fire <NA> 405 58
7 Charizard Fire Flying 534 78
8 CharizardMega Charizard X Fire Dragon 634 78
9 CharizardMega Charizard Y Fire Flying 634 78
10 Squirtle Water <NA> 314 44
# ... with 790 more rowsDe igual manera, se puede usar el operados : para descartar un intervalo de columnas
select(pokemon, -(Defense:Legendary))# A tibble: 800 x 7
ID Name Type1 Type2 Total HP Attack
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl>
1 1 Bulbasaur Grass Poison 318 45 49
2 2 Ivysaur Grass Poison 405 60 62
3 3 Venusaur Grass Poison 525 80 82
4 3 VenusaurMega Venusaur Grass Poison 625 80 100
5 4 Charmander Fire <NA> 309 39 52
6 5 Charmeleon Fire <NA> 405 58 64
7 6 Charizard Fire Flying 534 78 84
8 6 CharizardMega Charizard X Fire Dragon 634 78 130
9 6 CharizardMega Charizard Y Fire Flying 634 78 104
10 7 Squirtle Water <NA> 314 44 48
# ... with 790 more rowsSe puede seleccionar las columnas que empiezar con un determinado caracter con la función starts_with(); por ejemplo, si queremos enfocarnos a las columnas con del tipoe (Type):
select(pokemon, Name, starts_with("Type"))# A tibble: 800 x 3
Name Type1 Type2
<chr> <chr> <chr>
1 Bulbasaur Grass Poison
2 Ivysaur Grass Poison
3 Venusaur Grass Poison
4 VenusaurMega Venusaur Grass Poison
5 Charmander Fire <NA>
6 Charmeleon Fire <NA>
7 Charizard Fire Flying
8 CharizardMega Charizard X Fire Dragon
9 CharizardMega Charizard Y Fire Flying
10 Squirtle Water <NA>
# ... with 790 more rowsAlgunos operadores que funcionan de manera similar son:
ends_with()= Selecciona las columnas que terminan con una cadena de caracterescontains()= Selecciona las columnas que contienen una cadena de caracteres.
select(pokemon, Name, contains("Sp_"))# A tibble: 800 x 3
Name Sp_Atk Sp_Def
<chr> <dbl> <dbl>
1 Bulbasaur 65 65
2 Ivysaur 80 80
3 Venusaur 100 100
4 VenusaurMega Venusaur 122 120
5 Charmander 60 50
6 Charmeleon 80 65
7 Charizard 109 85
8 CharizardMega Charizard X 130 85
9 CharizardMega Charizard Y 159 115
10 Squirtle 50 64
# ... with 790 more rowsCon la función evrything() se puede seleccionar todo, y con esto es factible cambiar el order de las columnas. Por ejemplo, si quieremos que la primera columna sea “Type1” ejecutamos:
select(pokemon, Type1, everything())# A tibble: 800 x 13
Type1 ID Name Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<chr> <dbl> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Grass 1 Bulbasaur Pois~ 318 45 49 49 65 65 45
2 Grass 2 Ivysaur Pois~ 405 60 62 63 80 80 60
3 Grass 3 Venusaur Pois~ 525 80 82 83 100 100 80
4 Grass 3 VenusaurMeg~ Pois~ 625 80 100 123 122 120 80
5 Fire 4 Charmander <NA> 309 39 52 43 60 50 65
6 Fire 5 Charmeleon <NA> 405 58 64 58 80 65 80
7 Fire 6 Charizard Flyi~ 534 78 84 78 109 85 100
8 Fire 6 CharizardMe~ Drag~ 634 78 130 111 130 85 100
9 Fire 6 CharizardMe~ Flyi~ 634 78 104 78 159 115 100
10 Water 7 Squirtle <NA> 314 44 48 65 50 64 43
# ... with 790 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>5.2 filter()
Esta función nos permite seleccionar las filas que cumplan con las condiciones que especifiquemos
Por ejemplo, filtrar los pokemon con un nivel de ataque Attack superior a 100
filter(pokemon, Attack > 100)# A tibble: 170 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 6 CharizardM~ Fire Drag~ 634 78 130 111 130 85 100
2 6 CharizardM~ Fire Flyi~ 634 78 104 78 159 115 100
3 9 BlastoiseM~ Water <NA> 630 79 103 120 135 115 78
4 15 BeedrillMe~ Bug Pois~ 495 65 150 40 15 80 145
5 34 Nidoking Poison Grou~ 505 81 102 77 85 75 85
6 57 Primeape Fight~ <NA> 455 65 105 60 60 70 95
7 59 Arcanine Fire <NA> 555 90 110 80 100 80 95
8 68 Machamp Fight~ <NA> 505 90 130 80 65 85 55
9 71 Victreebel Grass Pois~ 490 80 105 65 100 70 70
10 76 Golem Rock Grou~ 495 80 120 130 55 65 45
# ... with 160 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>Filtrar los pokemon que tengan un nivel de ataque mayor a 100 y que sean de tipo fuego
filter(pokemon, Attack > 100, Type1 == "Fire")# A tibble: 13 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 6 CharizardMe~ Fire Drag~ 634 78 130 111 130 85 100
2 6 CharizardMe~ Fire Flyi~ 634 78 104 78 159 115 100
3 59 Arcanine Fire <NA> 555 90 110 80 100 80 95
4 136 Flareon Fire <NA> 525 65 130 60 95 110 65
5 244 Entei Fire <NA> 580 115 115 85 90 75 100
6 250 Ho-oh Fire Flyi~ 680 106 130 90 110 154 90
7 257 Blaziken Fire Figh~ 530 80 120 70 110 70 80
8 257 BlazikenMeg~ Fire Figh~ 630 80 160 80 130 80 100
9 323 CameruptMeg~ Fire Grou~ 560 70 120 100 145 105 20
10 392 Infernape Fire Figh~ 534 76 104 71 104 71 108
11 500 Emboar Fire Figh~ 528 110 123 65 100 65 65
12 555 DarmanitanS~ Fire <NA> 480 105 140 55 30 55 95
13 721 Volcanion Fire Water 600 80 110 120 130 90 70
# ... with 2 more variables: Generation <dbl>, Legendary <lgl>Filtrar los pokemons que tengan nivel de ataque mayor a 100 y que sean de tipo fuego y que sean de la primera generación
filter(pokemon, Attack > 100,
Type1 == "Fire",
Generation == 1)# A tibble: 4 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 6 CharizardMeg~ Fire Drag~ 634 78 130 111 130 85 100
2 6 CharizardMeg~ Fire Flyi~ 634 78 104 78 159 115 100
3 59 Arcanine Fire <NA> 555 90 110 80 100 80 95
4 136 Flareon Fire <NA> 525 65 130 60 95 110 65
# ... with 2 more variables: Generation <dbl>, Legendary <lgl>Ahora queremos filtrar los pokemon que tengan un nivel de ataque mayor a 100 o que sean de tipo fuego
filter(pokemon, Attack > 100 | Type1 == "Fire")# A tibble: 209 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 4 Charmander Fire <NA> 309 39 52 43 60 50 65
2 5 Charmeleon Fire <NA> 405 58 64 58 80 65 80
3 6 Charizard Fire Flyi~ 534 78 84 78 109 85 100
4 6 CharizardMe~ Fire Drag~ 634 78 130 111 130 85 100
5 6 CharizardMe~ Fire Flyi~ 634 78 104 78 159 115 100
6 9 BlastoiseMe~ Water <NA> 630 79 103 120 135 115 78
7 15 BeedrillMeg~ Bug Pois~ 495 65 150 40 15 80 145
8 34 Nidoking Pois~ Grou~ 505 81 102 77 85 75 85
9 37 Vulpix Fire <NA> 299 38 41 40 50 65 65
10 38 Ninetales Fire <NA> 505 73 76 75 81 100 100
# ... with 199 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>Si queremos filtrar distintas condiciones en una misma variable podemos usar el operador %in% por ejemplo para filtrar lo pokemons que sea de tipo1 de Fuego y Agua
filter(pokemon, Type1 %in% c("Fire", "Water"))# A tibble: 164 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 4 Charmander Fire <NA> 309 39 52 43 60 50 65
2 5 Charmeleon Fire <NA> 405 58 64 58 80 65 80
3 6 Charizard Fire Flyi~ 534 78 84 78 109 85 100
4 6 CharizardMe~ Fire Drag~ 634 78 130 111 130 85 100
5 6 CharizardMe~ Fire Flyi~ 634 78 104 78 159 115 100
6 7 Squirtle Water <NA> 314 44 48 65 50 64 43
7 8 Wartortle Water <NA> 405 59 63 80 65 80 58
8 9 Blastoise Water <NA> 530 79 83 100 85 105 78
9 9 BlastoiseMe~ Water <NA> 630 79 103 120 135 115 78
10 37 Vulpix Fire <NA> 299 38 41 40 50 65 65
# ... with 154 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>Es posible filtrar un intervalo de valores utilizando el argumento between(). Por ejemplo, para filtrar los pokemon que tengan un nivel de ataque mayor a 100 pero menor a 150
filter(pokemon,
between(Attack,100,150))# A tibble: 192 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 3 VenusaurMe~ Grass Pois~ 625 80 100 123 122 120 80
2 6 CharizardM~ Fire Drag~ 634 78 130 111 130 85 100
3 6 CharizardM~ Fire Flyi~ 634 78 104 78 159 115 100
4 9 BlastoiseM~ Water <NA> 630 79 103 120 135 115 78
5 15 BeedrillMe~ Bug Pois~ 495 65 150 40 15 80 145
6 28 Sandslash Ground <NA> 450 75 100 110 45 55 65
7 34 Nidoking Poison Grou~ 505 81 102 77 85 75 85
8 57 Primeape Fight~ <NA> 455 65 105 60 60 70 95
9 59 Arcanine Fire <NA> 555 90 110 80 100 80 95
10 67 Machoke Fight~ <NA> 405 80 100 70 50 60 45
# ... with 182 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>para hacer la selección opuesta, se puede agregar el operador !
filter(pokemon, !between(Attack, 100, 150))# A tibble: 608 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 Bulbasaur Grass Poison 318 45 49 49 65 65 45
2 2 Ivysaur Grass Poison 405 60 62 63 80 80 60
3 3 Venusaur Grass Poison 525 80 82 83 100 100 80
4 4 Charmander Fire <NA> 309 39 52 43 60 50 65
5 5 Charmeleon Fire <NA> 405 58 64 58 80 65 80
6 6 Charizard Fire Flying 534 78 84 78 109 85 100
7 7 Squirtle Water <NA> 314 44 48 65 50 64 43
8 8 Wartortle Water <NA> 405 59 63 80 65 80 58
9 9 Blastoise Water <NA> 530 79 83 100 85 105 78
10 10 Caterpie Bug <NA> 195 45 30 35 20 20 45
# ... with 598 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>filter(pokemon,
between(Attack, 0, 50),
between(Speed, 20, 30))# A tibble: 30 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 11 Metapod Bug <NA> 205 50 20 55 25 25 30
2 39 Jigglypuff Normal Fairy 270 115 45 20 45 25 20
3 43 Oddish Grass Poison 320 45 50 55 75 65 30
4 161 Sentret Normal <NA> 215 35 46 34 35 45 20
5 175 Togepi Fairy <NA> 245 35 20 65 40 65 20
6 191 Sunkern Grass <NA> 180 30 30 30 30 30 30
7 218 Slugma Fire <NA> 250 40 40 40 70 40 20
8 219 Magcargo Fire Rock 410 50 50 120 80 80 30
9 265 Wurmple Bug <NA> 195 45 45 35 20 30 20
10 270 Lotad Water Grass 220 40 30 30 40 50 30
# ... with 20 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>Tambien es posible filtrar una variable a partir de un caracter en especifico usando la funcion str_detect() *. Por ejemplo podemos buscar la informacion de Pikachu
str_detect()es parte del paquete Stringr pero es compatible con el uso de Dplyr
filter(pokemon, str_detect(Name, "Pikachu"))# A tibble: 1 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 25 Pikachu Electric <NA> 320 35 55 40 50 50 90
# ... with 2 more variables: Generation <dbl>, Legendary <lgl>Ejercicio: ¿Quien es ese pokemon?

Nuestro pokemon tiene un valor de HP entre 100 y 200; valor de velocidad menor a 50; es del tipo1 Normal; tiene un valor de defensa menor a 40 y pertenece a la 1er generación
filter(pokemon,
between(HP, 100, 200),
Speed < 50,
Type1 == "Normal",
Defense < 40,
Generation == 1,
)5.3 mutate()

La función mutate nos permite crear nuevas variables que contengan cálculos a partir de las que ya tenemos, añadir una nueva variable o transformar una variable ya existente
Por ejemplo, podemos generar una nueva columna llamada “At_Df_ratio” con la propoción de los valors de Ataque sobre Defensa
mutate(pokemon, At_df_ratio = Attack / Defense)# A tibble: 800 x 14
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 Bulbasaur Grass Pois~ 318 45 49 49 65 65 45
2 2 Ivysaur Grass Pois~ 405 60 62 63 80 80 60
3 3 Venusaur Grass Pois~ 525 80 82 83 100 100 80
4 3 VenusaurMeg~ Grass Pois~ 625 80 100 123 122 120 80
5 4 Charmander Fire <NA> 309 39 52 43 60 50 65
6 5 Charmeleon Fire <NA> 405 58 64 58 80 65 80
7 6 Charizard Fire Flyi~ 534 78 84 78 109 85 100
8 6 CharizardMe~ Fire Drag~ 634 78 130 111 130 85 100
9 6 CharizardMe~ Fire Flyi~ 634 78 104 78 159 115 100
10 7 Squirtle Water <NA> 314 44 48 65 50 64 43
# ... with 790 more rows, and 3 more variables: Generation <dbl>,
# Legendary <lgl>, At_df_ratio <dbl>De igual manera se pueden generar diversas nuevas columnas, separadas por comas.
mutate(pokemon, At_df_ratio = Attack / Defense,
#En porcentage
At_df_per = At_df_ratio * 100,
#Añadir una nueva columna con el nombre de los datos
data = "Pokemon")# A tibble: 800 x 16
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 Bulbasaur Grass Pois~ 318 45 49 49 65 65 45
2 2 Ivysaur Grass Pois~ 405 60 62 63 80 80 60
3 3 Venusaur Grass Pois~ 525 80 82 83 100 100 80
4 3 VenusaurMeg~ Grass Pois~ 625 80 100 123 122 120 80
5 4 Charmander Fire <NA> 309 39 52 43 60 50 65
6 5 Charmeleon Fire <NA> 405 58 64 58 80 65 80
7 6 Charizard Fire Flyi~ 534 78 84 78 109 85 100
8 6 CharizardMe~ Fire Drag~ 634 78 130 111 130 85 100
9 6 CharizardMe~ Fire Flyi~ 634 78 104 78 159 115 100
10 7 Squirtle Water <NA> 314 44 48 65 50 64 43
# ... with 790 more rows, and 5 more variables: Generation <dbl>,
# Legendary <lgl>, At_df_ratio <dbl>, At_df_per <dbl>, data <chr>con mutate, tambien es posible cambiar el tipo de dato de cada columna. Por ejemplo, cambiar la columna “Type1” de carácter a factor:
pokemon_fct <- mutate(pokemon, Type1 = factor(Type1))Si vemos el tipo de datos, veremos la diferencia:
str(pokemon_fct)tibble [800 x 13] (S3: tbl_df/tbl/data.frame)
$ ID : num [1:800] 1 2 3 3 4 5 6 6 6 7 ...
$ Name : chr [1:800] "Bulbasaur" "Ivysaur" "Venusaur" "VenusaurMega Venusaur" ...
$ Type1 : Factor w/ 18 levels "Bug","Dark","Dragon",..: 10 10 10 10 7 7 7 7 7 18 ...
$ Type2 : chr [1:800] "Poison" "Poison" "Poison" "Poison" ...
$ Total : num [1:800] 318 405 525 625 309 405 534 634 634 314 ...
$ HP : num [1:800] 45 60 80 80 39 58 78 78 78 44 ...
$ Attack : num [1:800] 49 62 82 100 52 64 84 130 104 48 ...
$ Defense : num [1:800] 49 63 83 123 43 58 78 111 78 65 ...
$ Sp_Atk : num [1:800] 65 80 100 122 60 80 109 130 159 50 ...
$ Sp_Def : num [1:800] 65 80 100 120 50 65 85 85 115 64 ...
$ Speed : num [1:800] 45 60 80 80 65 80 100 100 100 43 ...
$ Generation: num [1:800] 1 1 1 1 1 1 1 1 1 1 ...
$ Legendary : logi [1:800] FALSE FALSE FALSE FALSE FALSE FALSE ...5.4 rename()
Te permite cambiar el nombre de una variable
rename(pokemon,
Nombre = Name, Tipo1 = Type1)# A tibble: 800 x 13
ID Nombre Tipo1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 Bulbasaur Grass Pois~ 318 45 49 49 65 65 45
2 2 Ivysaur Grass Pois~ 405 60 62 63 80 80 60
3 3 Venusaur Grass Pois~ 525 80 82 83 100 100 80
4 3 VenusaurMeg~ Grass Pois~ 625 80 100 123 122 120 80
5 4 Charmander Fire <NA> 309 39 52 43 60 50 65
6 5 Charmeleon Fire <NA> 405 58 64 58 80 65 80
7 6 Charizard Fire Flyi~ 534 78 84 78 109 85 100
8 6 CharizardMe~ Fire Drag~ 634 78 130 111 130 85 100
9 6 CharizardMe~ Fire Flyi~ 634 78 104 78 159 115 100
10 7 Squirtle Water <NA> 314 44 48 65 50 64 43
# ... with 790 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>5.5 arrange()
La función arrange nos permite order los datos en función a los valores de otra variable
Si queremos mostrar ordenados de menor a mayor los valores de nuestro conjunto de datos en función al valor de ataque:
arrange(pokemon, Attack)# A tibble: 800 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 113 Chansey Normal <NA> 450 250 5 5 35 105 50
2 440 Happiny Normal <NA> 220 100 5 5 15 65 30
3 129 Magikarp Water <NA> 200 20 10 55 15 20 80
4 213 Shuckle Bug Rock 505 20 10 230 10 230 5
5 242 Blissey Normal <NA> 540 255 10 10 75 135 55
6 349 Feebas Water <NA> 200 20 15 20 10 55 80
7 11 Metapod Bug <NA> 205 50 20 55 25 25 30
8 63 Abra Psychic <NA> 310 25 20 15 105 55 90
9 165 Ledyba Bug Flying 265 40 20 30 40 80 55
10 175 Togepi Fairy <NA> 245 35 20 65 40 65 20
# ... with 790 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>Este ordenara los datos de menor a mayor, si se quiere ordenar de mayor a menor se debe agregar el operador - o desc()
arrange(pokemon, desc(Attack))# A tibble: 800 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 150 MewtwoMega~ Psych~ Figh~ 780 106 190 100 154 100 130
2 214 HeracrossM~ Bug Figh~ 600 80 185 115 40 105 75
3 383 GroudonPri~ Ground Fire 770 100 180 160 150 90 90
4 384 RayquazaMe~ Dragon Flyi~ 780 105 180 100 180 100 115
5 386 DeoxysAtta~ Psych~ <NA> 600 50 180 20 180 20 150
6 445 GarchompMe~ Dragon Grou~ 700 108 170 115 120 95 92
7 646 KyuremBlac~ Dragon Ice 700 125 170 100 120 90 95
8 354 BanetteMeg~ Ghost <NA> 555 64 165 75 93 83 75
9 409 Rampardos Rock <NA> 495 97 165 60 65 50 58
10 475 GalladeMeg~ Psych~ Figh~ 618 68 165 95 65 115 110
# ... with 790 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>arrange(pokemon, -Attack)# A tibble: 800 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 150 MewtwoMega~ Psych~ Figh~ 780 106 190 100 154 100 130
2 214 HeracrossM~ Bug Figh~ 600 80 185 115 40 105 75
3 383 GroudonPri~ Ground Fire 770 100 180 160 150 90 90
4 384 RayquazaMe~ Dragon Flyi~ 780 105 180 100 180 100 115
5 386 DeoxysAtta~ Psych~ <NA> 600 50 180 20 180 20 150
6 445 GarchompMe~ Dragon Grou~ 700 108 170 115 120 95 92
7 646 KyuremBlac~ Dragon Ice 700 125 170 100 120 90 95
8 354 BanetteMeg~ Ghost <NA> 555 64 165 75 93 83 75
9 409 Rampardos Rock <NA> 495 97 165 60 65 50 58
10 475 GalladeMeg~ Psych~ Figh~ 618 68 165 95 65 115 110
# ... with 790 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>Tambien es posible ordernar caracteres
arrange(pokemon, Name)# A tibble: 800 x 13
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 460 Abomasnow Grass Ice 494 90 92 75 92 85 60
2 460 AbomasnowMe~ Grass Ice 594 90 132 105 132 105 30
3 63 Abra Psyc~ <NA> 310 25 20 15 105 55 90
4 359 Absol Dark <NA> 465 65 130 60 75 60 75
5 359 AbsolMega A~ Dark <NA> 565 65 150 60 115 60 115
6 617 Accelgor Bug <NA> 495 80 70 40 100 60 145
7 681 AegislashBl~ Steel Ghost 520 60 150 50 150 50 60
8 681 AegislashSh~ Steel Ghost 520 60 50 150 50 150 60
9 142 Aerodactyl Rock Flyi~ 515 80 105 65 60 75 130
10 142 AerodactylM~ Rock Flyi~ 615 80 135 85 70 95 150
# ... with 790 more rows, and 2 more variables: Generation <dbl>,
# Legendary <lgl>5.6 pipes %>%

El operador %>% nos permite encadenar funciones sin tener que ir creando variables para uso temporal o sin tener que anidar funciones.
Dplyr importa este operador de otro paquete (magrittr). De manera general, el operador permite unir el resultado de una función a la entrada de otra función.
Por ejemplo, si yo quiere filtrar los pokemon con valor de ataque mayor a 100, seleccionar las columnas Name y Attack y posteriormente mostrar solo las primeras seis filas (head) tendria que correr la siguiente linea:
head(
select(
filter(pokemon, Attack <100),
Name, Attack)
)# A tibble: 6 x 2
Name Attack
<chr> <dbl>
1 Bulbasaur 49
2 Ivysaur 62
3 Venusaur 82
4 Charmander 52
5 Charmeleon 64
6 Charizard 84usando pipes, lo podemos reescribir de la siguiente manera:
pokemon %>%
filter(Attack < 100) %>%
select(Name, Attack) %>%
head# A tibble: 6 x 2
Name Attack
<chr> <dbl>
1 Bulbasaur 49
2 Ivysaur 62
3 Venusaur 82
4 Charmander 52
5 Charmeleon 64
6 Charizard 84De esta manera podemos unir varias funciones en una sola cadena. Por ejemplo, para seleccionar columnas y dentro de estas, ordenar las variables podemos ejecutar el código de la siguiente forma:
pokemon %>%
select(Name, Type1, Attack) %>%
arrange(desc(Type1), -Attack) %>%
head(10)# A tibble: 10 x 3
Name Type1 Attack
<chr> <chr> <dbl>
1 GyaradosMega Gyarados Water 155
2 SwampertMega Swampert Water 150
3 KyogrePrimal Kyogre Water 150
4 SharpedoMega Sharpedo Water 140
5 Kingler Water 130
6 Gyarados Water 125
7 Sharpedo Water 120
8 Crawdaunt Water 120
9 Palkia Water 120
10 Swampert Water 110Los resultados de una cadena pueden asignarse a un nuevo objeto.
poke_pipe <- pokemon %>%
select(Name, Type1, Attack) %>%
arrange(desc(Type1), -Attack) %>%
head(10)
poke_pipe# A tibble: 10 x 3
Name Type1 Attack
<chr> <chr> <dbl>
1 GyaradosMega Gyarados Water 155
2 SwampertMega Swampert Water 150
3 KyogrePrimal Kyogre Water 150
4 SharpedoMega Sharpedo Water 140
5 Kingler Water 130
6 Gyarados Water 125
7 Sharpedo Water 120
8 Crawdaunt Water 120
9 Palkia Water 120
10 Swampert Water 110Ejercicio 1: A partir de la tabla
pokemoncrea una nueva tabla que contenga:
- una columna llamada
Atack_ratiocon la proporción de Sp_Atk / Attack - seleccionando solamente las columnas “Name”, “Type1” y “Atack_ratio”
- filtrando las especies con una Attack_ratio > 2
- ordenados de forma descendente.
pokemon_Attack_ratio <- pokemon %>%
mutate(Attack_ratio = Sp_Atk / Attack) %>%
select(Name, Type1, Attack_ratio) %>%
filter(Attack_ratio > 2) %>%
arrange(desc(Attack_ratio))Ejercicio 2: Crea una nueva tabla que contenga:
- Seleccionar Name, Type1, Total, y ambos valores de Sp_
- Sin generar una columna nueva transforma los valores en Sp_Atk y SP_Def en porcentaje
- Ordena la tabla en función de los valores descendentes del % de Sp_Atk
- los primeros 10 valores
pokemon_pct <- pokemon %>%
select(Name, Type1, Total, contains("Sp_")) %>%
mutate(Sp_Atk = (Sp_Atk * 100) / Total,
Sp_Def = (Sp_Def * 100) / Total) %>%
arrange(desc(Sp_Atk)) %>%
head(10)5.7 summarise()
La función summarise() nos permite crear resúmenes estadísticos para nuestrs datos a partir de una determinada columna. Para esto, podemos usar las funciones ya conocidas, tales como mean(), min(), max(), sd(),n(). Por ejemplo
pokemon %>%
summarise(promedio = mean(Attack),
minimo = min(Attack),
maximo = max(Attack),
N = n())# A tibble: 1 x 4
promedio minimo maximo N
<dbl> <dbl> <dbl> <int>
1 79.0 5 190 800¿summarise o summarize?
En versiones viejas es posible que te topes con una función summarize(). Esta es exactamente la misma que summarise() pero en su versión inglesa y desaparecera en futuras versiones.
5.8 Operaciones de grupo usando group_by()
El verbo group_by() permite “separar” agrupar la tabla en función a una o mas variables y aplicar la funcion summarise().
pokemon %>%
group_by(Type1) %>%
summarise(promedio = mean(Attack),
desvest = sd(Attack),
N = n())# A tibble: 18 x 4
Type1 promedio desvest N
<chr> <dbl> <dbl> <int>
1 Bug 71.0 37.0 69
2 Dark 88.4 25.8 31
3 Dragon 112. 33.7 32
4 Electric 69.1 23.8 44
5 Fairy 61.5 29.8 17
6 Fighting 96.8 28.3 27
7 Fire 84.8 28.8 52
8 Flying 78.8 37.5 4
9 Ghost 73.8 29.6 32
10 Grass 73.2 25.4 70
11 Ground 95.8 33.1 32
12 Ice 72.8 27.3 24
13 Normal 73.5 30.3 98
14 Poison 74.7 19.6 28
15 Psychic 71.5 42.3 57
16 Rock 92.9 35.3 44
17 Steel 92.7 30.4 27
18 Water 74.2 28.4 112pokemon %>%
group_by(Type1, Type2) %>%
summarise(promedio = mean(Attack),
desvest = sd(Attack),
N = n())`summarise()` has grouped output by 'Type1'. You can override using the `.groups` argument.# A tibble: 154 x 5
# Groups: Type1 [18]
Type1 Type2 promedio desvest N
<chr> <chr> <dbl> <dbl> <int>
1 Bug Electric 62 21.2 2
2 Bug Fighting 155 42.4 2
3 Bug Fire 72.5 17.7 2
4 Bug Flying 70.1 35.4 14
5 Bug Ghost 90 NA 1
6 Bug Grass 73.8 20.4 6
7 Bug Ground 62 24.0 2
8 Bug Poison 68.3 34.3 12
9 Bug Rock 56.7 43.1 3
10 Bug Steel 115. 27.8 7
# ... with 144 more rowsEsto tambien nos permite generar una nueva columna con información relevante
pokemon %>%
group_by(Type1, Type2) %>%
mutate(promedio_grupo = mean(Attack))# A tibble: 800 x 14
# Groups: Type1, Type2 [154]
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 Bulbasaur Grass Pois~ 318 45 49 49 65 65 45
2 2 Ivysaur Grass Pois~ 405 60 62 63 80 80 60
3 3 Venusaur Grass Pois~ 525 80 82 83 100 100 80
4 3 VenusaurMeg~ Grass Pois~ 625 80 100 123 122 120 80
5 4 Charmander Fire <NA> 309 39 52 43 60 50 65
6 5 Charmeleon Fire <NA> 405 58 64 58 80 65 80
7 6 Charizard Fire Flyi~ 534 78 84 78 109 85 100
8 6 CharizardMe~ Fire Drag~ 634 78 130 111 130 85 100
9 6 CharizardMe~ Fire Flyi~ 634 78 104 78 159 115 100
10 7 Squirtle Water <NA> 314 44 48 65 50 64 43
# ... with 790 more rows, and 3 more variables: Generation <dbl>,
# Legendary <lgl>, promedio_grupo <dbl>5.9 summarise_all()
La función summarise_all() requiere una función como argumento, la cual aplica a todas las columnas de la tabla. En este ejemplo se agrego el argumento na.rm = TRUE el cual ignora los NAs
pokemon %>%
group_by(Type1) %>%
summarise_all(mean, na.rm =TRUE)# A tibble: 18 x 13
Type1 ID Name Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Bug 334. NA NA 379. 56.9 71.0 70.7 53.9 64.8 61.7
2 Dark 461. NA NA 446. 66.8 88.4 70.2 74.6 69.5 76.2
3 Dragon 474. NA NA 551. 83.3 112. 86.4 96.8 88.8 83.0
4 Electric 364. NA NA 443. 59.8 69.1 66.3 90.0 73.7 84.5
5 Fairy 450. NA NA 413. 74.1 61.5 65.7 78.5 84.7 48.6
6 Fighting 364. NA NA 416. 69.9 96.8 65.9 53.1 64.7 66.1
7 Fire 327. NA NA 458. 69.9 84.8 67.8 89.0 72.2 74.4
8 Flying 678. NA NA 485 70.8 78.8 66.2 94.2 72.5 102.
9 Ghost 486. NA NA 440. 64.4 73.8 81.2 79.3 76.5 64.3
10 Grass 345. NA NA 421. 67.3 73.2 70.8 77.5 70.4 61.9
11 Ground 356. NA NA 438. 73.8 95.8 84.8 56.5 62.8 63.9
12 Ice 424. NA NA 433. 72 72.8 71.4 77.5 76.3 63.5
13 Normal 319. NA NA 402. 77.3 73.5 59.8 55.8 63.7 71.6
14 Poison 252. NA NA 399. 67.2 74.7 68.8 60.4 64.4 63.6
15 Psychic 381. NA NA 476. 70.6 71.5 67.7 98.4 86.3 81.5
16 Rock 393. NA NA 454. 65.4 92.9 101. 63.3 75.5 55.9
17 Steel 443. NA NA 488. 65.2 92.7 126. 67.5 80.6 55.3
18 Water 303. NA NA 430. 72.1 74.2 72.9 74.8 70.5 66.0
# ... with 2 more variables: Generation <dbl>, Legendary <dbl>Como puedes ver, la función calcula el promedio de todas las variables, y cuando encuentra variables no numericas, devuelve NA
Esto se puede solucionar usando una combinación de across y where; donde across the permite seleccionar un intervalo de variables y where, especificar una cualidad
pokemon %>%
group_by(Type1) %>%
summarise(across(5:8, mean))# A tibble: 18 x 5
Type1 HP Attack Defense Sp_Atk
<chr> <dbl> <dbl> <dbl> <dbl>
1 Bug 56.9 71.0 70.7 53.9
2 Dark 66.8 88.4 70.2 74.6
3 Dragon 83.3 112. 86.4 96.8
4 Electric 59.8 69.1 66.3 90.0
5 Fairy 74.1 61.5 65.7 78.5
6 Fighting 69.9 96.8 65.9 53.1
7 Fire 69.9 84.8 67.8 89.0
8 Flying 70.8 78.8 66.2 94.2
9 Ghost 64.4 73.8 81.2 79.3
10 Grass 67.3 73.2 70.8 77.5
11 Ground 73.8 95.8 84.8 56.5
12 Ice 72 72.8 71.4 77.5
13 Normal 77.3 73.5 59.8 55.8
14 Poison 67.2 74.7 68.8 60.4
15 Psychic 70.6 71.5 67.7 98.4
16 Rock 65.4 92.9 101. 63.3
17 Steel 65.2 92.7 126. 67.5
18 Water 72.1 74.2 72.9 74.8Calcular el promedio solamente en las variables numericas
pokemon %>%
group_by(Type1) %>%
summarise(
across(where(is.numeric), mean, na.rm = TRUE)
)# A tibble: 18 x 10
Type1 ID Total HP Attack Defense Sp_Atk Sp_Def Speed Generation
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Bug 334. 379. 56.9 71.0 70.7 53.9 64.8 61.7 3.22
2 Dark 461. 446. 66.8 88.4 70.2 74.6 69.5 76.2 4.03
3 Dragon 474. 551. 83.3 112. 86.4 96.8 88.8 83.0 3.88
4 Electric 364. 443. 59.8 69.1 66.3 90.0 73.7 84.5 3.27
5 Fairy 450. 413. 74.1 61.5 65.7 78.5 84.7 48.6 4.12
6 Fighting 364. 416. 69.9 96.8 65.9 53.1 64.7 66.1 3.37
7 Fire 327. 458. 69.9 84.8 67.8 89.0 72.2 74.4 3.21
8 Flying 678. 485 70.8 78.8 66.2 94.2 72.5 102. 5.5
9 Ghost 486. 440. 64.4 73.8 81.2 79.3 76.5 64.3 4.19
10 Grass 345. 421. 67.3 73.2 70.8 77.5 70.4 61.9 3.36
11 Ground 356. 438. 73.8 95.8 84.8 56.5 62.8 63.9 3.16
12 Ice 424. 433. 72 72.8 71.4 77.5 76.3 63.5 3.54
13 Normal 319. 402. 77.3 73.5 59.8 55.8 63.7 71.6 3.05
14 Poison 252. 399. 67.2 74.7 68.8 60.4 64.4 63.6 2.54
15 Psychic 381. 476. 70.6 71.5 67.7 98.4 86.3 81.5 3.39
16 Rock 393. 454. 65.4 92.9 101. 63.3 75.5 55.9 3.45
17 Steel 443. 488. 65.2 92.7 126. 67.5 80.6 55.3 3.85
18 Water 303. 430. 72.1 74.2 72.9 74.8 70.5 66.0 2.86Hacer calculo en variables especificas
pokemon %>%
group_by(Type1) %>%
summarise(
across(contains("Sp_"), mean, na.rm = TRUE)
)# A tibble: 18 x 3
Type1 Sp_Atk Sp_Def
<chr> <dbl> <dbl>
1 Bug 53.9 64.8
2 Dark 74.6 69.5
3 Dragon 96.8 88.8
4 Electric 90.0 73.7
5 Fairy 78.5 84.7
6 Fighting 53.1 64.7
7 Fire 89.0 72.2
8 Flying 94.2 72.5
9 Ghost 79.3 76.5
10 Grass 77.5 70.4
11 Ground 56.5 62.8
12 Ice 77.5 76.3
13 Normal 55.8 63.7
14 Poison 60.4 64.4
15 Psychic 98.4 86.3
16 Rock 63.3 75.5
17 Steel 67.5 80.6
18 Water 74.8 70.5Ejercicio 3: Crea una nueva tabla en donde le restes el valor de ataque de cada individuo al valor total del grupo Type1
pokemon_grp <- pokemon %>%
group_by(Type1) %>%
mutate(Promedio_grupo = mean(Attack, na.rm = TRUE),
Attack_dif = Promedio_grupo - Attack) Ejercicio 4 Activida de LDH: Usando los valores de actividad enzimática que se encuentran en el directorio
data/:
- abre cada tibble y unelos en una sola con la función
rbind() - selecciona las columnas
ExpNum,TreatTemp,Meas_TempyActivityLDH - convierte la columna
ExpNumyMeas_Tempen factores - agrupa la tabla de acuerdo a esta columna
- calcula el promedio, desviación estandar y N para los valores de activtidad de LDH
- guarda esta nueva tabla en el directorio de resultados
tbl <-
list.files(path = "data", pattern = "Data_enzimas*", full.names = TRUE) %>%
map_df(~read.table(., sep = "\t", h = TRUE))
tbl %>%
tibble(.) %>%
select(ExpNum, TreatTemp, Meas_Temp, ActivityLDH) %>%
mutate(ExpNum = factor(ExpNum)) %>%
group_by(ExpNum, Meas_Temp) %>%
summarise(PromedioLDH = mean(ActivityLDH, na.rm = TRUE),
DesvLDH = sd(ActivityLDH, na.rm = TRUE),
N = n())5.10 count()
La función count() nos permite saber cuantas observaciones hay en una variable especifica. Al agregar el argumento sort = TRUEdevuelve una tabla descendiente con el número de observaciones.
Por ejemplo, para saber cuantos miembros de cada tipo hay en nuestra lista de pokemon:
pokemon %>%
count(Type1, sort = TRUE, name = "Numero")# A tibble: 18 x 2
Type1 Numero
<chr> <int>
1 Water 112
2 Normal 98
3 Grass 70
4 Bug 69
5 Psychic 57
6 Fire 52
7 Electric 44
8 Rock 44
9 Dragon 32
10 Ghost 32
11 Ground 32
12 Dark 31
13 Poison 28
14 Fighting 27
15 Steel 27
16 Ice 24
17 Fairy 17
18 Flying 45.11 ifelse()
La función ifelse() es utilizada en muchos contextos en los lenguajes de programación en donde se tiene la sintaxis:
ifelse(condicion, resultado positivo, regulado negativo)
por ejemplo
x = 20
ifelse(x == 20, "Si es", "no es")[1] "Si es"Entonces es posible utilizar la función ifelse dentro de mutate para transformar ciertas variables.
pokemon %>%
mutate(Tipo = ifelse(Type1 == "Grass", "Planta", Type1)) %>%
mutate(Rapidez = ifelse(Speed >= 100, "Muy rapido", "lento"))# A tibble: 800 x 15
ID Name Type1 Type2 Total HP Attack Defense Sp_Atk Sp_Def Speed
<dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 Bulbasaur Grass Pois~ 318 45 49 49 65 65 45
2 2 Ivysaur Grass Pois~ 405 60 62 63 80 80 60
3 3 Venusaur Grass Pois~ 525 80 82 83 100 100 80
4 3 VenusaurMeg~ Grass Pois~ 625 80 100 123 122 120 80
5 4 Charmander Fire <NA> 309 39 52 43 60 50 65
6 5 Charmeleon Fire <NA> 405 58 64 58 80 65 80
7 6 Charizard Fire Flyi~ 534 78 84 78 109 85 100
8 6 CharizardMe~ Fire Drag~ 634 78 130 111 130 85 100
9 6 CharizardMe~ Fire Flyi~ 634 78 104 78 159 115 100
10 7 Squirtle Water <NA> 314 44 48 65 50 64 43
# ... with 790 more rows, and 4 more variables: Generation <dbl>,
# Legendary <lgl>, Tipo <chr>, Rapidez <chr>5.12 Unir dos o mas tablas
5.12.1 Unir dos tablas con el mismo número de dimensiones
Para este ejercicio, vamos a generar dos tibbles sencillos:
x <- tibble(x1 = c("A", "B", "C"),
x2 = c(1, 2, 3))
x# A tibble: 3 x 2
x1 x2
<chr> <dbl>
1 A 1
2 B 2
3 C 3y <- tibble(x1 = c("B", "C", "D"),
x2 = c(2,3,4))
y# A tibble: 3 x 2
x1 x2
<chr> <dbl>
1 B 2
2 C 3
3 D 4Unir las columnas con bind_cols()
bind_cols(x, y)New names:
* x1 -> x1...1
* x2 -> x2...2
* x1 -> x1...3
* x2 -> x2...4# A tibble: 3 x 4
x1...1 x2...2 x1...3 x2...4
<chr> <dbl> <chr> <dbl>
1 A 1 B 2
2 B 2 C 3
3 C 3 D 4Unir las filas con bind_rows()
bind_rows(x, y)# A tibble: 6 x 2
x1 x2
<chr> <dbl>
1 A 1
2 B 2
3 C 3
4 B 2
5 C 3
6 D 4Unir los elementos en común con union()
union(x, y)# A tibble: 4 x 2
x1 x2
<chr> <dbl>
1 A 1
2 B 2
3 C 3
4 D 4intersect()toma las filas que aparecen en ambas tablas
intersect(x,y)# A tibble: 2 x 2
x1 x2
<chr> <dbl>
1 B 2
2 C 35.12.2 Unir dos tablas con diferentes dimensiones
La sintaxis para estas funciones es la misma xxxx_join(x1 = tabla1, x2 = tabla2, by = "columna en común")
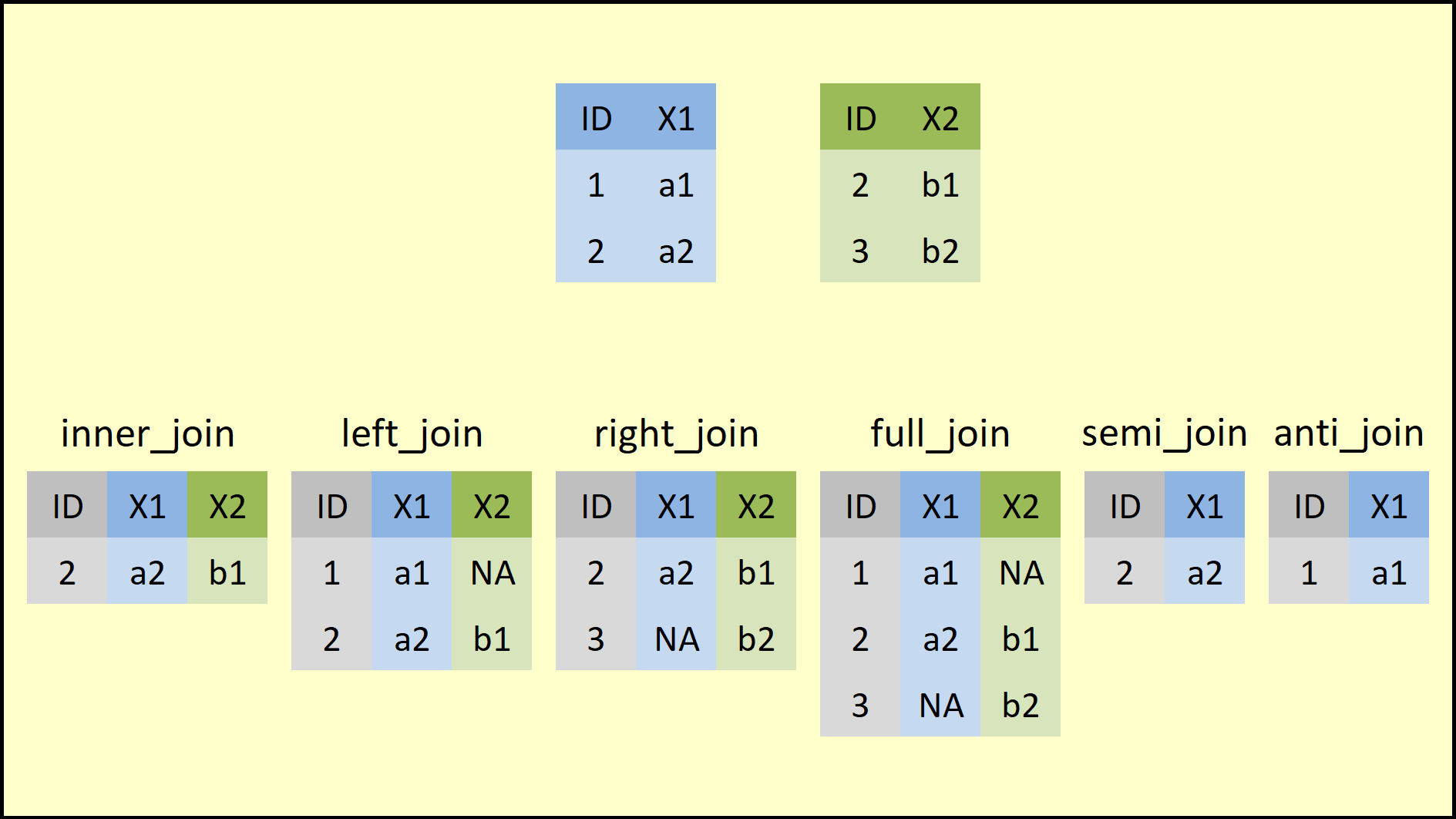
Para esto, vamos a construir otro par de tibbles
canciones <- tibble(cancion = c("I´am the warlus",
"Come together",
"Yesterday",
"Why"),
autor = c("John",
"John",
"Paul",
"Yoko"))
canciones# A tibble: 4 x 2
cancion autor
<chr> <chr>
1 I´am the warlus John
2 Come together John
3 Yesterday Paul
4 Why Yoko artista <- tibble(autor = c("George",
"John",
"Paul",
"Ringo"),
instrumento = c("sitara",
"guitarra",
"bajo",
"bateria"))
artista# A tibble: 4 x 2
autor instrumento
<chr> <chr>
1 George sitara
2 John guitarra
3 Paul bajo
4 Ringo bateria inner_join()
inner_join(canciones, artista, by = "autor")# A tibble: 3 x 3
cancion autor instrumento
<chr> <chr> <chr>
1 I´am the warlus John guitarra
2 Come together John guitarra
3 Yesterday Paul bajo left_join()
left_join(canciones, artista, by = "autor")# A tibble: 4 x 3
cancion autor instrumento
<chr> <chr> <chr>
1 I´am the warlus John guitarra
2 Come together John guitarra
3 Yesterday Paul bajo
4 Why Yoko <NA> right_join()
right_join(canciones, artista, by = "autor")# A tibble: 5 x 3
cancion autor instrumento
<chr> <chr> <chr>
1 I´am the warlus John guitarra
2 Come together John guitarra
3 Yesterday Paul bajo
4 <NA> George sitara
5 <NA> Ringo bateria full_join()
full_join(canciones, artista, by = "autor")# A tibble: 6 x 3
cancion autor instrumento
<chr> <chr> <chr>
1 I´am the warlus John guitarra
2 Come together John guitarra
3 Yesterday Paul bajo
4 Why Yoko <NA>
5 <NA> George sitara
6 <NA> Ringo bateria semi_join()
semi_join(canciones, artista, by = "autor")# A tibble: 3 x 2
cancion autor
<chr> <chr>
1 I´am the warlus John
2 Come together John
3 Yesterday Paul anti_join()
anti_join(canciones, artista, by = "autor")# A tibble: 1 x 2
cancion autor
<chr> <chr>
1 Why Yoko Ejercicio: Utiliza la función
read_csv()para abrir el archivo pokemon_extended.csv. Posteriormente, genera una nueva tabla donde en una sola cadena unas la información del peso y altura de cada pokemon con el el resto de la tabla original, conservando solamente aquellos individuos presentes en ambas tablas, selecciona solamente las columnas Name, Type1, Total, HP, height_m, weight_gk y filtrar aquellos pokemon que tengan un peso superior al promedio global
pokemon_ext <- read_csv("data/pokemon_extended.csv")
pokemon_E5 <- pokemon %>%
# Unir ambas tablas con inner join
inner_join(pokemon_ext, by = "Name") %>%
#seleccionar las columnas de interes
select(Name, Type1, Total, HP, height_m, weight_kg) %>%
#filtrar los individuos con peso mayor al promedio
filter(weight_kg > mean(weight_kg, na.rm = TRUE))Ejercicio: Utilizando
read_csv()genera un par de objetos nuevos con las tablas Trancriptome_DGE.csv y Transcriptome_Anotacion.csv. En una sola cadena, une ambas tablas de manera que cada transcrito de la tabla DGE tenga su anotación; filtra aquellos transcritos que tuvieron un valor de logFC mayor a 0; y genera una tabla con el numero de organismos de cada genero (genus).
expresion <- read_csv("data/Transcriptome_DGE.csv")
expresion
anotacion <- read_csv("data/Transcriptome_Anotacion.csv")
expresion %>%
inner_join(anotacion, by = "transcript") %>%
filter(logFC > 0) %>%
count(genus, sort = TRUE)5.13 Cambio de formato long y wide con pivot_longer() y pivot_wider()
Para entender la diferencia entre el formato ancho y largo, usemos la siguiente tabla con los datos de consumo de oxígeno de 10 individuos a lo largo de seis días:
MO2 <- read_csv("data/Consumo_oxigeno_wide.csv")
-- Column specification --------------------------------------------------------
cols(
individuo = col_character(),
dia1 = col_double(),
dia2 = col_double(),
dia3 = col_double(),
dia4 = col_double(),
dia5 = col_double(),
dia6 = col_double()
)MO2# A tibble: 10 x 7
individuo dia1 dia2 dia3 dia4 dia5 dia6
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 E1 11.7 46.6 8.10 18.7 59.6 47.5
2 E2 12.7 8.95 15.2 31.8 30.3 30.4
3 E3 14.5 27.2 11.9 29.3 42.2 40.5
4 E4 27.6 29.5 17.1 30.1 61.2 63.0
5 E5 7.70 29.7 19.0 31.8 71.0 42.7
6 E6 6.64 34.8 14.3 5.18 61.6 65.1
7 E7 10.4 36.3 9.66 24.1 60.6 65.4
8 E8 14.5 40.1 8.11 33.6 18.5 59.4
9 E9 5.62 48.8 14.5 10.4 21.0 52.4
10 E10 6.97 14.6 NA NA NA 45.2En este caso, los individuos estan en filas y cada dia en una columna diferentes. Para transformar en formato largo, usando la funcion pivot_longer()
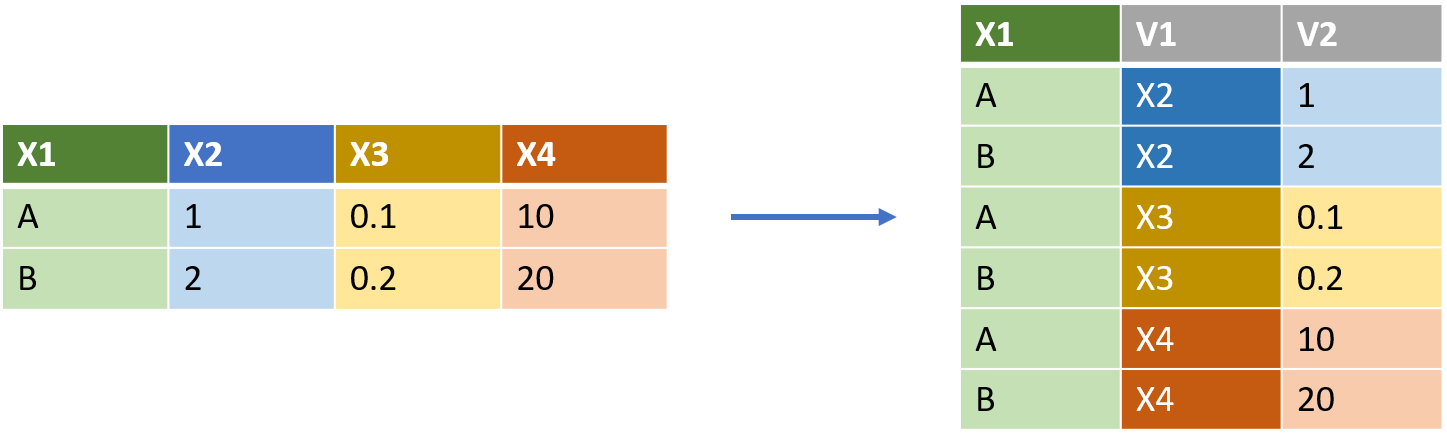
imagen tomada de: thinkr.fr/wp-content/uploads/wide_long.png
La sintaxis para utilizar estas funciones es la siguiente:
pivot_longer( data, cols, names_to = “NNN”, values_to = “NNN”)
en donde:
- data: Corresponde a la tabla (no es necesario especificar si viene de una cadena)
- cols: cuales son las columnas que se desea transformar. Si se desea mantener solo una variable fija, se puede espeficicar con
-variable - names_to: Nombre de la columna que contiene las columnas proporcionadas
- values_to: Nombre de la variable que contiene los valores asociados a las columnas proporcionadas
MO2_long = MO2 %>%
pivot_longer(-individuo, names_to = "Dia", values_to = "MO2")
MO2_long# A tibble: 60 x 3
individuo Dia MO2
<chr> <chr> <dbl>
1 E1 dia1 11.7
2 E1 dia2 46.6
3 E1 dia3 8.10
4 E1 dia4 18.7
5 E1 dia5 59.6
6 E1 dia6 47.5
7 E2 dia1 12.7
8 E2 dia2 8.95
9 E2 dia3 15.2
10 E2 dia4 31.8
# ... with 50 more rowsPor el contrario, podemos usar pivot_wider()para realizar la operación opuesta
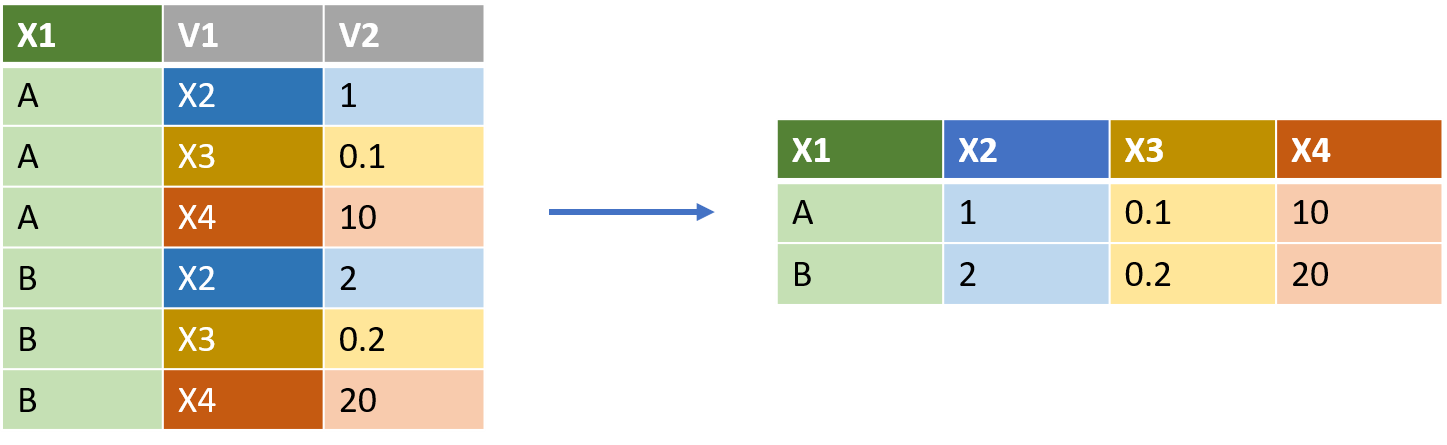
imagen tomada de:thinkr.fr/wp-content/uploads/long_wide.png
MO2_wide = MO2_long %>%
pivot_wider(individuo, names_from = Dia, values_from = MO2)
MO2_wide# A tibble: 10 x 7
individuo dia1 dia2 dia3 dia4 dia5 dia6
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 E1 11.7 46.6 8.10 18.7 59.6 47.5
2 E2 12.7 8.95 15.2 31.8 30.3 30.4
3 E3 14.5 27.2 11.9 29.3 42.2 40.5
4 E4 27.6 29.5 17.1 30.1 61.2 63.0
5 E5 7.70 29.7 19.0 31.8 71.0 42.7
6 E6 6.64 34.8 14.3 5.18 61.6 65.1
7 E7 10.4 36.3 9.66 24.1 60.6 65.4
8 E8 14.5 40.1 8.11 33.6 18.5 59.4
9 E9 5.62 48.8 14.5 10.4 21.0 52.4
10 E10 6.97 14.6 NA NA NA 45.2
imagen tomada de:thinkr.fr/wp-content/uploads/long_wide.png
Ejercicio: De la tabla pokemon, utiliza filter() para filtrar a las evolcuiones de Eevee

pokemon %>%
filter(str_detect(Name, "eon"),
!str_detect(Name, "Charm"))
sessionInfo()R version 4.0.5 (2021-03-31)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows 10 x64 (build 19043)
Matrix products: default
locale:
[1] LC_COLLATE=English_United States.1252
[2] LC_CTYPE=English_United States.1252
[3] LC_MONETARY=English_United States.1252
[4] LC_NUMERIC=C
[5] LC_TIME=English_United States.1252
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] readxl_1.3.1 forcats_0.5.1 stringr_1.4.0 dplyr_1.0.5
[5] purrr_0.3.4 readr_1.4.0 tidyr_1.1.3 tibble_3.0.4
[9] ggplot2_3.3.5 tidyverse_1.3.1 workflowr_1.6.2
loaded via a namespace (and not attached):
[1] tidyselect_1.1.1 xfun_0.23 bslib_0.2.5.1 haven_2.3.1
[5] colorspace_2.0-0 vctrs_0.3.8 generics_0.1.0 htmltools_0.5.1.1
[9] yaml_2.2.1 utf8_1.2.1 rlang_0.4.11 jquerylib_0.1.4
[13] later_1.1.0.1 pillar_1.6.0 withr_2.4.2 glue_1.4.2
[17] DBI_1.1.0 dbplyr_2.1.1 modelr_0.1.8 lifecycle_1.0.0
[21] cellranger_1.1.0 munsell_0.5.0 gtable_0.3.0 rvest_1.0.0
[25] evaluate_0.14 knitr_1.30 ps_1.5.0 httpuv_1.5.4
[29] fansi_0.4.2 highr_0.8 broom_0.7.6 Rcpp_1.0.5
[33] promises_1.1.1 backports_1.2.1 scales_1.1.1 jsonlite_1.7.2
[37] fs_1.5.0 hms_1.0.0 digest_0.6.27 stringi_1.5.3
[41] rprojroot_2.0.2 grid_4.0.5 cli_2.5.0 tools_4.0.5
[45] magrittr_2.0.1 sass_0.4.0 crayon_1.4.1 whisker_0.4
[49] pkgconfig_2.0.3 ellipsis_0.3.1 xml2_1.3.2 reprex_2.0.0
[53] lubridate_1.7.10 rstudioapi_0.13 assertthat_0.2.1 rmarkdown_2.9
[57] httr_1.4.2 R6_2.5.0 git2r_0.27.1 compiler_4.0.5